今天给同学们分享一篇生信文章“Identification of potential biomarkers for lung adenocarcinoma: a study based on bioinformatics analysis combined with validation experiments”,这篇文章于2024年9月19日发表在Front Oncol期刊上,影响因子为3.5。

结果:
共有 401 个关键模块差异基因与 LUAD 相关
在 TCGA-LUAD 中,对所有样本进行聚类分析,结果显示没有明显的异常值,R2 设置为 0.85,软阈值 β 确定为 4(图 2A)以构建无标度网络。通过 WGCNA 分析,共鉴定出 12 个共表达模块(图 2B)。在这些模块中,MEblue 模块与 LUAD 表现出最强的负相关 (cor=-0.85) (图 2C)。
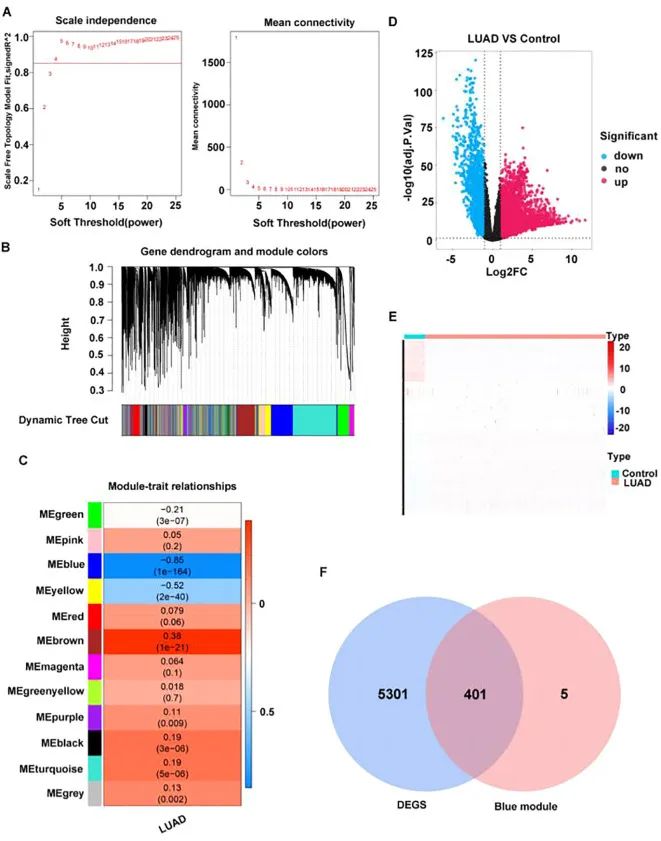
通过R包edgeR(3.36.0版本)分析TCGA数据集中LUAD和正常样本的差异基因,鉴定出5,702个DEGs,其中3,785个上调基因,1,917个下调基因(图2D,E)。然后,确定 5702 个 DEGs 与 406 个关键模块基因之间的交集,产生 401 个关键模块差异基因(
图 2F)。
通过机器学习获得候选生物标志物
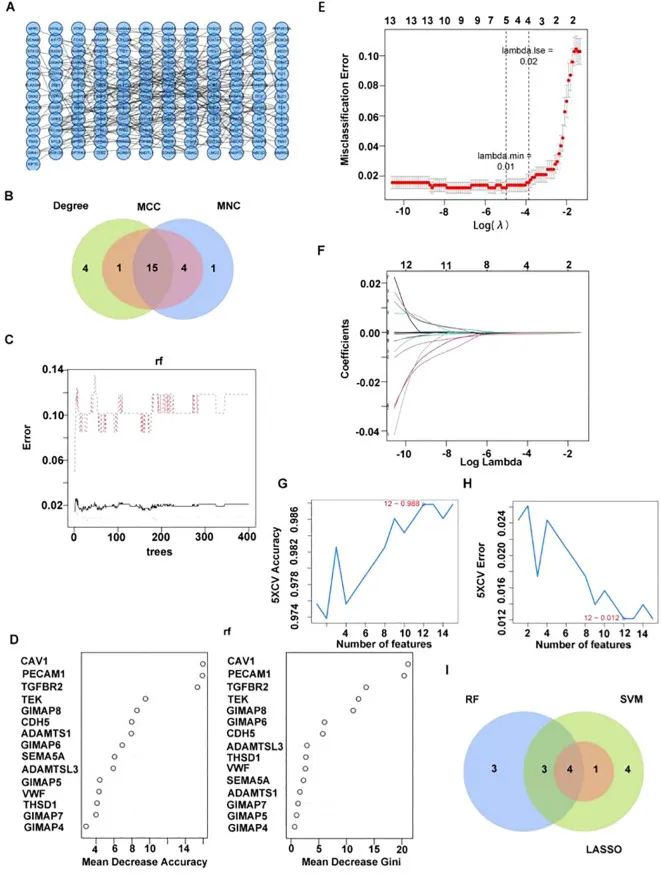
基于 401 个关键模块差异基因构建 PPI 网络 ( 图 3A )。此后,对网络拓扑参数 MCC、MNC 和 Degree 的交集分析产生了 15 个候选基因(图 3B)。然后对 15 个候选基因进行排名,根据基尼系数算法(平均减少基尼系数)选择前 10 个作为关键候选枢纽基因(图 3C、D)。通过应用机器学习 LASSO 回归算法对基因进行分析,以筛选 3 个关键的候选枢纽基因(图 12E、F)。使用 SVM-RFE 方法共获得 3 个候选枢纽基因(图 6G、H)。最后,将从三种机器学习算法获得的基因进行交集,以获得四个枢纽基因(GIMAP1、CAV1、PECAM2 和 TGFBR3)(图 <>I)。
候选生物标志物都具有极强的预测能力
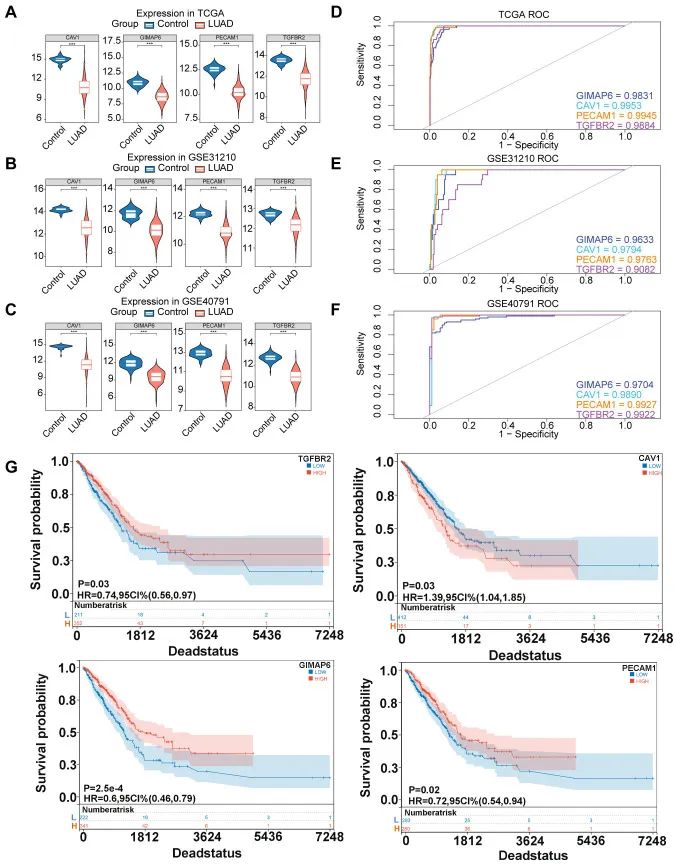
在 TCGA-LUAD、GSE40791 和 GSE31210 中,观察到与对照组相比,LUAD 患者四种候选生物标志物的表达水平显着降低(图 4A-C
)。有趣的是,ROC 分析显示,这些生物标志物在所有数据集中的 AUC 值为 >0.9 (TCGA-LUAD:GIMAP6,AUC=0.9831;CAV1,AUC=0.9953;PECAM1, AUC=0.9945;TGFBR2, AUC=0.9884;GSE31210:GIMAP6, AUC=0.9633;CAV1,AUC=0.9794;PECAM1, AUC=0.9763;TGFBR2,AUC=0.9082;GSE40791:GIMAP6,AUC= 0.9704;CAV1,AUC= 0.9890;PECAM1,AUC= 0.9927;TGFBR2,AUC= 0.9922),表明它们对 LUAD 具有很强的预测能力(图 4D-F)。此外,KM 生存分析表明这四种生物标志物的表达与患者预后之间存在显着关联(图 4G)。
在正向 MR 分析中,TGFBR2 和 LUAD 之间存在因果关系
在评估基因表达与疾病状态之间是否存在因果关系时,作者使用表达数量性状位点 (eQTL) 作为工具变量来推断基因表达变化与疾病状态之间的因果关系。TGFBR2 、 GIMAP6 和 CAV1 被视为暴露因素,而 LUAD 被视为结局。TGFBR2 相关数据集 (eqtl-a-ENSG00000163513) 包括 31,470 个样本和 19,824 个 SNP。GIMAP6 相关数据集 (eqtl-a-ENSG00000133561) 包含 31,684 个样本和 20,006 个 SNP,CAV1 相关数据集 (eqtl-a-ENSG00000105974) 包含 31,470 个样本和 16,582 个 SNP。基于 IVW 结果,只有 eqtl-a-ENSG00000163513 (p = 0.04, b=−0.06) 和 ieu-a-984 之间的关系满足 p < 0.05;而 EQTL-A-ENSG00000133561 (P=0.35) 和 IEU-A-984 之间的效应满足 P > 0.05,表明 TGFBR2 与 LUAD 有显著的因果关系,是 LUAD 的安全因素。
在 GWAS 中,作者筛选了与 LUAD 显著相关的 SNP(r 2 =0.001;kb=10,000) 用于 IVW 分析。未发现 GIMAP6 和 LUAD 的因果效应。由于少于 1 个 SNP,无法使用 IVW 方法评估 CAV5 和 LUAD 之间的关联。随后,散点图显示 IVW 的斜率为负,截距可以忽略不计(图 0A)。进一步的森林图显示 IVW 的总体效果为 <5(图 2B)。因此,TGFBR0 是 LUAD 的安全因子。随后,通过敏感性分析验证了 MR 分析结果的可靠性。首先,异质性检验的Q_pval为 949.0,表明没有异质性。接下来,水平多向性检验的 p 值为 673.2,表明没有水平多向性。最后,在通过留一法试验去除单个 SNP 后,残留的 SNP 对整体影响不大,证明 MR 结果是可靠的(图 <>C)。
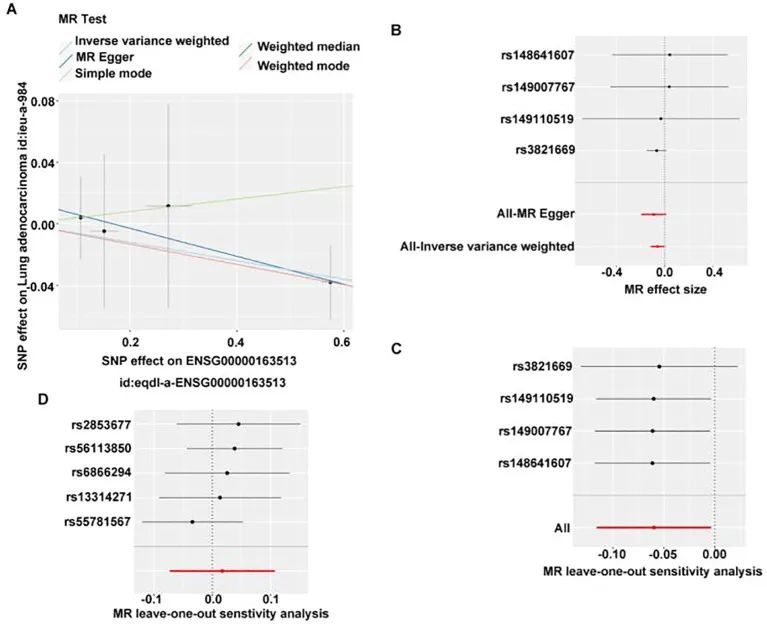
随后,作者使用肺腺癌作为暴露因子,TGFBR2 作为结果。使用 5×10−8 筛选工具变量,r2 = 0.001;kb=10,000 个条件产生了与肺腺癌相关的 2 个工具变量,但与 TGFBR3 无关,IVW 分析显示 LUAD 对 TGFBR0 表达水平没有显着的因果影响 (p > 05.4)。同时,水平多效性的 p 值为 494.5,表明没有水平多效性,留一法检验也表明去除 SNP 后残留的 SNPs 对整体效应影响不大(
图 2D),综合表明反向 MR 分析结果是可靠的。最后,反向 MR 分析显示 LUAD 不会导致 TGFBR<> 表达发生变化。
TGFBR2 与 LUAD 临床特征的关系
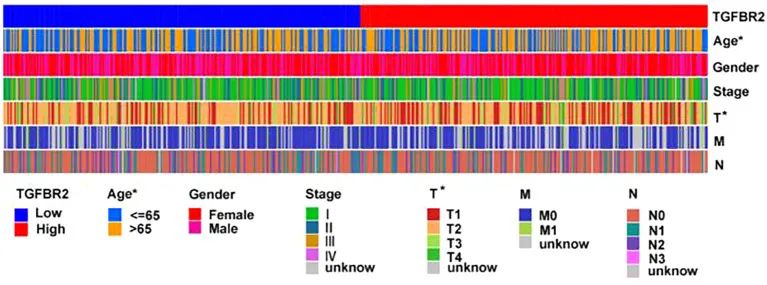
为了进一步探索临床特征与 TGFBR2 表达之间的关系,作者使用了来自 TCGA-LUAD 数据库的 LUAD 样本数据来验证作者的发现。结果显示 LUAD 患者 TGFBR2 表达与 t 期 (T1、T2) 和年龄相关 ( 图 6 )。
LUAD 中的 TGFBR2 和免疫细胞浸润
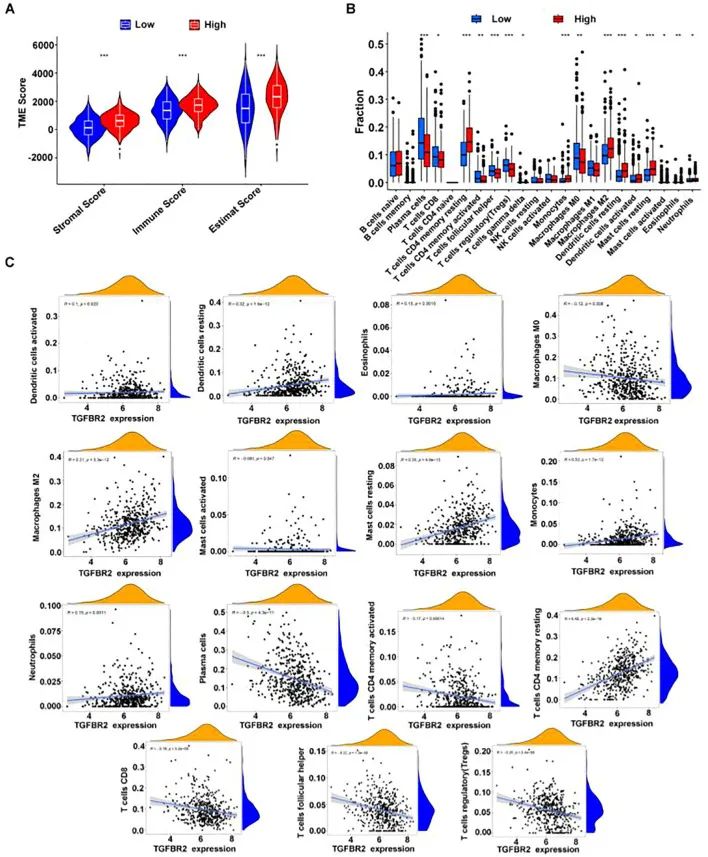
利用 Estimate 程序评估免疫微环境,结果表明,与低表达组相比,高 TGFBR2 表达组的免疫评分显着更高(图 7A)。使用 CIBERSORT 算法的分析揭示了高 TGFBR15 表达组和低 TGFBR2 表达组之间 7 种免疫细胞类型比例的差异(图 2B)。进一步的相关性分析表明,TGFBR2 表达与树突状细胞、嗜酸性粒细胞、巨噬细胞 M4、肥大细胞、单核细胞、中性粒细胞和静息 CD0 记忆 T 细胞的存在呈正相关,与巨噬细胞 M4、浆细胞、T 细胞 CD8 记忆激活、T 细胞 CD7、T 细胞滤泡辅助细胞和 T 细胞调节 (Tregs) 呈负相关(图0C ).TGFBR047 和肥大细胞之间的 p 值 = 2.2 表明,尽管这种相关性在统计学上是显着的,但它略微接近常规阈值。这些观察结果表明 TGFBR<> 表达与 LUAD 中的免疫浸润之间存在合理的联系。
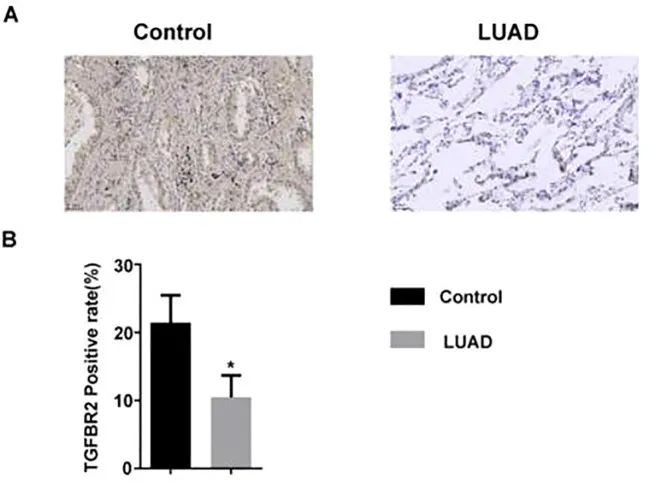
TGFBR2 在 LUAD 组织中的表达增加
免疫组化检测 LUAD 患者 TGFBR2 的表达水平。结果表明,与正常肺组织相比,TGFBR2 在 LUAD 组织中的表达降低(
图 8)。
GFBR2 siRNA 促进肺癌细胞 A549 的增殖、迁移和侵袭,并抑制其凋亡
为了进一步验证 TGFBR2 的功能,作者在本研究中设计了两个靶向 TGFBR2 的 siRNA,旨在诱导肺癌细胞中 TGFBR2 基因的静止或休眠状态。通过 qRT-PCR 和 Western blotting 评估转染效率。结果显示,与对照组和 NC-siRNA 组相比,siTGFBR2-2 和 siTGFBR1-2 组 TGFBR2 在 mRNA 和蛋白水平上的表达均降低;这证实了转染测定的成功,并且 siTGFBR2-2 具有更好的干扰效果,使其适合用于后续实验(图 9A、B)。
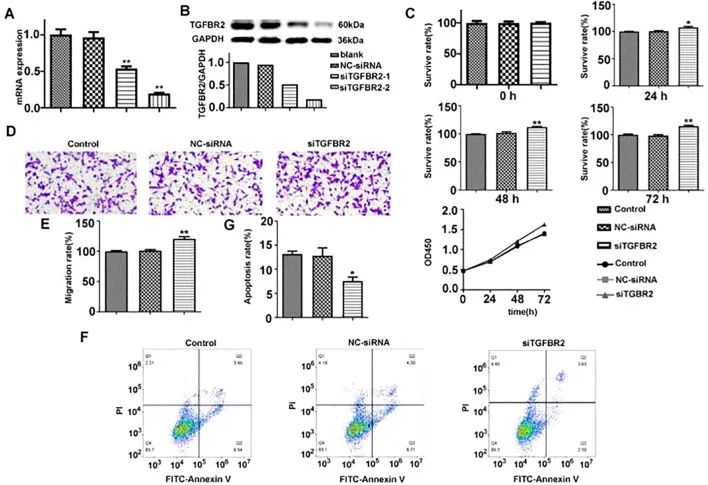
CCK8 法评价 TGFBR2 siRNA 对 A549 细胞增殖的影响。结果显示,siTGFBR549 转染后 A2 细胞的增殖呈时间依赖性增加。与对照组和 NC-siRNA 组相比,siTGFBR2 组在同一时间点的增殖率显着增加 (p<0.01) (图 9C),表明 TGFBR2 基因在抑制 A549 细胞增殖中发挥作用。
进行 Transwell 实验以评估 TGFBR2 siRNA 对 A549 细胞迁移的有效性。结果显示,与对照组和 NC-siRNA 组相比,siTGFBR2 组迁移细胞的数量显着增加 (p<0.01) (图 9D、E)。这一发现表明 TGFBR2 基因在抑制 A549 细胞迁移中发挥作用。
流式细胞术评价 TGFBR2 siRNA 对 A549 细胞凋亡的影响。结果表明,与对照组和 NC-siRNA 组相比,siTGFBR2 组细胞凋亡率显著降低 (p<0.01) ( 图 9F、G ),表明 TGFBR2 基因有助于促进 A549 细胞凋亡。
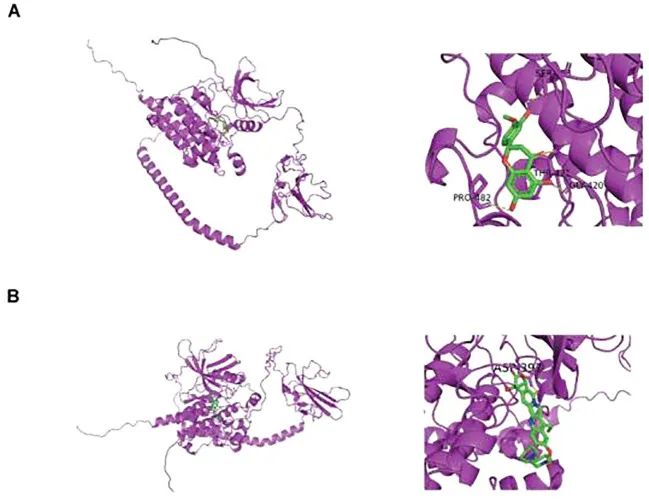
在 DGIDB 数据库中,确定了两种靶向 TGFBR2 的潜在药物,伊立替康和橙皮素。然后,通过分子对接分析药物与蛋白质之间的结合亲和力。发现伊立替康和 TGFBR2 之间的结合亲和力为 -8.7 kcal/mol,表明它们具有很强的结合活性,而橙皮素和 TGFBR2 之间的结合亲和力为 -6.2 kcal/mol,表明它们具有优雅的结合活性(图 10)。