酶在合成生物学和生物催化领域引起了相当大的关注,被广泛誉为光学活性化学品生物合成的首选。酶在活性中心内立体化学结构通常表现为独特的空间、疏水和静电特性,构成了高立体选择性的基础。然而,在对于具有几乎对称结构的底物时,酶在保持高立体选择性方面面临挑战。手性环己-3-烯-1-羧酸(CHCA)具有一个几乎对称的六原子环,是合成各种药物、农用化学品和天然产物的关键组成部分。这些底物通常被认为是“难以区分”的,被化学催化剂和生物催化剂都难以识别。定向进化在加速立体选择性酶的开发中起着关键作用,在催化“难以区分”的底物时,也面临一定的挑战。
机器学习(ML)作为一种强大的计算工具,它为基于大量高质量数据和统计模型促进定向进化提供了更直接的捷径。ML是数据驱动的,可以识别催化模式,预测有用的突变体,并擅长预测定向进化的新替代组合。ML预测的成功取决于训练数据集的质量,生成高质量的数据集和选择合适的描述符是ML预测成功的关键。
在这项研究中,首先,我们提出了一种高通量方法来生成羧酸酯酶AcEst1的高质量数据集。通过将水解反应与醇脱氢酶(ADH)催化的氧化反应偶联来确定对(R)-和(S)-CHCE的初始反应速率,(R)-和(S)-CHCE之间的初始反应速率之比,称为表观对映选择性(Eapp)。这种方法依赖于“真实底物”并准确反映了实际的反应动力学。我们对醇脱氢酶进行筛选并对ADH10进行了催化活性的改造,得到催化活性显著提高和亲和力提升的双突变体ADH10V84L/F197V,并对催化剂量、辅酶、pH和底物浓度等进行优化,获得最佳的高通量筛选条件。
随后,为了收集关于AcEst1突变体对映选择性的多样化和高质量数据集以构建ML预测因子,确定了位于催化S201周围20个非保守残基用于饱和突变。在删除失活突变体的数据后,在1920个突变体中获得760个高质量数据集,利用羧酯酶的生化特征包括体积、疏水性、亲水性、静电、氢键、π-π相互作用和到催化残基的距离用于训练ML模型。我们评估了包括核岭回归(KRR)、高斯过程回归(GPR)、梯度提升回归树(GBRT)、随机森林回归(RFR)、支持向量回归(SVR)和贝叶斯岭回归(BRR)6个回归模型在AcEst1的对映选择性与7个生化特征之间的相关性。根据回归结果,GBRT的表现优于GPR、KRR、
RFR、SVR和BRR。决定系数(R2)GBRT模型的值达到0.93,均方误差(MSE)为0.12。景观分析显示数据分布平滑,表明GBRT性能优异。KRR、SVR和BRR对Eapp的预测无效,表现出较低的R2值(低于0.55)。RFR显示出比GPR更好的性能,具有更高的R2和较低的MSE。然而,RFR预测Eapp升高突变体的能力不如GBRT和GPR稳定。在我们的模型中高质量的数据集和生化特征的结合获得更高的R2,尝试减少特征数量并重新训练GBRT模型导致相关性降低,证明了这七个特征的协同效应。
为了提高经过训练的GBRT预测模型的准确性,我们系统地组合了有利的单突变体来生成双突变体。Eapp增加或降低的所有单个突变体配对,获得各种双突变体。V257M/Y228M的Eapp值最高(13.8),而L297F/L249A的Eapp最低(0.36)。这些双突变体的加入进一步丰富了用于重新训练GBRT模型的数据集。然后使用单突变体和双突变体对GBRT预测因子进行重新训练。重新训练的GBRT预测因子表现出出色的性能(R2为0.97,MSE为0.11),应用于下一步指导AcEst1立体发散进化的组合突变(图1)。
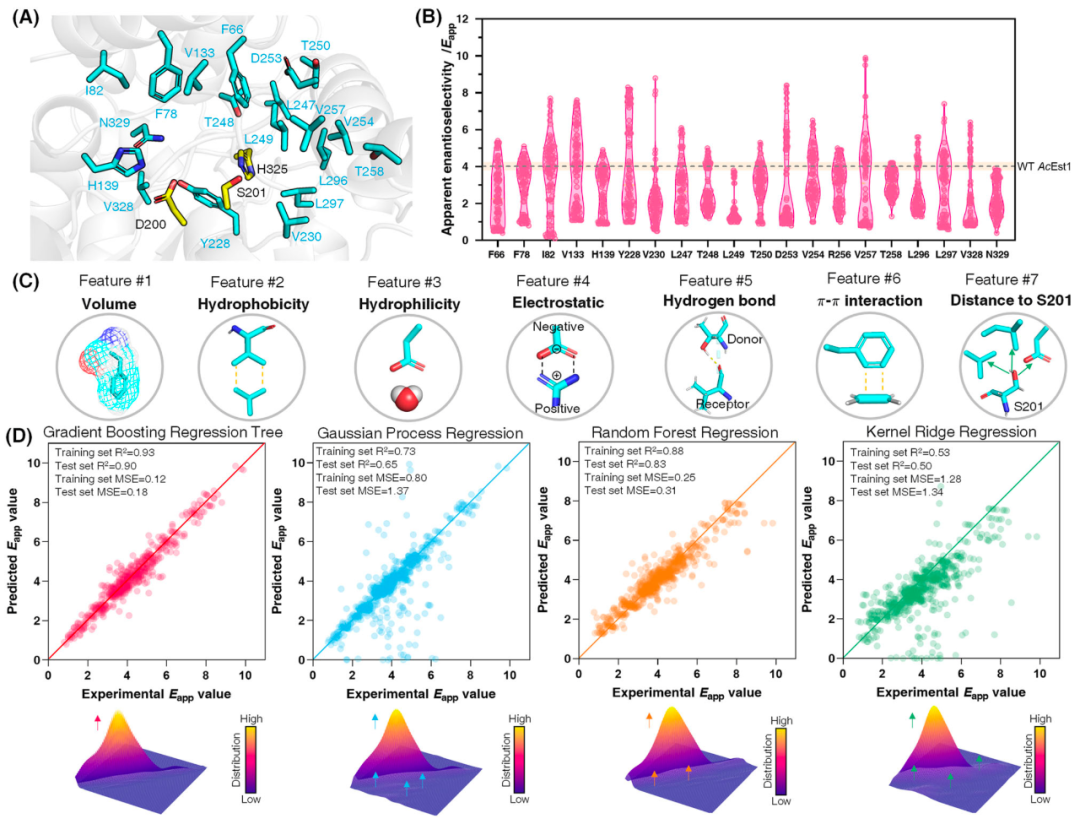
图1 高质量的位点特异性饱和突变结果和预测AcEst1的Eapp机器学习模型的开发将经过训练的ML预测模型应用于设计组合突变体。考虑到V257M和L297F是Eapp最高和最低的单个突变体,它们被选为(R)-选择性和(S)-选择性AcEst1突变体立体发散进化的起点。因此,V257M和L297F分别被指定为
DR1和DS1,并利用GBRT预测组合突变的Eapp。对于(R)-选择性进化,为了验证GBRT预测结果的准确性,我们实验构建了DR1、DR2和DR3,通过实验确定它们的对映选择性值(E值)。E值从WT的7.3逐渐增加到DR1的40.1、DR2的59.1和DR3的103。同样构建了(S)-选择性的突变体DS1-6,DS6的E值为−11,显著低于WT。虽然DS6的对映选择性不如DR3高,但考虑到(R)-和(S)-S1近乎对称的结构,对AcEst1的选择性已经算是相当显著的变化。因此,使用我们训练的GBRT预测因子,已经实现了AcEst1向近乎对称酯的立体发散进化,从而产生了两个立体互补突变体(图2)。这些突变体进一步用于合成手性CHCA的两种对映异构体。
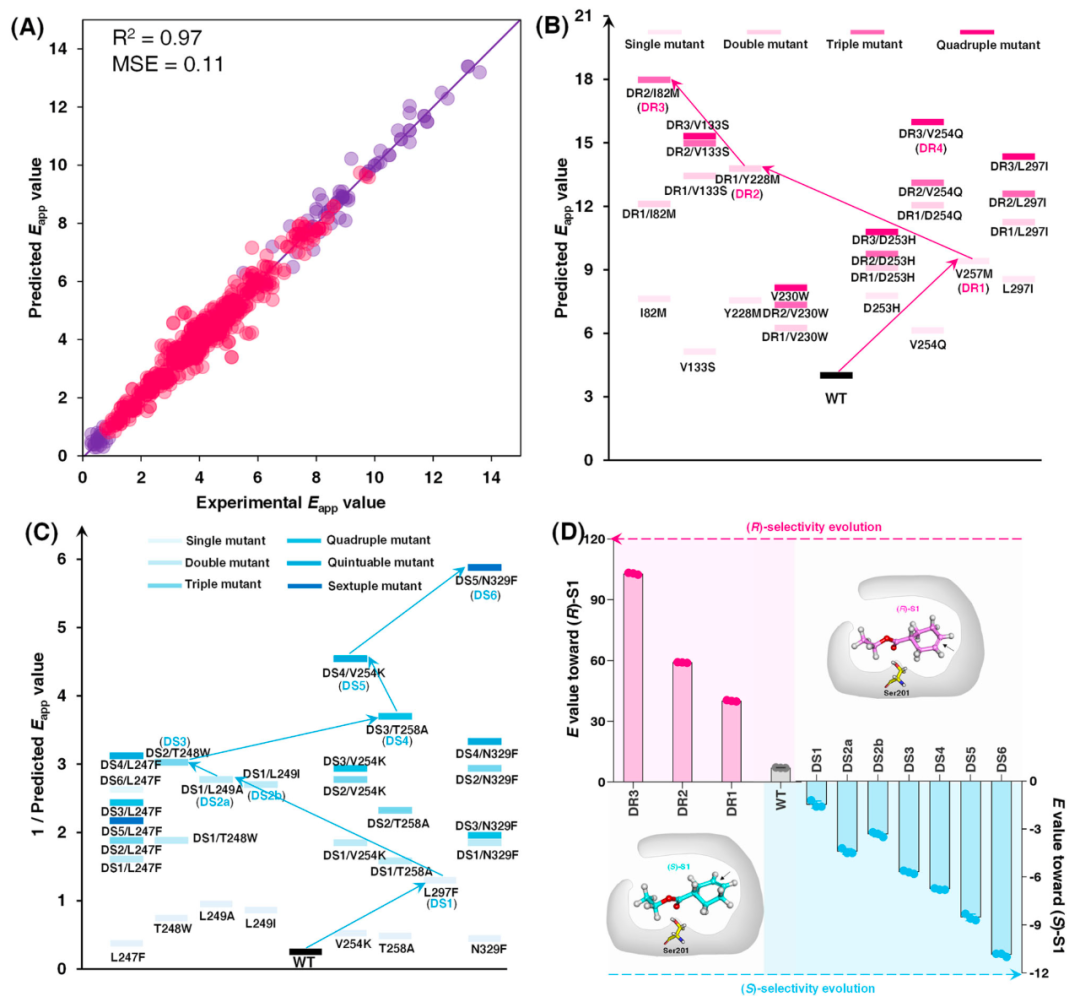
图2 ML 指导的AcEst1的(R)和(S)-选择性突变体的立体发散进化最后我们对互补突变体的催化潜力进行评估,并在1 M底物浓度下实现(R)和(S)-CHCE的生产与分离。采用分子动力学(MD)模拟及QM/MM计算解释了羧酯酶及其突变体立体选择性控制的分子机制(图3)。
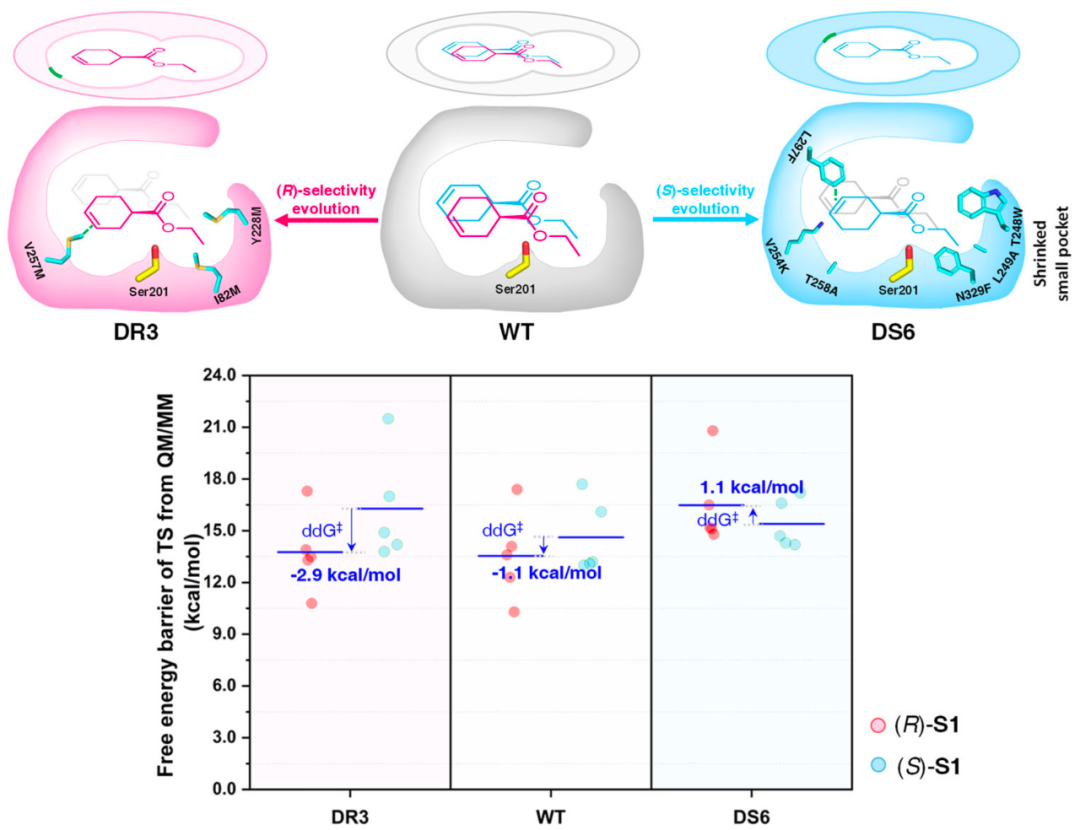
图3
使用QM/MM计算进行WT、DR3和DS6的互补对映选择性和自由能分析江南大学博士研究生窦哲为论文第一作者,倪晔教授和许国超副教授为论文共同通讯作者。上述研究得到了国家重点研发计划(2019YFA0906401)、国家自然科学基金(22078127, 22378169)等项目的资助。