
柏林工业大学(Technische
Universität Berlin)Juri
Rappsilber以及Oliver
Brock等研究人员合作开发新蛋白结构预测工具-AlphaLink,将交联质谱 (crosslinking
mass spectrometry,这里用的noncanonical
amino acid photo-leucine来交联) 获取的蛋白结构限制信息整合到AlphaFold2蛋白预测框架。从而提高了:1. 少同源序列的蛋白(比如病毒、抗体可变区以及从头设计的蛋白等常出现这种情况)的预测效果;2. 定位在实验条件下的蛋白构象(1–3)。
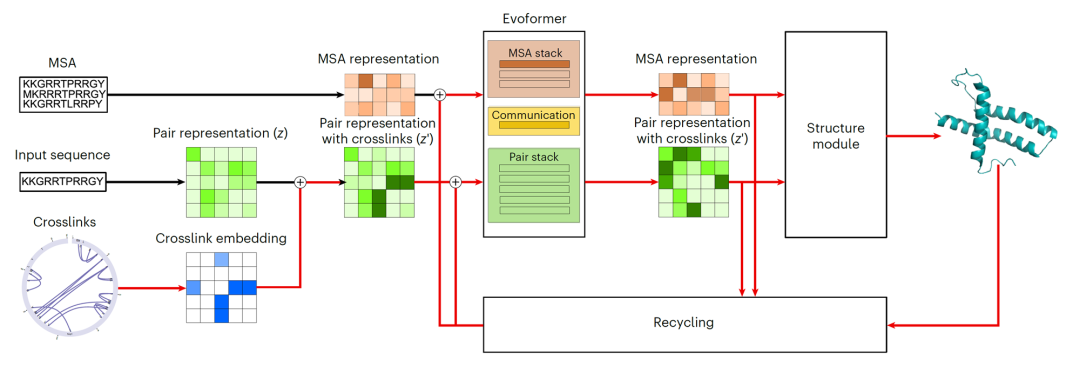 AlphaLink框架(3)
AlphaLink框架(3)
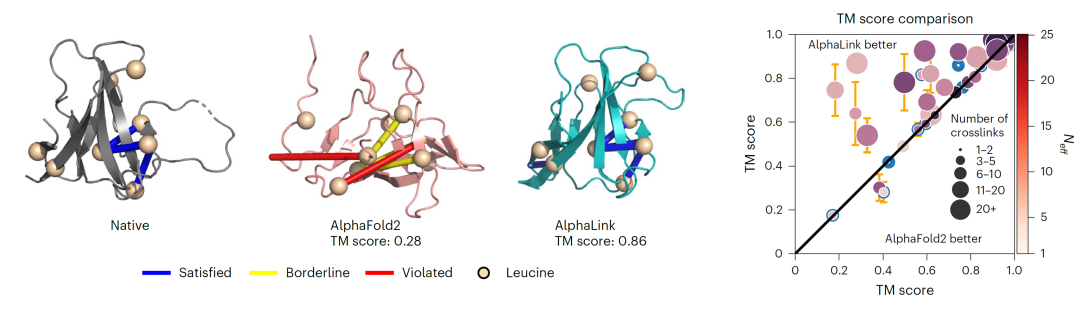 AlphaLink提高少同源序列蛋白(Neff偏低)结构预测质量(注:模拟数据)(3)
AlphaLink提高少同源序列蛋白(Neff偏低)结构预测质量(注:模拟数据)(3)
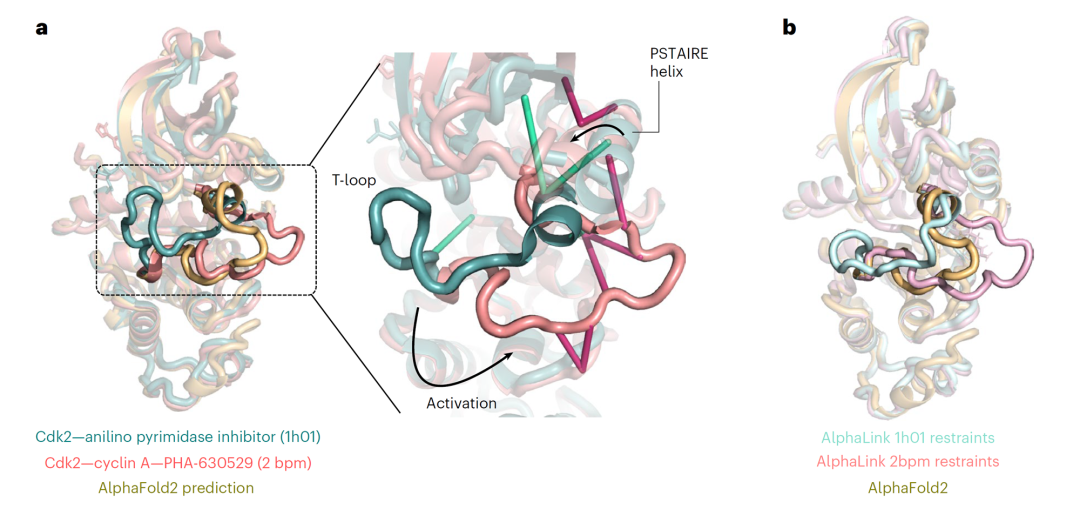 AlphaLink预测Cdk2蛋白在活跃与抑制状态的构象(注:模拟数据)(3)
AlphaLink预测Cdk2蛋白在活跃与抑制状态的构象(注:模拟数据)(3)
该项工作2023年3月20日发表在nature biotechnology;研究人员表示该研究提供了通用的使用实验数据辅助基于深度学习的算法预测原位蛋白结构/构象的框架(3)。
Comment(s):
结合多种蛋白酶解深度质谱可进一步提高此工具效果,参考:Nature Biotechnology | 深度人蛋白质组测序系统检测蛋白突变与可变剪切。
另外,该交联质谱数据整合到基于大语言模型的蛋白预测工具也是不错的选择,在基本不影响速度的条件下有望提高预测准确性,参考: Science | 大语言模型极速预测蛋白结构。
参考文献:
1. J. Jumper et al., Highly accurate
protein structure prediction with AlphaFold. Nature. 596, 583–589
(2021).
2. M. Suchanek, A. Radzikowska, C. Thiele,
Photo-leucine and photo-methionine allow identification of protein-protein
interactions in living cells. Nat. Methods. 2, 261–267 (2005).
3. K. Stahl, A. Graziadei, T. Dau, O.
Brock, J. Rappsilber, Protein structure prediction with in-cell
photo-crosslinking mass spectrometry and deep learning. Nat. Biotechnol.(2023), doi:10.1038/s41587-023-01704-z.
原文链接:
https://www.nature.com/articles/s41587-023-01704-z