
看着落日倾泻而下的光晖,高楼大厦慢慢亮起点点灯光,不禁感叹现在社会的发展真的很迅速,忽然联想到现正进入快速发展期、被广泛应用到研究的各方面的机器学习,大家是不是也会困扰于如何创新地使用机器学习呢?最近火火搜索到一篇基于机器学习开发的新框架并应用于ecDNA的识别研究的高分文章,快来和火火一起看看吧~
文章于2024年2月发表在Nature Communications杂志上,影响因子达到14.7。作者开发了一个基于机器学习的计算框架GCAP,能够从全外显子测序(WES)数据中识别和表征ecDNA扩增,GCAP也得到了多组学数据的广泛验证。这篇文章通过开发新的框架,有助于更好地识别ecDNA并揭示其在癌症中的临床意义,这种新思路可以为大家如何应用生信开辟自己的科研提供很好的参考。(ps:随着生信的广泛应用,大家是不是会因为想不到如何创新地应用生信而感到困扰呢?快来联系火火,火火这有大量好文,可以为大家提供超多素材!)
定制生信分析
云服务器租赁
加好友备注“99”领取试用

题目:基于机器学习的大规模队列中染色体外DNA的识别揭示了其在癌症中的临床意义
杂志:Nature Communications
影响因子:14.7
发表时间:2024年2月
公众号回复“666”,即可领取原文献,文献编号20240722
ecDNA随着技术的进步逐渐凸显出其作为一种新兴癌症标志的关键作用,具备作为癌症诊断的分子标记和癌症治疗的药物靶点的潜力。但目前仍缺乏临床上可行的方法来识别ecDNA扩增,其在常见异质性恶性肿瘤或晚期治疗中的临床意义还不清晰,而应用于研究ecDNA的技术和成本也还有待改进。因此作者开发了一个计算框架 GCAP,能利用全外显子测序数据集对临床癌症样本中的 ecDNA 扩增进行鉴定和表征。
研究开发基于机器学习的计算框架GCAP,利用WES数据识别和表征细胞外染色体DNA(ecDNA)扩增,为进一步探索其临床意义提供基础。然后通过大规模临床样本分析,揭示ecDNA扩增在不同癌症类型中的特征模式,以及与临床分子特征的关联。探讨ecDNA扩增在结直肠癌分子分型和预后预测中的作用,为个体化治疗提供新的生物标志物。此外,还研究了ecDNA扩增与免疫检查点抑制剂治疗疗效的关系,为指导免疫治疗提供新的生物标志物,并进一步优化和改进基于WES数据识别ecDNA扩增的计算方法,以提高检测的准确性和适用性。综上所述,本研究为深入理解ecDNA在肿瘤发生发展中的作用,以及开发基于ecDNA的新型诊断和治疗策略提供了重要的基础。
1.构建基于机器学习的计算框架并实施染色体外DNA扩增鉴定
通过观察大量的数据,发现ecDNA通常具有较高的拷贝数,而拷贝数与测序读数的数量成比例变化,因此作者设计了一个基于机器学习的计算框架(简称为GCAP)来优化拷贝数原的特征工程,用于预测ecDNA扩增,并评估其性能和在临床癌症研究中的潜在应用(图1a)。研究使用XGBOOST算法训练机器学习模型,设计了11个特征变量,结果表明其性能远远高于仅使用基因总拷贝数的模型(图1c)。研究还在样本水平上测量了XGB11用于ecDNA扩增检测的性能评分,获得了auPRC(0.970)、auROC(0.863)、精度(0.988)、灵敏度(0.776)和特异性(0.950)的高指标(图1d)。
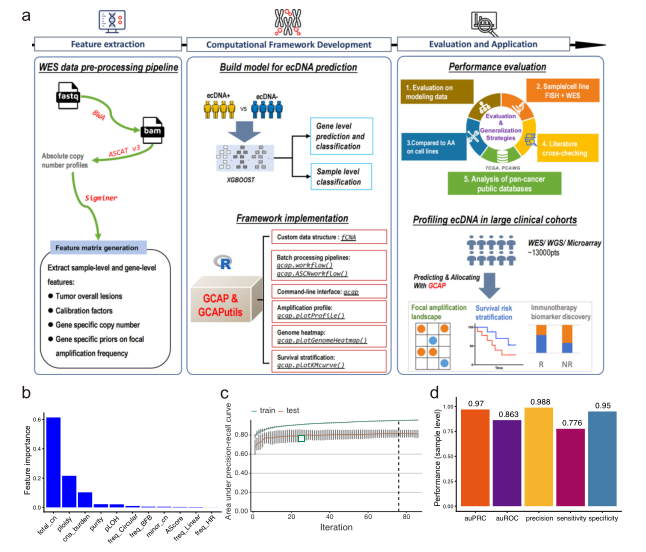
图1 面向癌症染色体外DNA扩增鉴定、评估、分析和应用的全外显子组测序数据框架
2.GCAP对染色体外DNA扩增鉴定的评估
研究使用来自不同癌细胞系的WES、WGS和Circle-Seq数据集,以及来自胃癌和结直肠癌的临床样本,以及验证性的实验和计算方法验证了GCAP的可靠性和实用性(图2)。结果显示GCAP不仅有助于检测癌症WES数据中的ecDNA扩增,而且还可以扩展到分析来自WGS和微阵列的拷贝数数据。
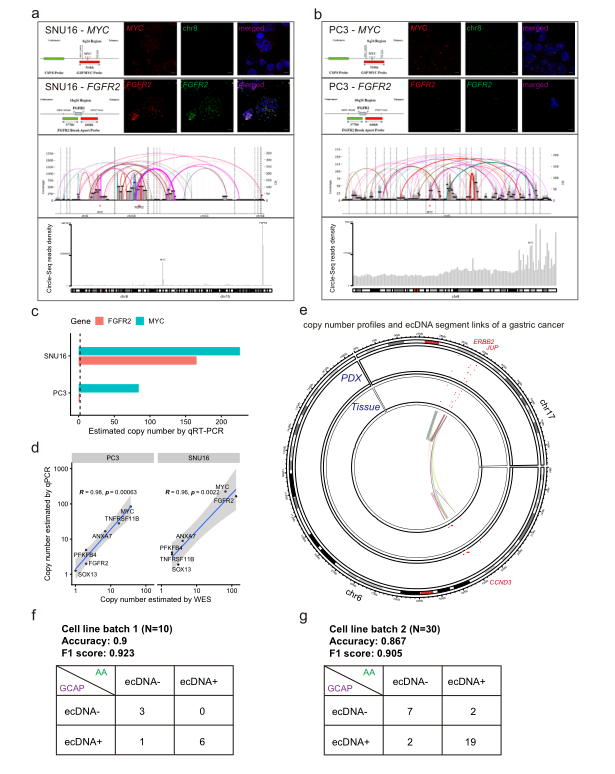
图2 染色体外DNA扩增鉴定对癌症细胞系基因组的评估
3.通过泛癌症分析重现与 ecDNA 相关的生存结果和基因组特征
研究先进行了样本水平的病灶扩大分型,然后比较了病灶扩大亚型(包括无病灶、非环状和环状)的生存结果和基因组特征,结果显示TCGA和PCAWG两个泛癌症数据库的分析结果一致,这进一步证实了GCAP是一种以癌症队列为导向的可靠方法,可用于识别 ecDNA 扩增的肿瘤,并从病灶扩增方面区分不同的肿瘤异质性。研究还发现两个数据库环状扩增和非环状扩增的全基因组分布模式是一致的。考虑 DNA 拷贝数时,染色体外扩增的癌基因比染色体内扩增的癌基因表现出更高的基因表达水平(图 4c),研究还揭示了在 ecDNA 阳性和 ecDNA 阴性肿瘤中表达差异最大的 50 个癌基因(图 4d) 。
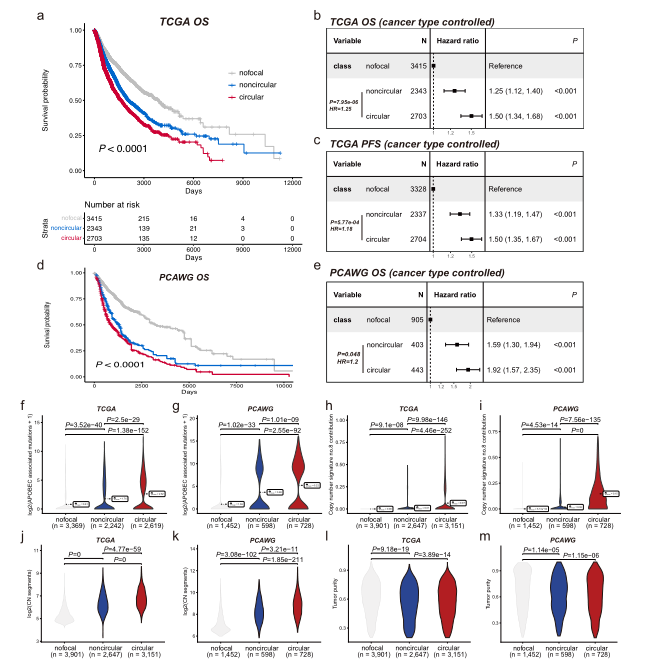
图3 在泛癌症数据库中一致地再现与 ecDNA 相关的生存结果和基因组改变模式
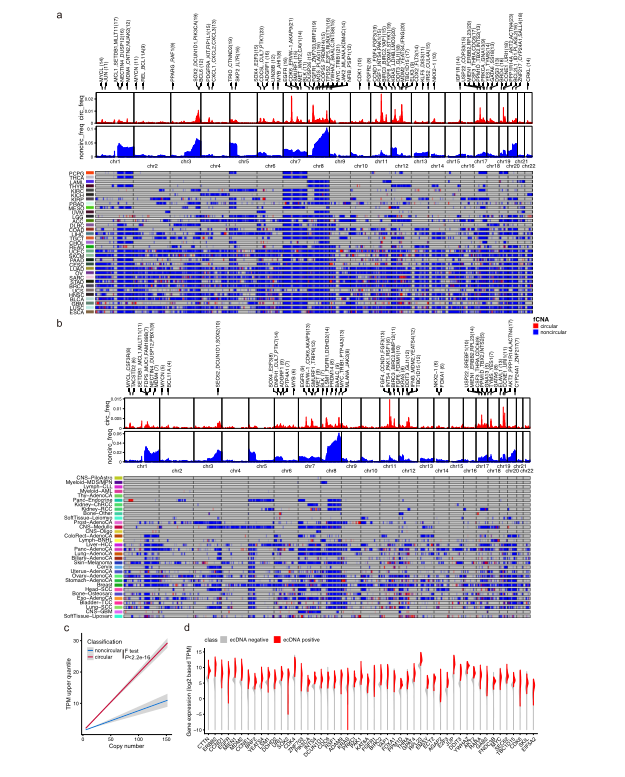
图4 病灶扩增和染色体外 DNA 相关癌基因的全基因组分布
4.ecDNA扩增是结直肠癌的独立危险因素
通过比较三种病灶Kaplan-Meier的生存曲线,发现其拷贝数扩增(fCNA)亚型的总生存期(OS)和无病生存期(DFS)都有不同的生存结果,其中环状亚型的风险最高(图5a)。通过将fCNA亚型与临床特征、预设的SYSUCC亚型等纳入多变量Cox回归分析,我们发现环状扩增(ecDNA+)是OS(P=0.01,危险比=1.57)和DFS(P<0.001,危险比=2.14)的独立生存风险因素,而非环状扩增不是(图5b)
图5(a ,b) 结直肠癌的局灶扩增分型预测患者的总体生存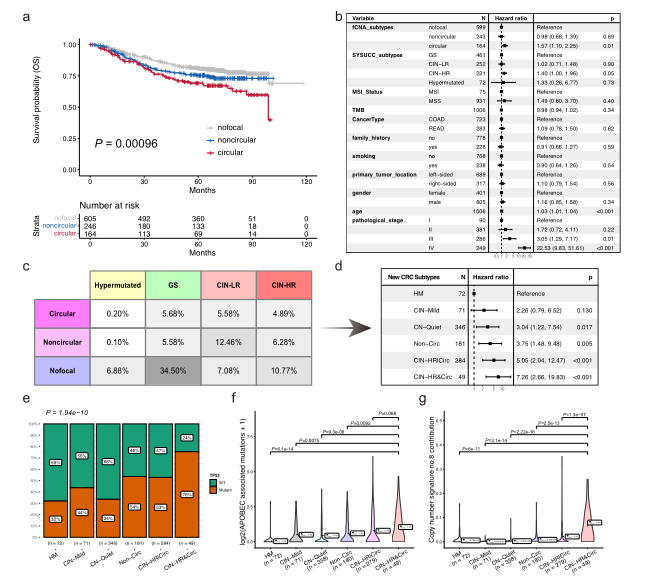
5.基于基因组的结直肠癌分子分型的改进
SYSUCC亚型和fCNA亚型的交集显示,GS亚组、CIN-LR亚组CIN-HR亚组CRC患者中,环状扩增的比例接近(图5c),表明染色体不稳定指数不能区分环状扩增肿瘤,因此研究重新定义并建立六个CRC基因组亚型(图5d),结果显示相较于HM,剩余的亚型的风险更高(图5d)。通过比较分析发现在以ecDNA扩增为特征的亚型中,与ecDNA相关的APOBEC 诱导突变和CN8活性富集,其中CIN-HR&Circ亚型表现出明显的富集(图5f,g),这为之前的泛癌分析数据提供了独立的验证。此外,研究还将基因组亚型进一步应用于TCGA胃肠癌数据集,结果表明,病灶扩增分型可单独用于鉴定基因组亚型,也可以通过补充更多的病灶基因组扩增变化来扩展现有的基因组亚型。
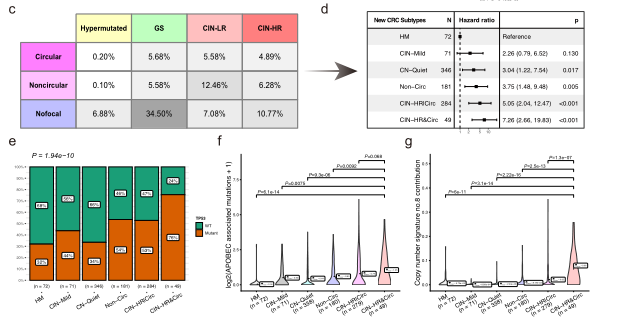
图5(c-g) 结直肠癌产生的具有不同突变过程的精细基因组亚型
6.ecDNA扩增对癌症免疫治疗具有预测和预后作用
通过 GCAP,作者利用前瞻性临床试验中的 WES 数据集,揭示 ecDNA 扩增与免疫检查点抑制剂(ICI)治疗反应之间的关系。在不同的癌症队列中,均有一定比例可归类于ecDNA扩增 。进一步分析了SYSUCC AGC和SYSUCC NPC队列中的总生存率,均有观察到有ecDNA扩增的癌症患者比没有ecDNA扩增的患者总生存率更低(图6a,b)。对两个队列进行的多变量Cox回归分析表明无论是否存在已知的免疫疗法生物标志物和临床变量(图 6c,d),圆形亚型(ecDNA+)仍然是导致生存率下降的统计学意义上的重要预后因素。细胞带水平的分析数据表明,ecDNA扩增可作为胃肠癌抗PD1单药治疗的阴性预测和预后生物标志物。在扩展病灶扩增分型分析中,发现在晚期 ESCC中,ecDNA扩增,尤其是与11q13相关的ecDNA扩增,对化疗+免疫疗法的敏感性高于单纯化疗。
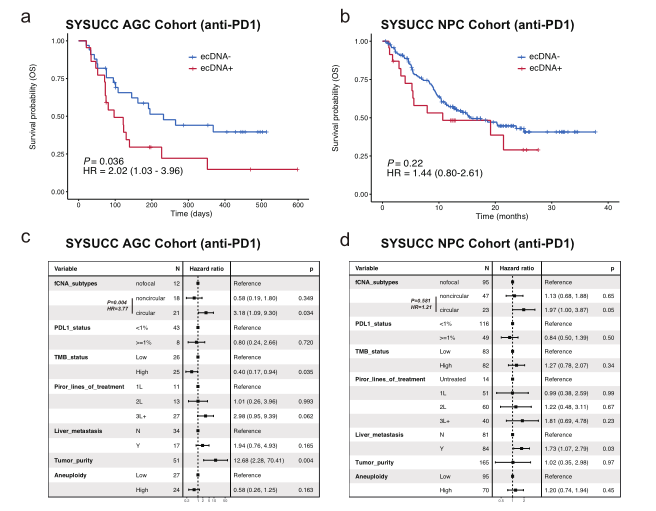
图6染色体外DNA扩增是抗PD1单药治疗总生存期的预后指标
到这里,文章的阅读就接近尾声啦!该研究基于机器学习开发了一个新的计算框架GCAP,并通过大数据和临床样本进行验证和不断的改进,可以对染色体外DNA进行识别和表征,有助于更好地理解ecDNA与癌症之间的关系,为癌症的治疗及后续的监测提供新方法。阅读完这篇文章,大家是否都对于自己的科研有了更多的新想法呢?生信技术发展愈发快速,只有掌握更扎实的生信技能,跟对热点方向,才能走在科研前列,快来扫码联系火火,这里有意想不到的前沿生信技术服务哦~不想落人一步,还不快联系火火获取思路设计!
生信火公众号持续为大家带来最新!最优!最赞的生信思路,想复现思路或者定制更多适合自己的创新性思路,欢迎直接扫码联系火火,生信火团队竭诚为您的科研助力!
定制生信分析
云服务器租赁
加好友备注“99”领取试用