
蛋白-蛋白间相互作用(protein-protein interactions,PPI)在几乎所有的生物学过程中都发挥着重要作用,包括信号转导、细胞生长和免疫防御等,其介导对细胞生理学重要的生物学功能。相互作用的蛋白质通过对突变的采样(主要是在蛋白-蛋白界面)共同进化了数千年,以实现所需功能的“最佳匹配”,这一过程很难在实验室中重现。蛋白质工程方法可以在蛋白质结合位点产生大量氨基酸库,用于筛选固定序列的其他蛋白质,反映了一半的进化过程。然而,通过使用“库中库”方法来回收匹配的共进化蛋白质对,开发两种蛋白质相互对抗的体外系统一直是一项挑战。一个有效的双向、同时进行蛋白-蛋白协同进化的合成系统可以作为模拟自然协同进化的平台。这也可能是一种为生物技术应用设计大量具有不同识别特性的蛋白-蛋白复合物的方法。
最近,斯坦福大学K. Christopher Garcia教授团队描述了一个合成蛋白-蛋白协同进化的平台,可以从复杂库中分离出相互作用的匹配对的突变蛋白。这个共同进化复合物的大型数据集推动了对Z结构域-粘附体对之间的分子识别的系统级分析,涵盖了广泛的结构、亲和性、交叉反应性和正交性,并捕获了广泛的共同进化网络。此外,作者利用预训练的蛋白质语言模型在计算机中扩展了协同进化筛选的氨基酸多样性,预测超出实验库范围的重塑界面。这些方法的整合为生物技术和合成生物学提供了一种模拟蛋白质协同进化和产生具有多种分子识别特性的蛋白质复合物的手段。相关研究成果以“Deploying synthetic coevolution and machine learning to engineer protein-protein interactions”为题发表在国际知名期刊Science上。

利用蛋白质复合物模型,生成了在疏水界面内六个位置氨基酸变化的合成蛋白质库。随后在在一个共同进化网络中绘制了这些序列,并确定了10对提供特异性细节的结构。最后使用预训练的蛋白质语言模型来扩展氨基酸对的范围,证明了这种混合实验-计算方法对该系统中蛋白-蛋白相互作用的预测能力。
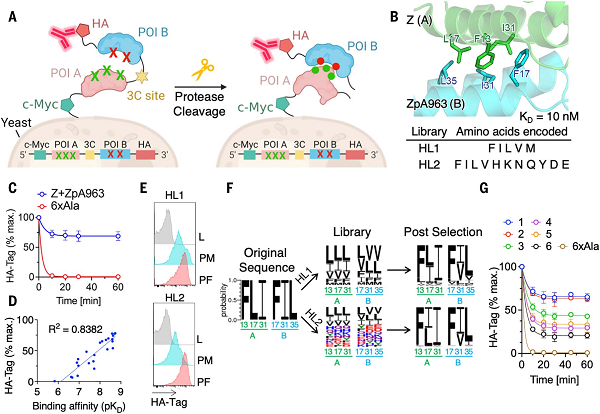
图1 蛋白-蛋白协同进化策略的设计和验证 © 2023 AAAS
(a)蛋白质-蛋白质协同进化工作流程示意图。
(b)Z结构域(绿色)和亲和体ZpA963(蓝色)(PDB:2M5A)疏水腔中关键残基的特写视图。
(c)相互作用对(Z+ZpA963)和非作用对(6×Ala)的酵母裂解捕获实验。
(d)酵母裂解捕获分析与表面等离子体共振技术(SPR)测量的Z结构域-亲和体二聚体突变体的结合亲和力之间的相关性分析。
(e)流式细胞仪分析的直方图。
(f)下一代测序(NGS)数据在本地库和荧光激活细胞分选(FACS)最后一轮之后的序列频率标志。
(g)从HL1和HL2 NGS数据中对六种最常见的突变体进行酵母裂解捕获分析。
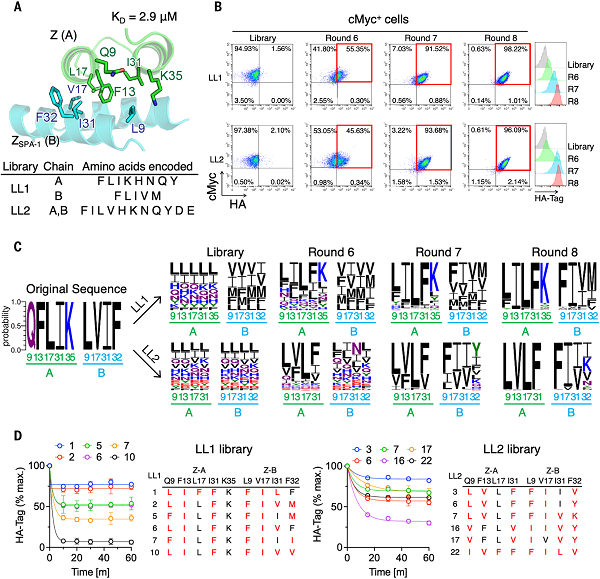
图2 通过共同进化工程重塑的二聚体界面 © 2023 AAAS
(a)(上)来自Z结构域(绿色,A链)和ZSPA-1(蓝色,B链)的络合物(PDB: 1LP1)的界面上库的位置。(下)用于制作两个独立文库LL1和LL2的编码氨基酸。
(b)(左)流式细胞术分析显示第6轮至第8轮后库中HA标签荧光的富集。(右)直方图显示在选择期间HA标签荧光的升高,从第6轮到第7轮和第8轮。
(c)本地库中NGS数据的序列频率标志,第6轮、第7轮和第8轮揭示了LL1和LL2库中选择过程中一致序列的出现。
(d)LL1(左)和LL2(右)库的突变体的酵母裂解捕获分析。
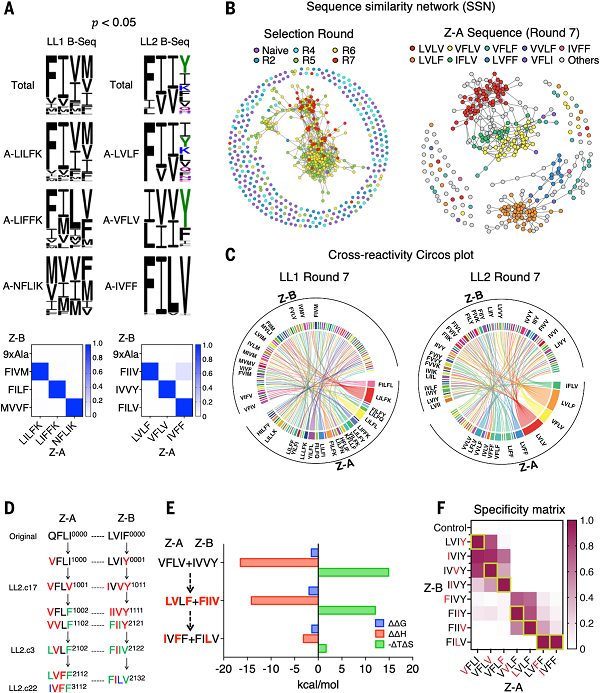
图3 协同进化网络的可视化和映射 © 2023 AAAS
(a)统计富集的NGS数据的Z-B序列与每个Z-A序列配对的序列标志,以及通过酵母裂解捕获测定测量的实际结合特异性,标准化为每个Z-A序列的最高亲和力。
(b)来自LL2库的所有筛选轮(左)和第7轮(右)的串联的8个氨基酸Z-A+Z-B库位置序列的SSN。
(c)LL1和LL2的100个样品对的Circos交叉反应性图。
(d)LL2库突变体的单一突变途径,连接原始序列(QFLI+LVIF)和显著的LL2库突变株。
(e)三种途径的突变体与原始序列相比的 G, H和– T S变化。
(f)显示来自该途径的Z-A变体的结合特异性变化的矩阵。
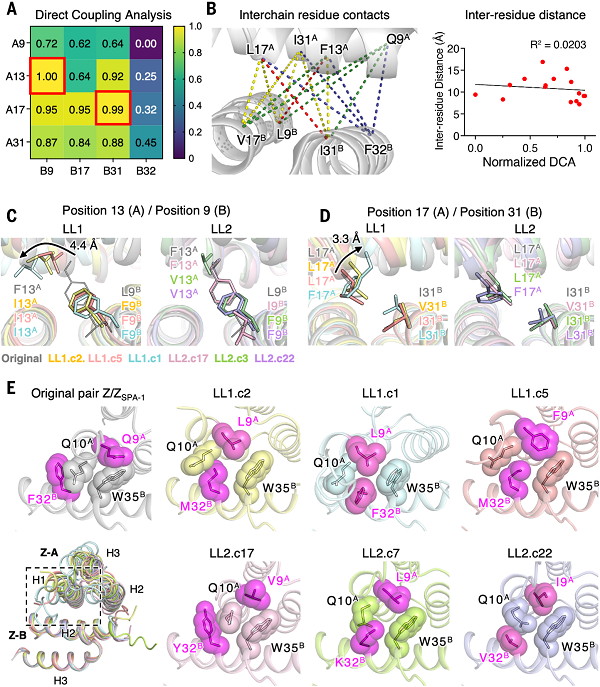
图4 共同进化变体的耦合分析和结构适应 © 2023 AAAS
(a)直接耦合分析(DCA)矩阵来预测LL2库序列的残基间共变。
(b)(左)残基间连接和(右)DCA与残基间距离的关系。
(c-e)库位置的特写视图,以显示局部侧链重排。
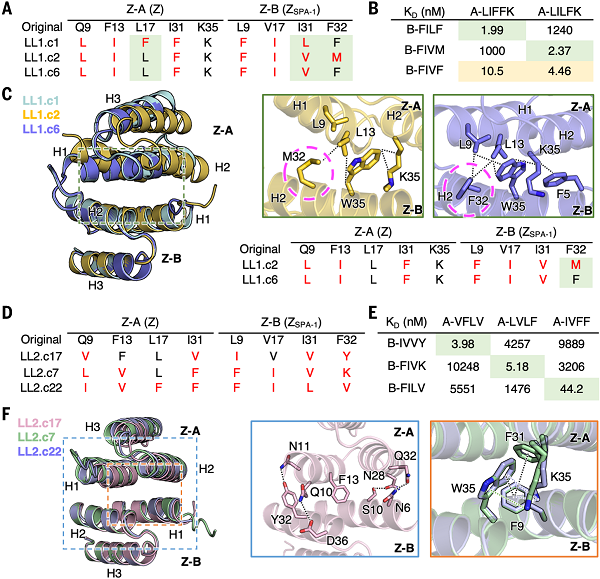
图5 正交高亲和力突变体的特异性决定簇 © 2023 AAAS
(a)与原始氨基酸相比,改变的位置用红色表示,突变体之间的变化位置用绿色方框突出显示。
(b)用SPR测定的Z-A和Z-B单体亲合力表。
(c)(左)LL1.c1,LL1.c2和LL1.c6结构的叠加。(右上)32B位置附近LL1.c2和LL1.c6结构的对比显示了单个突变M32BF如何通过Trp35B的侧链旋转和其周围疏水相互作用的增加来诱导大的构象变化。(右下)每个突变体的特写显示以trp35为中心的疏水相互作用与周围残基。
(d)显示三个正交LL2突变体LL2.c17(VFLV+IVVY)、LL2.c7(LVLF+FIVK)和LL2.c22(IVFF+FILV)的库位置中的氨基酸的表,以比较它们的亲和力和结构的差异。
(e)三个突变体的Z-A和Z-B突变体各组合的结合亲和力。
(f)LL2.c17与其他两个突变体在界面上存在的显著结构差异。
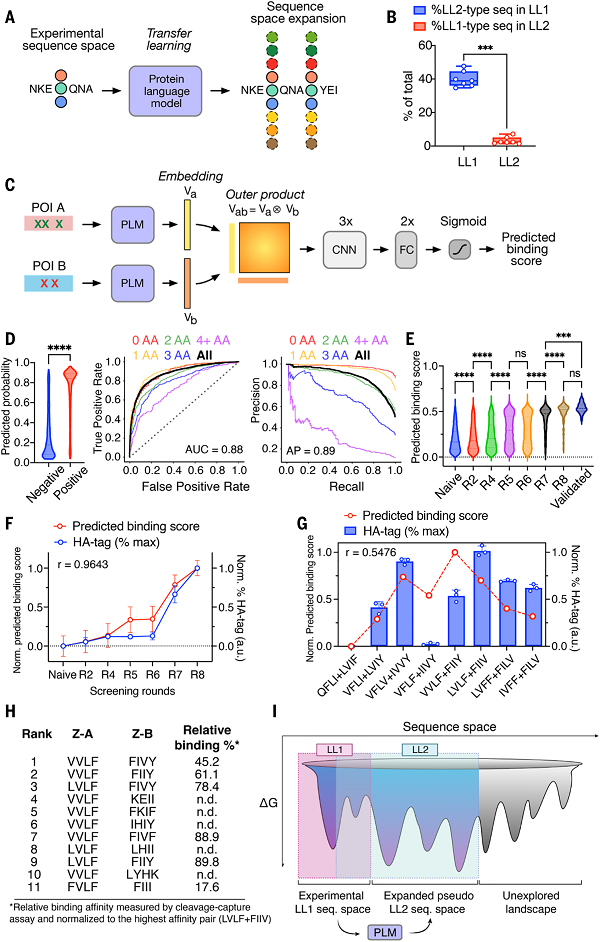
图6 利用蛋白质语言模型进行序列空间扩展 © 2023 AAAS
(a)通过蛋白质语言模型进行序列空间扩展的示意图。
(b)LL2测序数据中LL1型序列的部分,以及LL1测序数据中LL2型序列的部分。
(c)通过基于外产物的卷积神经网络(CNN)预测二聚体与扩展的氨基酸相互作用的方法图示。
(d)LL1训练模型在LL2测试集上的分类效率。
(e)每轮筛选LL2测序数据的预测结合分数用小提琴图表示。
(f)LL2测序数据的预测结合评分与蛋白酶切割后HA-tag MFI实际百分比的相关性。
(g)图3D中突变途径中预测的结合分数和配对的相对亲和力之间的相关性。
(h)根据LL2 NGS数据预测结合评分排名前11位的序列。
(i)通过蛋白质语言模型和迁移学习,描述了从实验LL1数据到预测LL2序列空间的序列空间扩展。
本研究开发了一种简单的蛋白质协同进化方法,以解决大规模库选择中连接表型与基因型的问题。生成的相互作用的Z结构域-粘附体对的大量集合使该模型系统中的分子识别的系统级结构-功能分析成为可能。在DCA和高分辨率晶体结构的基础上,可以成功地推断出Z结构域-粘附体二聚体界面之间的上位相互作用。本研究的共同进化策略产生的实验数据可以用作机器学习算法的训练数据,以扩展比实验获得的序列空间更宽的序列空间,并预测蛋白质-蛋白质的相互作用。
原文详情:Deploying synthetic coevolution and machine learning to engineer protein-protein interactions (Science 2023, 381, eadh1720)
本文由赛恩斯供稿。
cailiaorenVIP
