第一节课:Python系统讲解 ,jupyter notebook的环境搭建和基础知识讲解。第二节课:Linux系统讲解 ,Anaconda的安装和conda环境创建,rstudio-server的系统讲解。第三节课:以Nature文章为例系统讲解单细胞数据质控、归一化处理、PCA降维、聚类、tSNE、UMAP可视化第四节课:Scrublet去除双细胞【Science】,DoubletFinder | 使用ANN算法识别单细胞数据中的Doublet【Nature Medicine】
第五节课:CellTypist 免疫细胞自动注释【Science】,非负最小二乘回归的单细胞注释【Nature】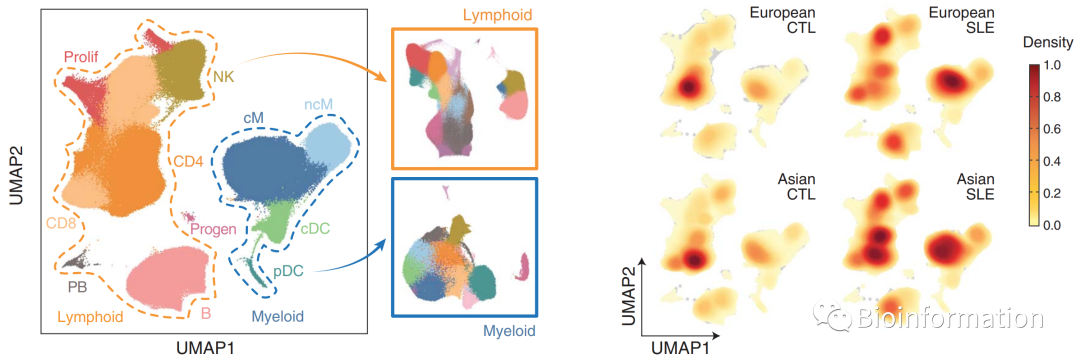
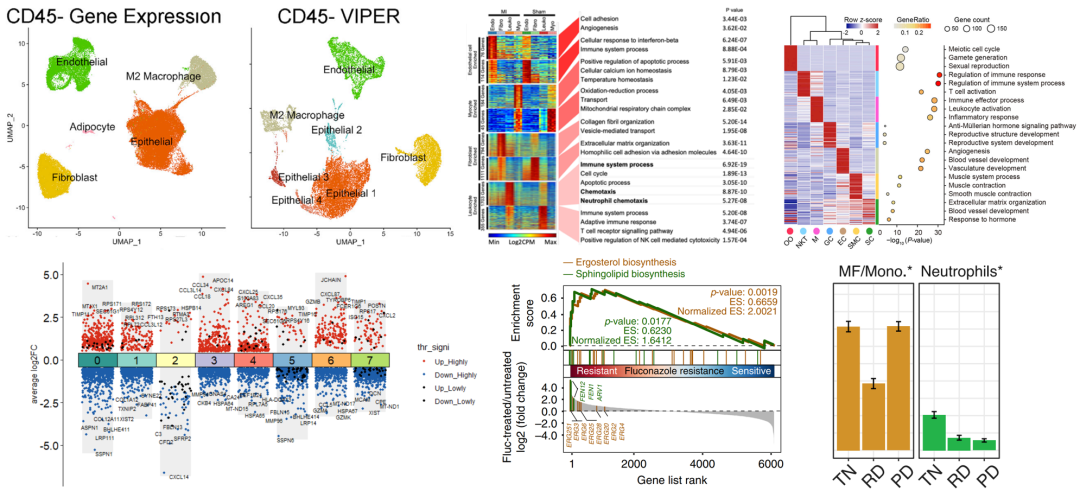
第六节课:利用VIPER算法来定量单细胞蛋白的活性
Cell:Single-cell protein activity analysis identifies recurrence-associated renal tumor macrophages
第七节课:使用VISION算法推断单细胞代谢活性【Cancer Discovery】
第八节课:展示不同分组marker基因展示,多个重复的细胞类型比例展示 【Cell】
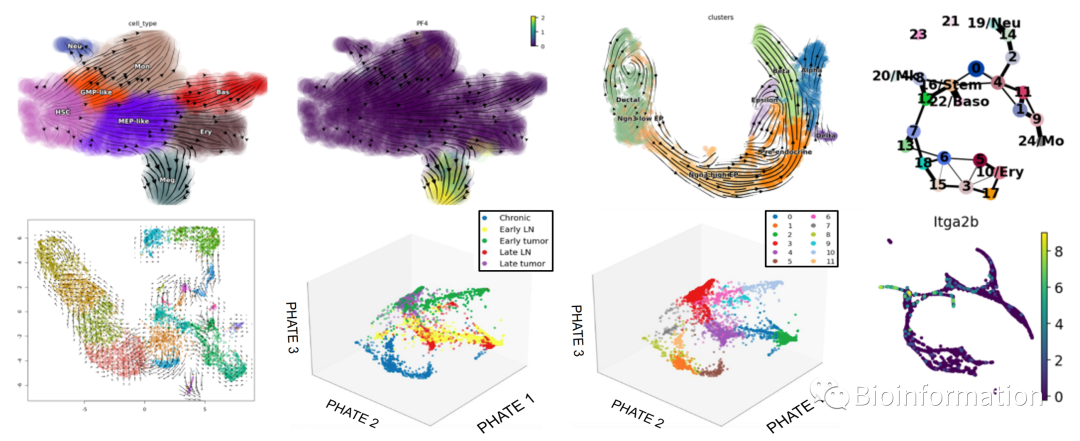
第九节课:CellRanger处理单细胞上游数据,loom文件生成,velocyto.R 分析RNA速率【Nature】
第十节课:scVelo三种模型来确定动态变化过程驱动基因【Nature Biotechnology】
第十一节课:将velocity 与PAGA联合分析【Nature】
第十二节课:Dynamo分析animating neural activity response predictions【Cell】
第十三节课:PHATE同时保留局部结构(local structure) 以及全局结构(global structure)对单细胞数据降维【Nature】
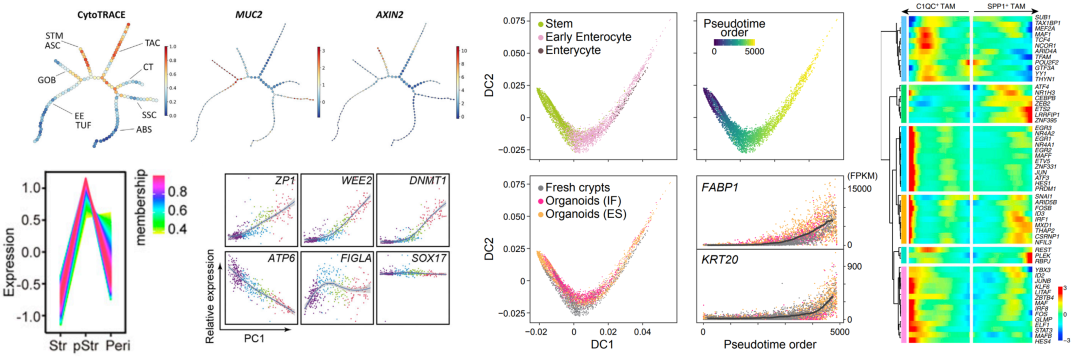
第十四节课:CytoTRACE进行拟时间分析,基因表达量随PC1轴的变化 【Cell】
第十五节课:Diffusion Pseudotime Analysis diffusion component【Cell Stem Cell】
第十六节课:Monocle2 和Monocle3系统讲解【Nature】,TCSeq分析基因随时间的变化【PNAS】
第十七节课:单细胞染色体拷贝数变异【Science】
第十八节课:CellPhoneDB v.3.0系统讲解细胞互作和个性化作图展示【Nature Genetics】
第十九节课:NMF鉴定不同的生物学功能【Cell】,不同cluster的异质性确定【Cancer Cell】
第二十节课:Mfuzz、 BioNet调控网络构建
Cell:Molecular Choreography of Acute Exercise(第五批学员为文章一作)
第二十一节课:使用SCENIC进行转录调控网络分析和核心驱动基因鉴定【Nature Medicine】
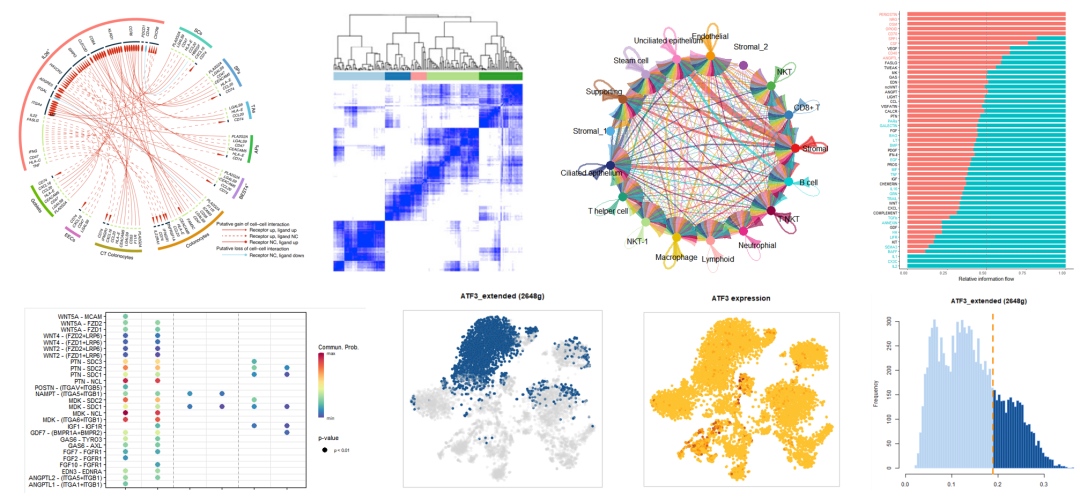
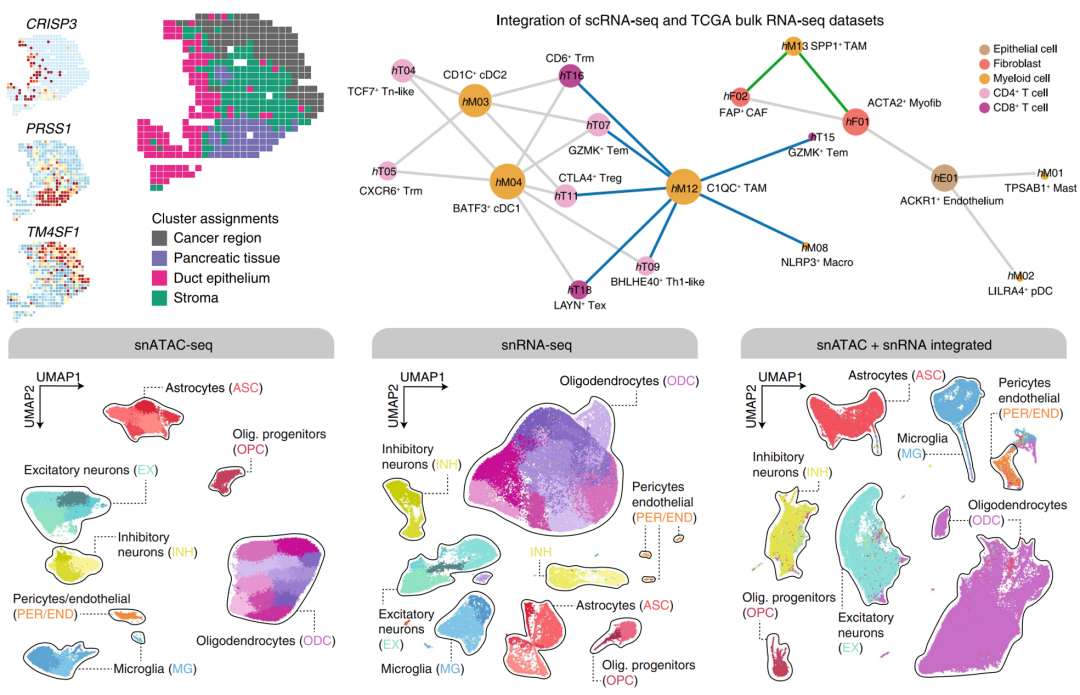
第二十二节课:scRNA-seq和bulk RNA-seq数据联合分析推断细胞类群互作网络【Cell】
第二十三节课:以Nature文章为例系统讲解单细胞空间转录组数据分析【Nature】
第二十四节课:以Nature文章为例系统讲解scRNA-seq与scATAC-seq联合分析【Nature Genetics】
第二十五节课:CITE–seq对单细胞转录组和膜蛋白进行定量分析【Nature Medicine】
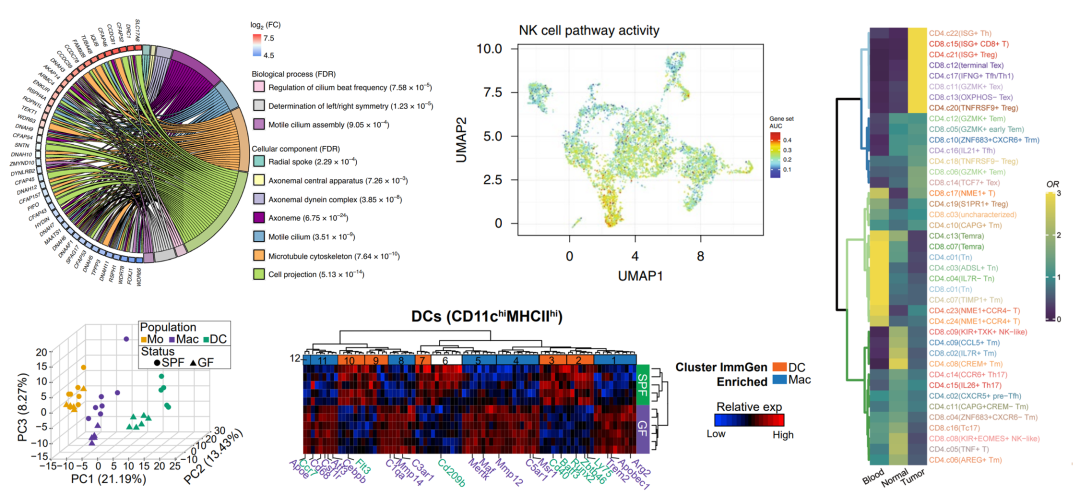
第二十六节课:富集分析、差异结果展示
26.1 圈图展示富集结果【Nature Medicine】
26.2 AUCell | 分析单细胞测序数据中不同基因集(通路)的活性【Nature Medicine】
26.3 Differences in pathway activities scored per cell by GSVA【Nature Medicine】
26.4 利用OR比值查看不同处理组的各单细胞亚群的分布差异【Science】
26.5 各个细胞类型的差异基因统一展示 【Cell】
26.6 单细胞测序不同分组的相同cluster差异程度3维PCA展示以及差异热图展示【Cell】
第二十七节课:MAGIC 利用流形学习还原单细胞的基因表达【Cell】