空间转录组的旅程让我们先停一下,来让我们的视线回顾到机器学习上,今天晨曦将与大家一起运用mlr3包来体验一下完整的机器学习流程并拿真实数据进行演示,相信给位小伙伴如果有耐心完整跟下来,相信一定会有所收获
本次我们的真实数据来源于Kaggle上一个很经典的比赛Titanic - Machine Learning from Disaster,首先我们的第一步自然就是要了解我们的数据来思考我们应该用什么模型来解决这个问题
目的:根据数据构建一个模型来预测在泰坦尼克号沉没的时候存活的情况
数据:
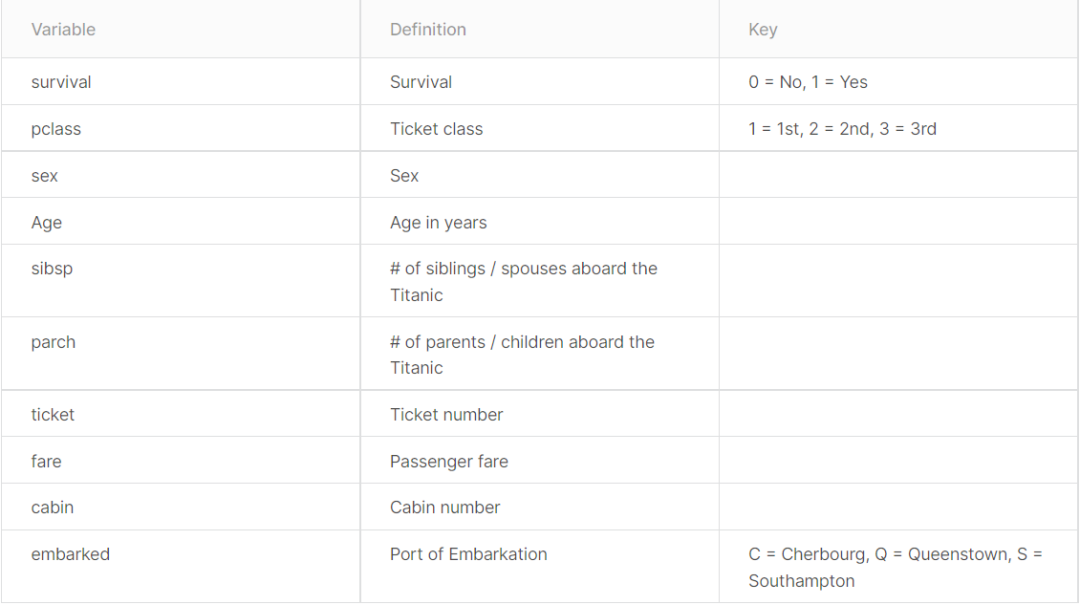
我们可以很清楚的看到,整个数据包含两大类,总共10个变量,分别是:
1. 响应变量:生存状态(0:死亡;1:生存)
注意:在进行模型构建的时候,我们的习惯是把我们感兴趣/阳性结局标记为1
2. 预测变量:
a. pclass-票价(反映了社会经济地位)(1:上流;2:中产;3:平民)
b. sex-性别
c. Age-年龄
d. sibsp-亲人的数量(兄弟、姐妹、同父异母的妹妹)
e. parch-家庭成员的数量(父母、儿女)
f. ticket-船票编号
g. fare-票价
h. cabin-船舱编号
i. embarked-出发港口
好,到这里我们就了解了我们的数据都是什么,那么接下来,我们就开始尝试用代码来进行模型的构建
注意:各位小伙伴可以思考晨曦在构建模型的过程中的一些思考,说不定可以给你一丢丢帮助哦~
#准备工作library(tidyverse)library(mlr3verse)library(paradox)library(knitr)library(dplyr)library(Hmisc)
###2.准备数据####train_org = read_csv("Kaggle/Titanic - Machine Learning from Disaster/train.csv")test_org = read_csv("Kaggle/Titanic - Machine Learning from Disaster/test.csv")
train_org

test_org

晨曦解读
注意,我们下载的数据比kaggle界面介绍的数据会多出几个变量,但是无关紧要(后续会删掉),然后测试集上是没有响应变量的
#Exploratory Data Analysis(EDA)Hmisc::describe(train_org)skimr::skim(train_org)
使用skim函数探索数据的结果如下:
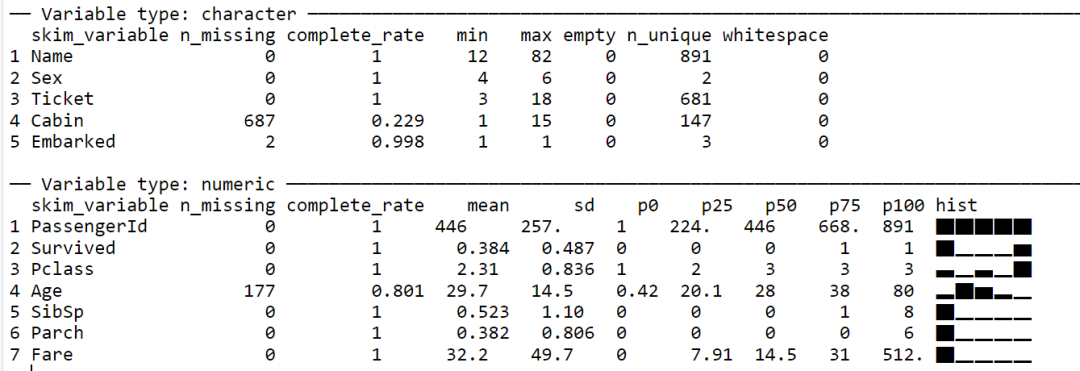
晨曦解读
两种探索数据的函数,describe函数提供的信息更加全面,skim函数提供的信息则相对精简,各有所长,选择一种即可
#特征选择#首先我们需要剔除一些依照我们朴素的感觉觉得没有什么关系的预测变量train % select(-c(PassengerId,Ticket,Cabin))#上述的三个预测变量和响应变量之间并没有什么关系,比如说船舱编号,船到都沉没了,你在哪个船舱能够幸免?#至于乘客ID因为和响应变量存在绝对的关系,因为乘客编号只对应一个乘客,然后这个乘客的死或者生是肯定的,所以对于这种编号类型的数据我们也是主张剔除
train
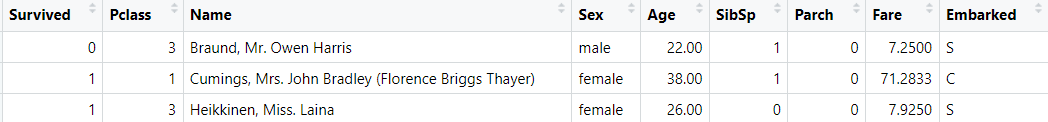
晨曦解读
然后这个时候我们再来想一个问题,名字这个预测变量重不重要?很显然你叫什么名字并不能够让你在还难中幸存下来,所以这个时候我们完全可以把名字变量直接去掉,但是,如果想要考虑的更深入,我们可以来观察一下名字这个变量
Braund,Mr.Owen Harris
Cumings,Miss.Laina
上述这两个名字很显然包含了一些重要信息——性别(或者说是状态——Mr:先生;Mrs:夫人;Miss:女士),而且很显然据我们的背景知识可以知道,泰坦尼克号沉没的时候,是首先让女性上了救护船,所以绝大多数幸存的是女性,那么这里面的信息很显然就和我们的响应变量有关系了,那么我们自然是需要提取这些信息的
#特征选择eq identity train$identity = sapply(identity, function(x) x[1])table(train$identity)

晨曦解读
这个时候我们又会面临一个问题,就是一个预测变量下含有太多的分类,因为我们在平时构架预测模型的时候,一个预测变量下的分类变量我们都是最好设置为两个(男或者女,生存或者死亡)因为这样才是最好计算且最合适的,所以我们这个分类变量的数目太多了,所以我们需要合并一下(简单来说就是把出现频率低的给直接合并掉)
eq = as.data.frame(table(train$identity))other_identity = eq[which(eq$Freq < 10), "Var1"]train[which(train$identity %in% other_identity), "identity"] train$identity = as.factor(train$identity)train % select(-Name)table(train$identity)

晨曦解读
这个时候看起来就会舒服很多,然后我们再次通过select函数把Name变量去掉,然后我们的数据集就被整理成了下面这种形式
然后我们需要按照我们整理数据的流程把测试集也整理一下,也就是上面步骤的一个复刻
#整理测试集数据test % select(-c(PassengerId,Ticket,Cabin))eq_test identity test$identity = sapply(identity, function(x) x[1])table(test$identity) eq_test = as.data.frame(table(test$identity))other_identity = eq_test[which(eq_test$Freq < 10), "Var1"]test[which(test$identity %in% other_identity), "identity"] test$identity = as.factor(test$identity)test % select(-c(Name)) table(test$identity)
test数据如下:
晨曦解读
到这里我们数据内容上就整理完毕了,我们做到了每一个预测变量我们都知道是什么意思,以及每一个预测变量内的数据都是和响应变量具有相关性的,那么我们接下来还需要考虑最后一点就是数据的形式
注意:模型本质上来说其实就是数学公式的具体化,所以依照我们最朴素的理解来说,其预测变量需要时数值型变量,那么为什么有的时候我们选择分类变量也可以呢,是因为R包自动帮助我们进行了转换,也就是所谓的哑变量,但是我们最好先把分类变量转化为因子的形式,这一点需要注意
#转换数据类型test$Embarked = as.factor(test$Embarked)test$Sex = as.factor(test$Sex)train$Embarked train$Sex train$Survived
至此,我们的数据整理就结束了:内容+格式,以后我们每一次整理数据的时候只要从这两方面入手绝大部分问题都会被我们提前避开掉
那么下面我们就开始进行mlr3包的整体流程
Ps:各位小伙伴如果感兴趣mlr3包的具体细节话,欢迎在评论区留言,点赞多就加更哦~

lrn "classif.rpart",predict_type = "prob")lrn$param_set
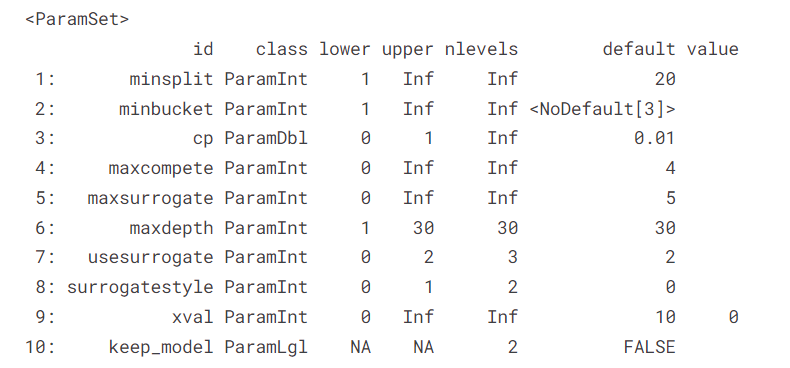
晨曦解读
机器学习相对于我们普通的三大模型(线性、logistic、cox)其最大的魅力可能就是调节超参数了,我们可以通过调节超参数来让模型的性能得到更进一步的提高,当然这里我们先不着急调节超参数,我们再来探索一下我们创建的学习器里还有什么其它内容
#获取学习器拥有的特性lrn$properties#[1] "importance" "missings" #[3] "multiclass" "selected_features"#[5] "twoclass" "weights"
晨曦解读
这个意思就是学习器(随机森林)本身可以支持变量重要性筛选、缺失值的处理、多个响应变量、特征选择(自身)、二分类响应变量、权重等
注意:没有任何一个算法天生支持缺失值的处理,都是作者自行定义了出现缺失值的情况,比如说来源于rpart包的随机森林作者就定义了缺失值出现时应该如何,所以这个算法就支持了缺失值处理
#查看学习器lrn#: Classification Tree#* Model: -#* Parameters: xval=0#* Packages: mlr3, rpart#* Predict Type: prob#* Feature types: logical, integer,# numeric, factor, ordered#* Properties: importance, missings,# multiclass, selected_features, twoclass,# weights
晨曦解读
通过前面的解释,现在学习器的内容我们大致就可以看懂了,然后接下来我们就需要拟合任务开始构建模型
lrn$train(task)print(lrn$model)
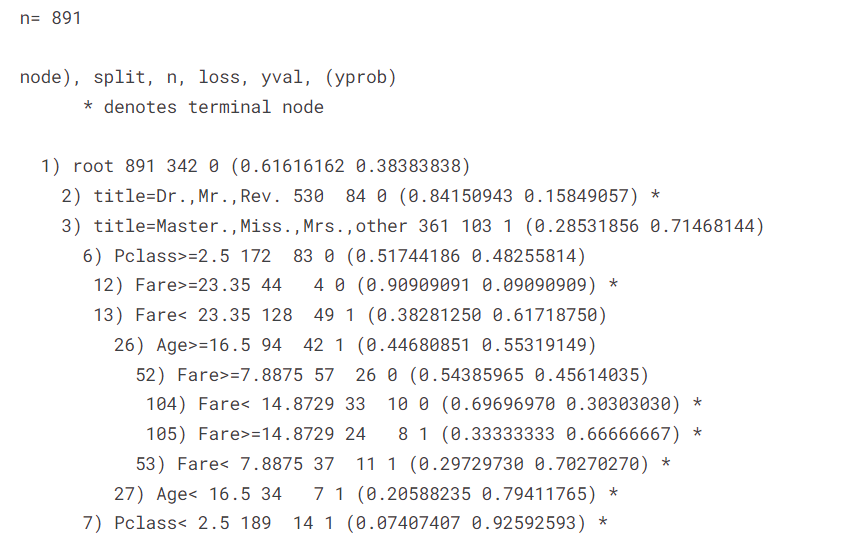
pred $predict(task)pred$confusion
measure "classif.acc","classif.ce"))pred$score(measure)
晨曦解读
注意,我们这个时候使用训练集构建模型,又用训练集测试模型,这个时候的模型评价指标属于“王婆卖瓜自卖自夸”,但是,虽然不是很准确,但是我们也已经大概知道了这个模型的一个性能评价,那么我们接下来就需要调节超参数
resampling "cv",folds = 10)measure "classif.acc")search_space cp = p_dbl(lower = 0.001,upper = 0.1), minsplit = p_int(lower = 1,upper = 100))learner.tuned = AutoTuner$new( learner = lrn, resampling = resampling, measure = measure, search_space = search_space, terminator = trm("evals", n_evals = 100), tuner = tnr("random_search"))learner.tuned$train(task)learner.tuned$tuning_resultlearner.tuned$predict(task)$confusion
晨曦解读
注意,我们这里的10折交叉验证是针对超参数调优的,最后获得的模型是模型性能评价指标最优下超参数的组合
pred $predict_newdata(test)
晨曦解读
好,到这里我们就通过mlr3完成了整体的模型构建再到预测的部分,相信各位小伙伴经过这一趟流程下来,对于机器学习以及mlr3包的一些细节应该是有所体会了
随后的几期推文都是围绕着机器学习和空间转录组
Ps:取决于晨曦写完了哪篇.......呜呜QAQ
各位小伙伴还有感兴趣的内容也可以在评论区提问,如果恰好是晨曦有所了解的,也会写成推文来和各位小伙伴一起学习
那么这期推文到这里就结束了~
我是晨曦,我们下期再见
Ps:回复“kaggle1"可以得到本期推文的示例代码以及示例数据哦~
晨曦的空间转录组笔记系列传送门
1. 拿去耍!!空间转录组实战来了!你在实验室的装逼利器!
2. 来领你的空间转录组救急包!你的装逼速成教程来了!
3. 新贵分析!单细胞联合空转分析,R语言手把手教学,你学废了吗?
晨曦单细胞文献阅读系列
晨曦单细胞笔记系列传送门
1. 首次揭秘!不做实验也能发10+SCI,CNS级别空间转录组套路全解析(附超详细代码!)
2. 过关神助!99%审稿人必问,多数据集联合分析,你注意到这点了吗?
3. 太猛了!万字长文单细胞分析全流程讲解,看完就能发文章!建议收藏!(附代码)
4. 秀儿!10+生信分析最大的难点在这里!30多种方法怎么选?今天帮你解决!
5. 图好看易上手!没有比它更适合小白入手的单细胞分析了!老实讲,这操作很sao!
6. 毕业救星!这个R包在高分文章常见,实用!好学!
7. 我就不信了,生信分析你能绕开这个问题!今天一次性帮你解决!
晨曦单细胞数据库系列传送门
1. 宝儿,5min掌握一个单细胞数据库,今年国自然就靠它了!(附视频)
2. 审稿人返修让我补单细胞数据咋办?这个神器帮大忙了!
3. 想白嫖、想高大上、想有高大上的SCI?这个单细胞数据库,你肯定用得上!(配视频)
4. What? 扎克伯格投资了这个数据库?炒概念?跨界生信?
5. 不同物种也能合并做生信?给你支个妙招,让数据起死回生!
6. 零成本装逼指南!单细胞时代,教你用单细胞数据库巧筛基因,做科研!
7. 大佬研发的单细胞数据库有多强? 别眼馋 CNS美图了!零基础的小白也能10分钟学会!
8. 纯生信发14分NC的单细胞测序文章,这个北大的发文套路,你可以试下!实在不行,拿来挖挖数据也行!
9. 如何最短时间极简白嫖单细胞分析?不只是肿瘤方向!十分钟教你学会!
10. 生信数据挖掘新风口!这个单细胞免疫数据库帮你一网打尽了!SCI的发文源头!

欢迎大家关注解螺旋生信频道-挑圈联靠公号~