临床样本采集
本研究主要利用生物信息学方法,并采用临床样本来验证一些感兴趣的发现。本研究以健康牙龈组织标本为对照组。招募了24名签署知情同意书的参与者,包括12名患有全身性中度或重度牙周炎(II期和III期,B级)的患者和12名需要进行冠延长手术的牙周健康患者。局部麻醉后,在重庆医科大学口腔医院进行约2 mm³的牙龈组织活检。活检从颊龈缘获得,所有程序在无菌条件下进行。从接受冠延长手术的个体和牙周炎患者严重炎症和骨质流失的区域进行牙龈活检。共采集
组织24片,每3对(包括健康牙龈组织3片和牙周炎牙龈组织3片)组织分别用于定量实时荧光定量pcr(qRT-PCR)、免疫印迹(WB)、苏木精-伊红(HE)和免疫组化(IHC)分析
组织学分析
收集牙周炎患者和健康对照者的牙龈组织,依次在4%的缓冲福尔马林中固定48小时。随后,用分级乙醇脱水,石蜡包埋,切成5 μm的切片。对切片进行HE染色,以便进行组织学分析。
免疫组织化学分析
使用4%多聚甲醛溶液固定牙龈组织样品24小时,然后按照上述步骤脱水、包埋。细胞色素c是线粒体呼吸链的重要组成部分,其释放受Bcl-2蛋白的调节。鉴于观察到线粒体呼吸链和免疫细胞之间的显著相关性,细胞色素c被选为感兴趣的标记物。为了研究这两个经典标记在人类牙龈组织中的表达,进行了免疫组织化学染色。
免疫印迹
进行蛋白提取和western blot分析。牙龈组织样品首先在液氮中粉碎,然后在RIPA裂解缓冲液中裂解,并加入蛋白酶抑制剂。样品的蛋白浓度采用增强型BCA蛋白测定试剂盒测定。随后,将30 μg的蛋白质加载到15%的十二烷基硫酸钠聚丙烯酰胺凝胶上并电泳。然后将蛋白质转移到聚偏二氟乙烯膜上,然后用5%的牛血清白蛋白(BSA)在室温下阻断2小时。然后将膜与一抗、细胞色素C和β-肌动蛋白在4°C下孵育过夜。用TBST洗涤后,用酶标二抗室温孵育2小时。采用增强型化学发光试剂和ChemiDoc™MP成像系统对靶蛋白结合进行可视化。
定量实时PCR
手术切除后立即将新鲜组织放入RNA保存剂中,然后在4°C下保存过夜,随后在- 80°C下保存,直到提取RNA用于PCR分析。从6例患者中收集6块牙龈组织,其中3块来自对照组,3块来自牙周炎组。
从组织样本中分离总RNA,并使用NanoDrop ND-1000分析仪进行定量。GoScript™逆转录混合试剂(Promega)用于将RNA逆转录为cDNA。在CFX96 qRT-PCR检测系统上,使用TB Green Premix Ex Taq II(Tli RNaseH Plus)(2×)(Takara编号RR820A)以及针对两个关键失调的线粒体相关基因(DMRGs),即CYCS和BCL2的引物进行qRT-PCR检测,同时以GAPDH作为内参基因。该实验重复进行了三次。通过公式计算倍数变化(FC)。
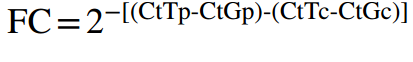
公共数据的收集和准备
在基因表达数据库(GEO)获取数据,包括微阵列数据集(GSE16134、GSE23586、GSE10334和GSE106090),RNA测序数据集(GSE173078)以及单细胞RNA测序数据集(GSE164241和GSE152042)。为了解决重复的基因符号问题,计算GSE16134、GSE23586和GSE10334的基因表达值为中位数值。接下来,使用R软件包“inSilicoMerging”将微阵列数据集合并,然后通过R软件包“sva”中的ComBat方法去除批次效应。随后,将合并后的数据集随机划分为训练队列(n=394)和测试队列(n=169)。对于单细胞RNA测序数据集,使用R软件包“Seurat”来执行诸如质量控制、标准化、整合、批次校正、主成分分析、细胞聚类、均匀流形近似和投影(UMAP)降维、数据集整合等各项任务,并借助R软件包“single R”基于免疫细胞的流式细胞术纯化转录组数据集(MonacoImmuneData)对细胞类型进行注释。
DMRGs的鉴定
使用R软件包“limma”评估训练队列中的差异表达基因(DEGs),统计显著性由p值<0.05和|log₂倍数变化(FC)|>0.5确定。从MitoCarta3.0数据库中获得了1136个基因和149条有强烈线粒体定位支持的线粒体途径(MitoPathways)。通过将1136个线粒体相关基因和DEGs进行交集,确定了牙周炎中的差异表达线粒体相关基因(DMRGs)。然后,使用R软件包“factoextra”(https://cloud.r-project.org/package=factoextra/)对DMRGs进行主成分分析(PCA),以基于前两个主成分在健康组和牙周炎组中样本的聚类情况可视化。随后,使用sangerbox对DMRGs进行GO/KEGG/线粒体(Mitopathway)途径功能富集分析,并通过R软件包“circlize”进行可视化。
Hub DMRGs的识别
使用了五种独立的机器学习算法来筛选出牙周炎中的关键差异表达线粒体相关基因(DMRGs),包括单变量逻辑回归、最小绝对收缩和选择算子(LASSO)回归、支持向量机递归特征消除(SVM-RFE)、XGBoost和随机森林。单变量逻辑回归通过R软件包“glmnet”进行,选取p值<0.05的基因为逻辑回归诊断基因候选。LASSO回归也通过“glmnet”进行,通过十折交叉验证生成诊断基因候选。SVM-RFE通过R软件包“e1071”进行,通过训练队列训练样本,对基因候选的得分进行排序,并估计前20个基因组合的十折交叉验证误差。选择具有最少十折交叉验证误差的基因组合作为SVM-RFE生成的诊断基因候选。XGBoost通过R软件包“XGBoost”进行,选取具有前20个特征的基因为诊断基因候选。使用随机森林软件工具构建了随机森林模型,并选取具有前20个基尼系数的DMRGs作为诊断基因候选。最后,基于上述五种机器学习算法筛选出的重叠DMRGs被选为牙周炎中的关键线粒体相关基因(MRGs),并进行了可视化。
人工神经网络模型的构建与验证
根据综合机器学习算法选出的关键MRGs,使用R软件包“neuralnet”以训练队列为基础开发了一个人工神经网络(ANN)模型。处理过的训练数据被输入到ANN模型中。该ANN模型设计为包含两个输入层、四个隐藏层和两个输出层。为了防止过拟合并优化模型,使用R软件包“Caret”进行了五折交叉验证。此外,为了评估ANN模型的稳健性和临床影响,进行了受试者工作特征(ROC)分析和决策曲线分析(DCA)。随后,在测试队列和其他外部验证数据集中验证了关键MRGs,并使用单细胞RNA测序(scRNA-seq)数据集分析了关键DMRGs的细胞特异性表达水平。
牙周炎免疫特性的探讨
根据综合机器学习算法选出的关键MRGs,使用R软件包“neuralnet”以训练队列为基础开发了一个人工神经网络(ANN)模型。处理过的训练数据被输入到ANN模型中。该ANN模型设计为包含两个输入层、四个隐藏层和两个输出层。为了防止过拟合并优化模型,使用R软件包“Caret”进行了五折交叉验证。此外,为了评估ANN模型的稳健性和临床影响,进行了受试者工作特征(ROC)分析和决策曲线分析(DCA)。随后,在测试队列中验证了关键MRGs。
线粒体呼吸链和线粒体病理通路的生物信息学评价
从MITOMAP数据库中提取线粒体呼吸链相关基因,采用Spearman相关分析检测线粒体呼吸链与枢纽DMRGs的相关性。此外,利用单样本GSEA (ssGSEA)方法测定每个标本中149条mitpathways的活性,并选择12条最活跃的mitpathways和12条最受抑制的mitpathways进行进一步分析。
WGCNA
根据它们的平均表达值,选取了排名前5000的mRNAs,并利用R软件包加权相关网络分析(“WGCNA”)来识别与MitoPathways相关的模块中的基因(教程可参考:一文搞定单细胞/空间转录组的WGCNA分析)。构建基因树状图时,最小基因组大小设定为30,并确定了16的幂次。使用R软件包“ggplot2”来可视化模块特征基因与MitoPathway之间的相关性。根据最高的相关系数和显著的p值,识别出与最活跃的MitoPathways相关的关键模块。在关键模块中,Module Membership(MM)>0.8且基因重要性(GS)>0.1的与MitoPathway相关的基因被视为显著基因。此外,还对这些基因进行了基于KEGG的功能富集分析(手把手教你做单细胞测序数据分析|7基因集富集分析)。
揭示牙周炎的线粒体表型模式
作者使用R软件包“ConsensuClusterPlus”基于41个DMRGs表达谱的无监督聚类分析来识别与线粒体相关的牙周炎亚型。这种新颖的亚型分类策略已在肿瘤学领域得到应用,并可能为临床管理提供一些新的见解。为了确保聚类的稳健性,进行了1000次迭代,每次迭代包含80%的样本。根据共识得分的累积分布函数(CDF)曲线确定最佳聚类数量。为了探索牙周炎的免疫和线粒体特征,检查了以下指标:估计免疫细胞丰度、MitoPathway活性、Reactome通路活性以及线粒体呼吸链基因表达状态,这些指标得到了R软件包“jjPlot”(https://github.com/junjunlab/jjPlot)的验证。最后,通过R软件包“ggcor”研究了在不同牙周炎亚型中关键基因与mtDAMPs之间的相关性。mtDAMPs及其相应的PRRs是从先前的文献中提取的。