纳米抗体Nanobodies,可以提供不同抗原的特异性结合。近年来,产生了许多有前景的治疗和检测应用。传统的基于羊驼免疫和噬菌体的纳米抗体发现技术,耗时耗力。尽管最近在纳米抗体研究方面取得了进展,但迫切需要开发快速准确的计算工具,以预测纳米抗体-抗原相互作用nanobody–antigen interaction (NAI) 。今日,清华大学自动化系Juntao Deng,刘民Min Liu等,在Nature Machine Intelligence上发文,提出了基于集成深度学习的框架,称为DeepNano-seq ,用于从纯序列信息中,预测包含纳米抗体-抗原相互作用NAI的一般蛋白质-蛋白质相互作用protein–protein interaction (PPI)。
定量比较表明,在现有蛋白质-蛋白质相互作用PPI算法中,DeepNano-seq具有最好的跨物种泛化能力。然而,几种最有效的蛋白质-蛋白质相互作用PPI方法,包括DeepNano-seq,由于在模式和数据水平上,纳米抗体-抗原相互作用NAI和蛋白质-蛋白质相互作用PPI的区别,表现出了对纳米抗体-抗原相互作用NAI预测的次优性能。
因此,从公共数据库中组织纳米抗体-抗原相互作用NAI数据,用于专门纳米抗体-抗原相互作用NAI建模。此外,通过基于提示的方法,将模型的注意力引导到抗原结合位点,以呈现最终的DeepNano,从而增强了DeepNano-Seq的预测流程。
综合评价表明,DeepNano在纳米抗体-抗原相互作用NAI预测和纳米抗体虚拟筛选方面,具有较好的性能。总的来说,DeepNano-seq和DeepNano可为纳米抗体的发现提供强大的工具。
Nanobody–antigen interaction prediction with ensemble deep learning and prompt-based protein language models.基于集成深度学习和提示的蛋白质语言模型,预测纳米抗体-抗原相互作用。
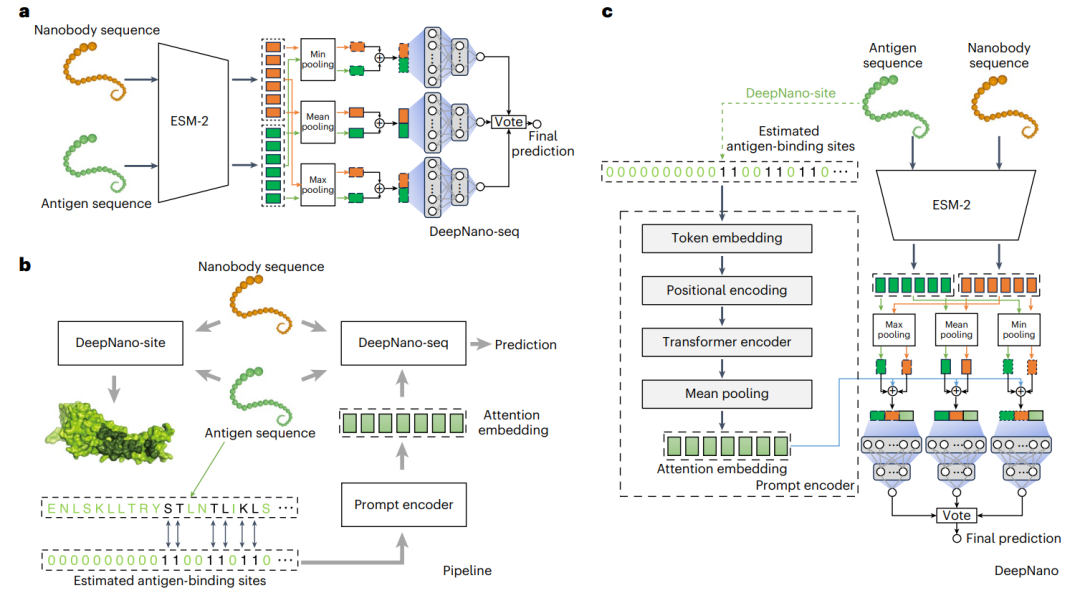
图1: DeepNano-seq和DeepNano概述。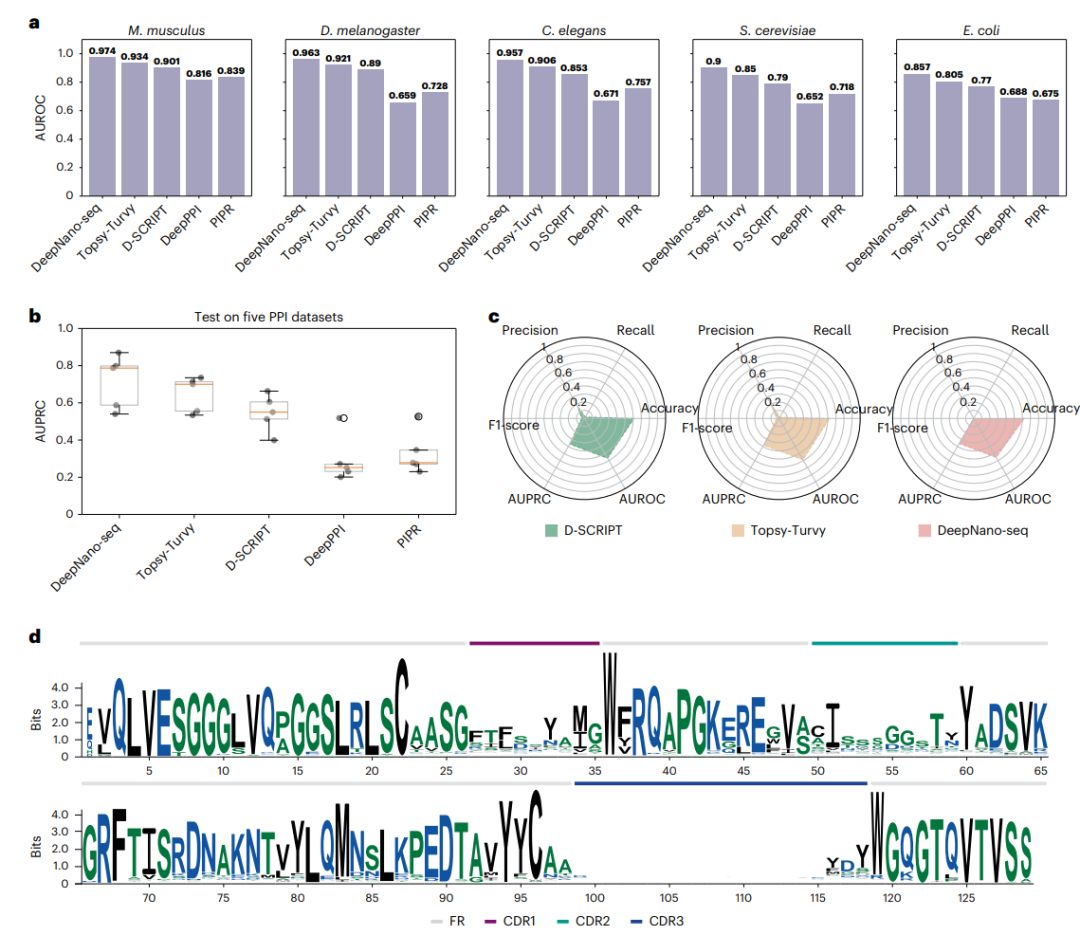
图2: DeepNano-seq方法与现有蛋白质-蛋白质相互作用protein–protein interaction,PPI算法比较。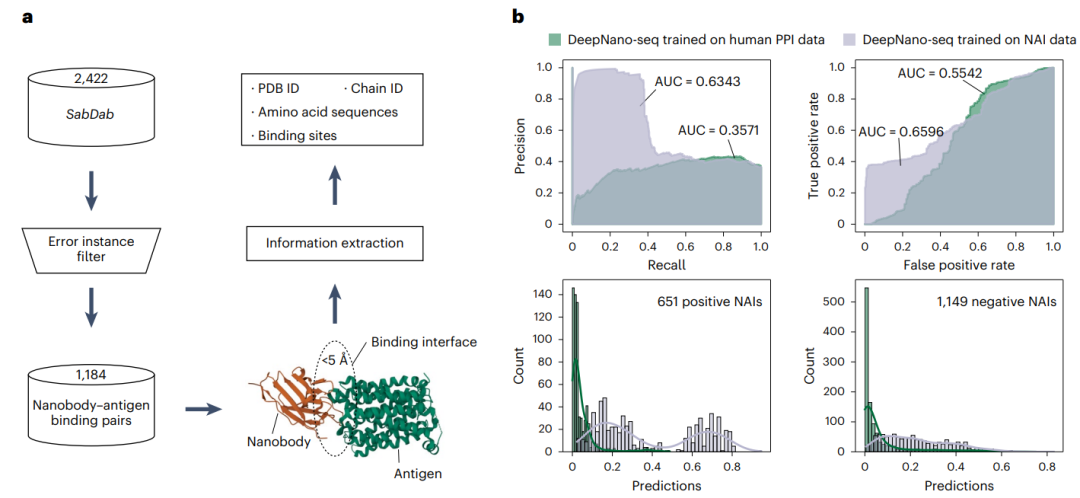
图3: 专用纳米抗体-抗原相互作用nanobody–antigen interaction,NAI预测模型的构建。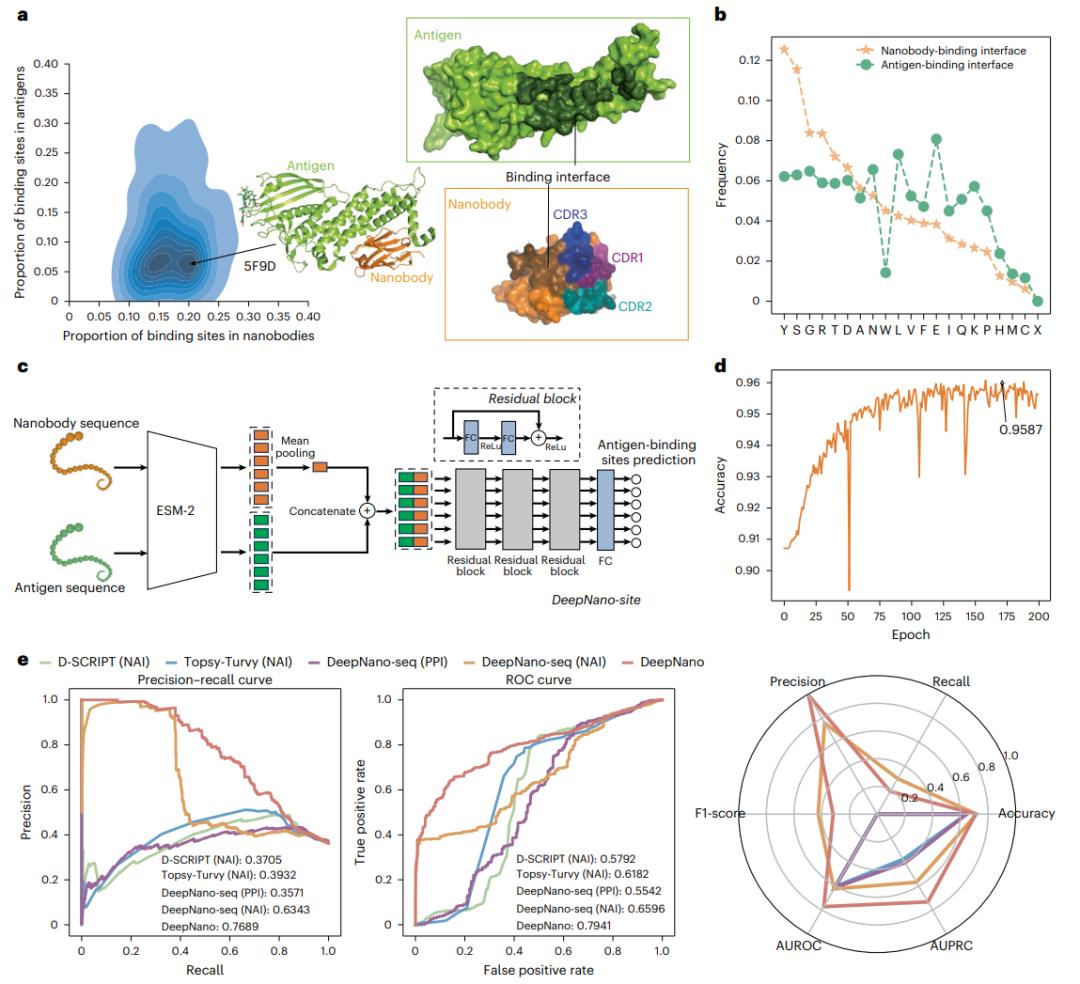
图4: 纳米抗-抗原结合界面的分析,以及DeepNano-site和DeepNano相关实验。
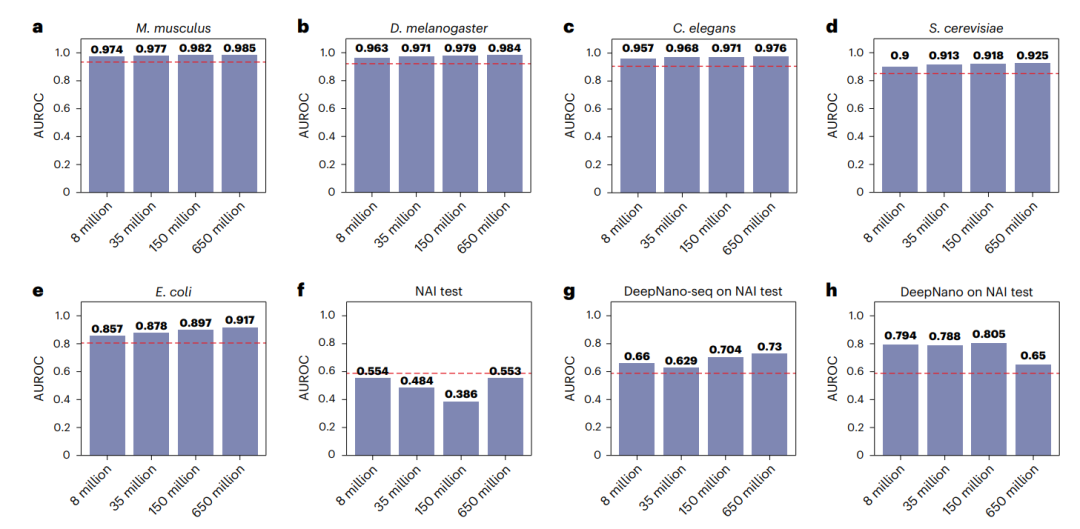
图6: 在四种ESM-2模型尺度下,DeepNano-site和DeepNano预测性能。文献链接
Deng, J., Gu, M., Zhang, P. et al. Nanobody–antigen interaction prediction with ensemble deep learning and prompt-based protein language models. Nat Mach Intell (2024).
https://doi.org/10.1038/s42256-024-00940-5
https://www.nature.com/articles/s42256-024-00940-5
本文译自Nature。
来源:今日新材料
声明:仅代表译者观点,如有不科学之处,请在下方留言指正!