
各位宝子们大家好呀!充满创新思路与科研方向的馆长又来啦,馆长今天来给大家分享一篇发表在《Journal of Translation Medicine》上的纯生信文章!小伙伴们可能都或多或少了解过这个期刊,这个期刊接收纯生信文章,且以积极开放的态度。而且这个期刊的纯生信文章思路极其新颖,对想发纯生信的小伙伴来说,绝对是优秀的学习平台!此外,审稿速度相对较快,平均2.33月,被同学们称为“毕业神刊”!所以馆长今天分享一篇线粒体相关基因的纯生信文章,大家一起来看一看这篇文章的新颖之处!
1.本研究选择了热度很高的线粒体相关基因,是国自然项目的一大热门方向呦!有热度有新颖性,选它就对了!
2.本研究通过多组学数据+机器学习+免疫微环境+全基因组关联研究等,深入分析了肺腺癌预后和个性化治疗的线粒体基因特征。研究思路简直无敌了,新颖性直接拉满!选题热+思路强,具备这两点,纯生信依然可以轻松发文!(ps:听完馆长的介绍,小伙伴们是不是瞬间信心倍增?线粒体相关基因+多组学数据+机器学习就是杠杠滴厉害! 如果你有思路,不知怎么复现,赶紧联系馆长吧!

题目:全面的多组学整合揭示了肺腺癌预后和个性化治疗的线粒体基因特征
杂志:Journal of Translational Medicine
影响因子:IF=6.1
发表时间:2024年10月
研究背景
肺腺癌(LUAD)是原发性肺癌中最常见的组织学亚型,其治疗效果仍然不足,在准确的预后评估方面仍有重大挑战。本研究旨在通过综合多组学方法阐明线粒体相关基因在LUAD中的预后意义,本研究不仅为改善LUAD的预后评估提供了一种新的方法,而且为个性化治疗干预的发展奠定了坚实的基础。
研究思路
本研究利用转录组学和单细胞RNA测序数据,以及临床数据,对LUAD单细胞数据进行降维和聚类。随后通过TCGA-LUAD数据鉴定线粒体相关预后基因,并通过共识聚类将LUAD病例分层为不同的分子亚型。利用机器学习算法,开发了基于线粒体相关基因的人工智能衍生预后模型(AIDPS),并在多个独立数据集中验证了其预后准确性。最后,本研究对免疫微环境和全基因组关联研究数据进行了深入分析,为线粒体相关基因在LUAD发病中的机制研究提供了额外的见解。
主要结果
1.LUAD单细胞表达图谱
本研究对单细胞数据进行降维和聚类分析,确定了31个不同的细胞簇(图1A)。t-SNE图显示了来自正常和肿瘤组织的细胞之间的差异分布(图1B)。随后,本研究分析了这些集群中31个标记基因的表达模式(图1C)。根据这些标记基因的表达谱,本研究将这些细胞簇标注为八种不同的细胞类型:成纤维细胞、内皮细胞、上皮细胞、T细胞、B细胞、骨髓细胞、肥大细胞和NK细胞(图1D)。气泡图通过显示标记基因在这八种细胞类型中的表达谱,有效地验证了这种分类的准确性(图1E)。正常和肿瘤样本的细胞类型组成存在显著差异,肿瘤组织中T细胞和B细胞的比例较高(图1F-G)。(ps:单细胞测序这么大的工作量一定得用服务器,要不然跑得太慢了,想试用服务器联系馆长吧!)
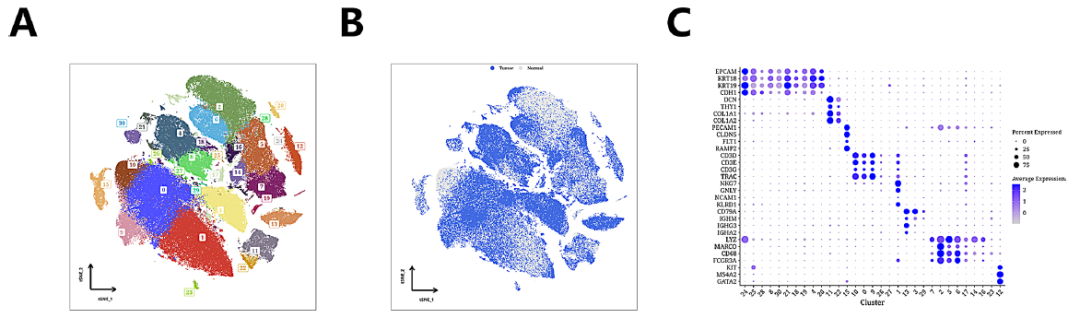
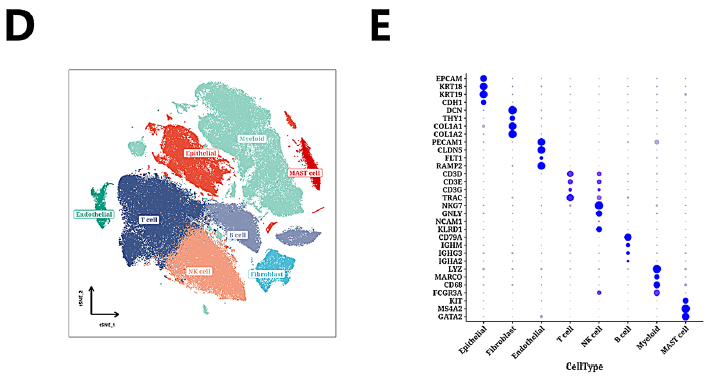
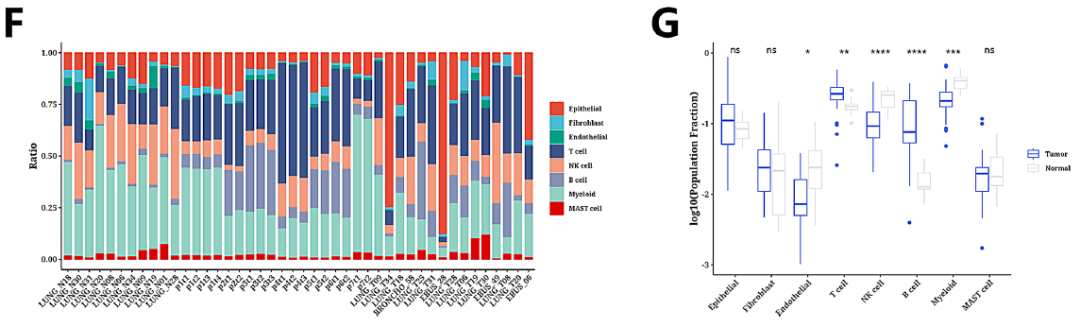
图1 LUAD单细胞分类结果
2. 构建和验证PRGs
本研究分离上皮细胞进行独立降维和聚类分析。tSNE图显示了24个上皮细胞簇及其样本来源(图2A-B)。本研究发现簇7、8和12主要由正常上皮细胞组成,而其余簇则富含肿瘤细胞(图2C)。本研究计算每个集群的差异表达基因(DEGs),并将其用作标记(图2D)。在热图中,聚类16、21、20、10、18和22聚在一起,统一为S2;聚类1、13和23被归为S3;其余的集群被指定为S1,从而产生三个子组(图2E)。亚组轨迹图显示S1包含大部分上皮细胞,S2主要由肿瘤细胞组成,S3具有相对较高比例的正常细胞(图2F)。肿瘤和正常样本之间的比较显示,肿瘤衍生的轨迹包含更多的上皮细胞,分布在所有六种状态(图2G)。此外,本研究选择了100个在拟时间内表现出表达变化的基因(图2H)。转录因子分析发现S1中RXRB、TCF7L1、RARB、ZNF197和SP4富集;S2中FOXJ2、CTCF、KLF3、NFKB2、TCF7L2富集;S3中的NFIC、SNAI1、HNF4G、HFYA、ZEB2富集(图2I)。
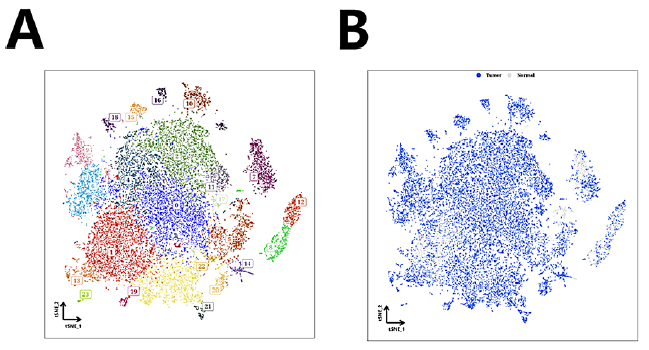
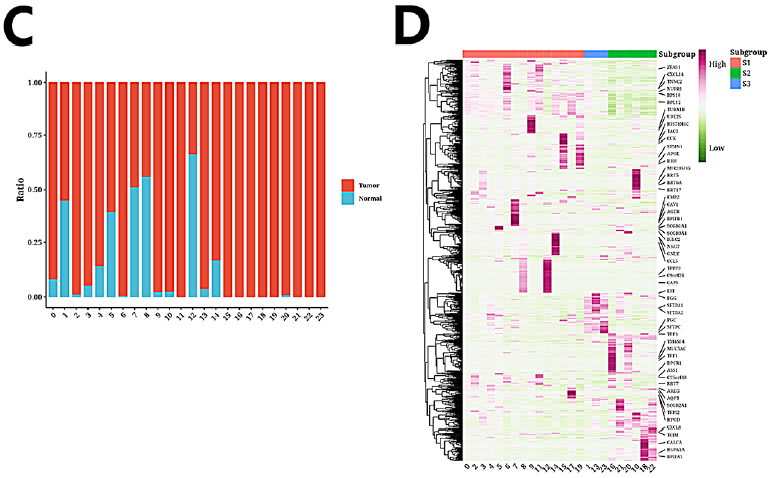
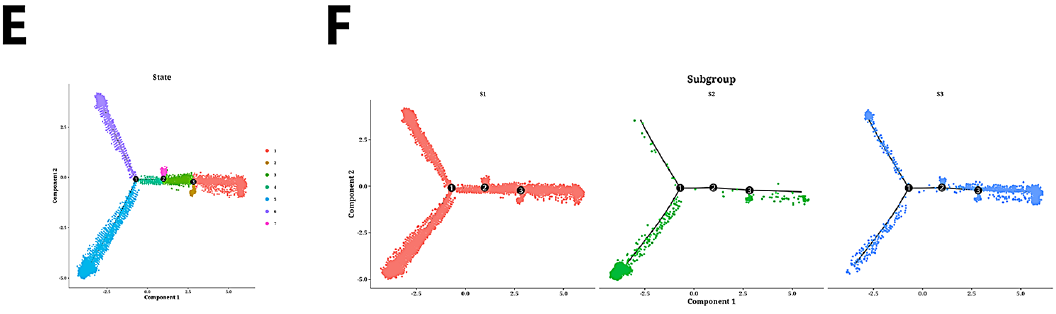
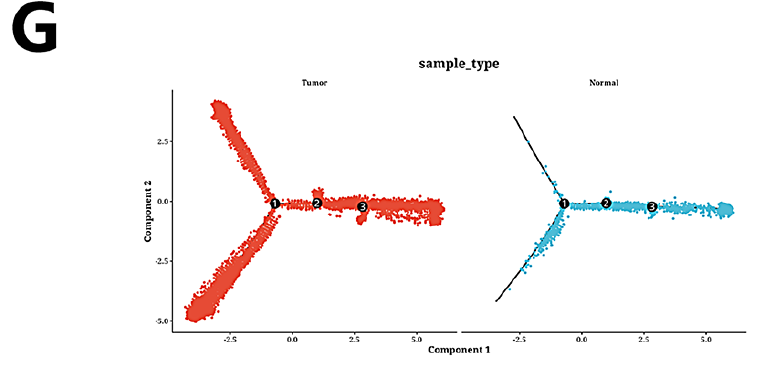
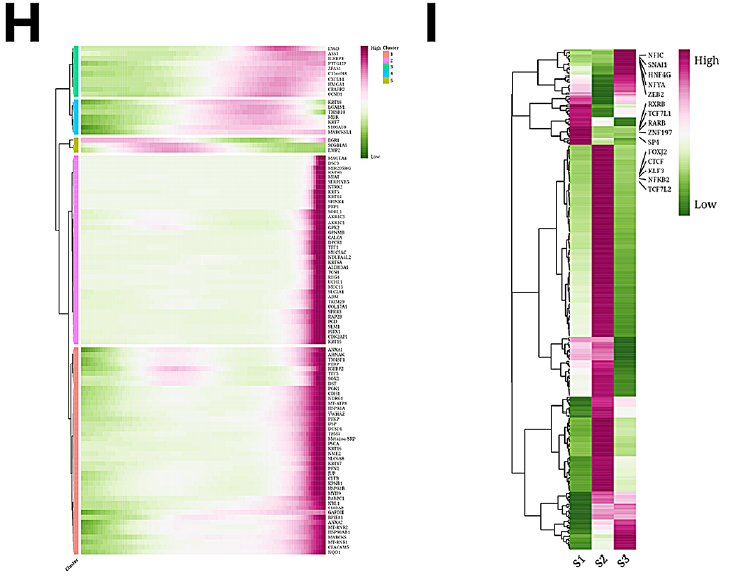
图2上皮细胞亚分类及细胞轨迹分析
3. 基于机器学习集成方法构建的预后风险模型
本研究对2030个线粒体相关基因进行了单因素Cox回归分析,鉴定出220个具有显著预后价值的基因。在TCGA-LUAD数据集中,使用LOOCV框架拟合了101个预测模型,计算了每个模型在11个验证数据集中的c指数,最优模型Enet[a = 0.2]具有最高的平均c指数(0.655)(图3A)。利用最优模型,根据预后基因的表达水平计算每位患者的风险评分,将样本分为高风险组和低风险组。在TCGA-LUAD数据集和其余11个验证数据集中,高危组患者的总生存率明显低于低危组(图3B-M)。本研究在合并所有样本的荟萃队列中观察到类似的趋势(图3N)。
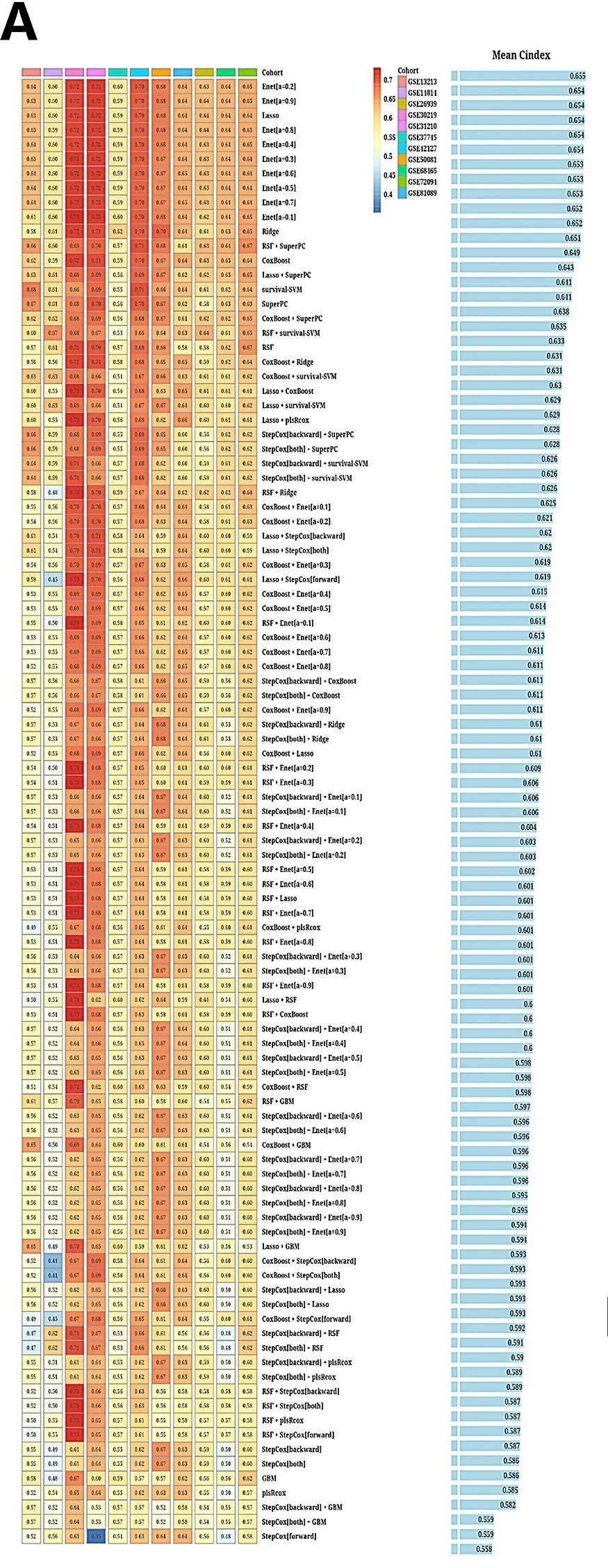
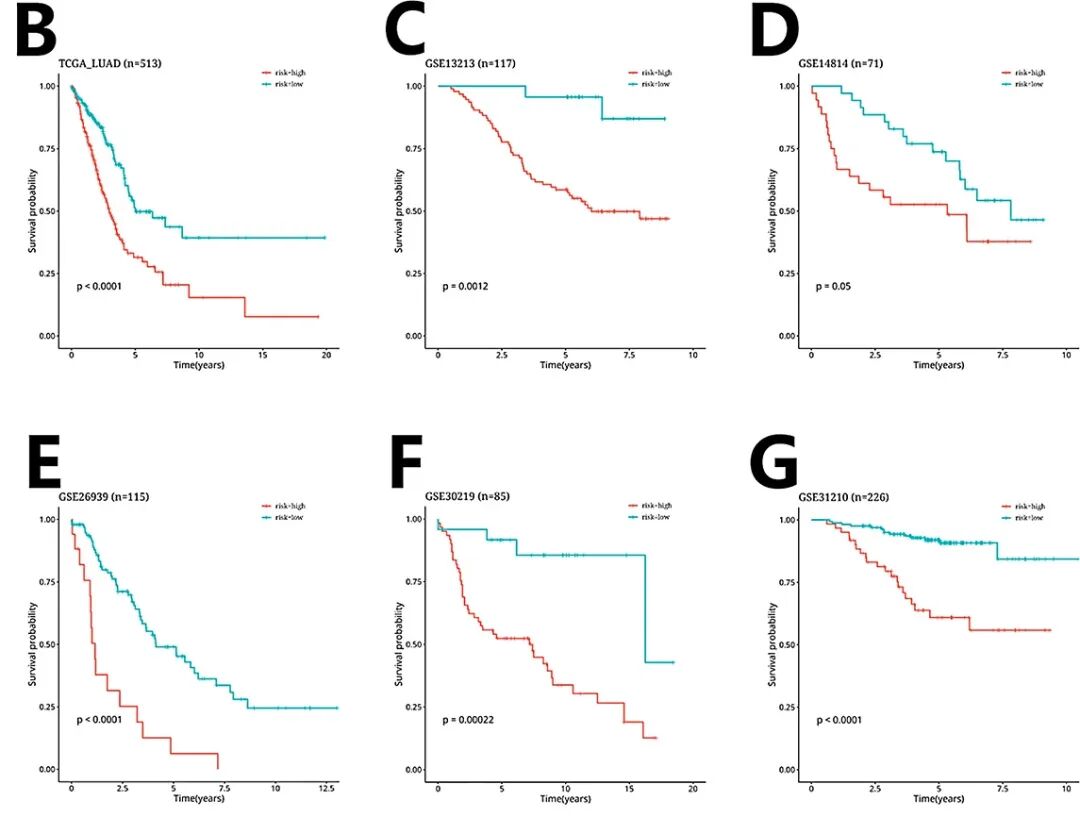
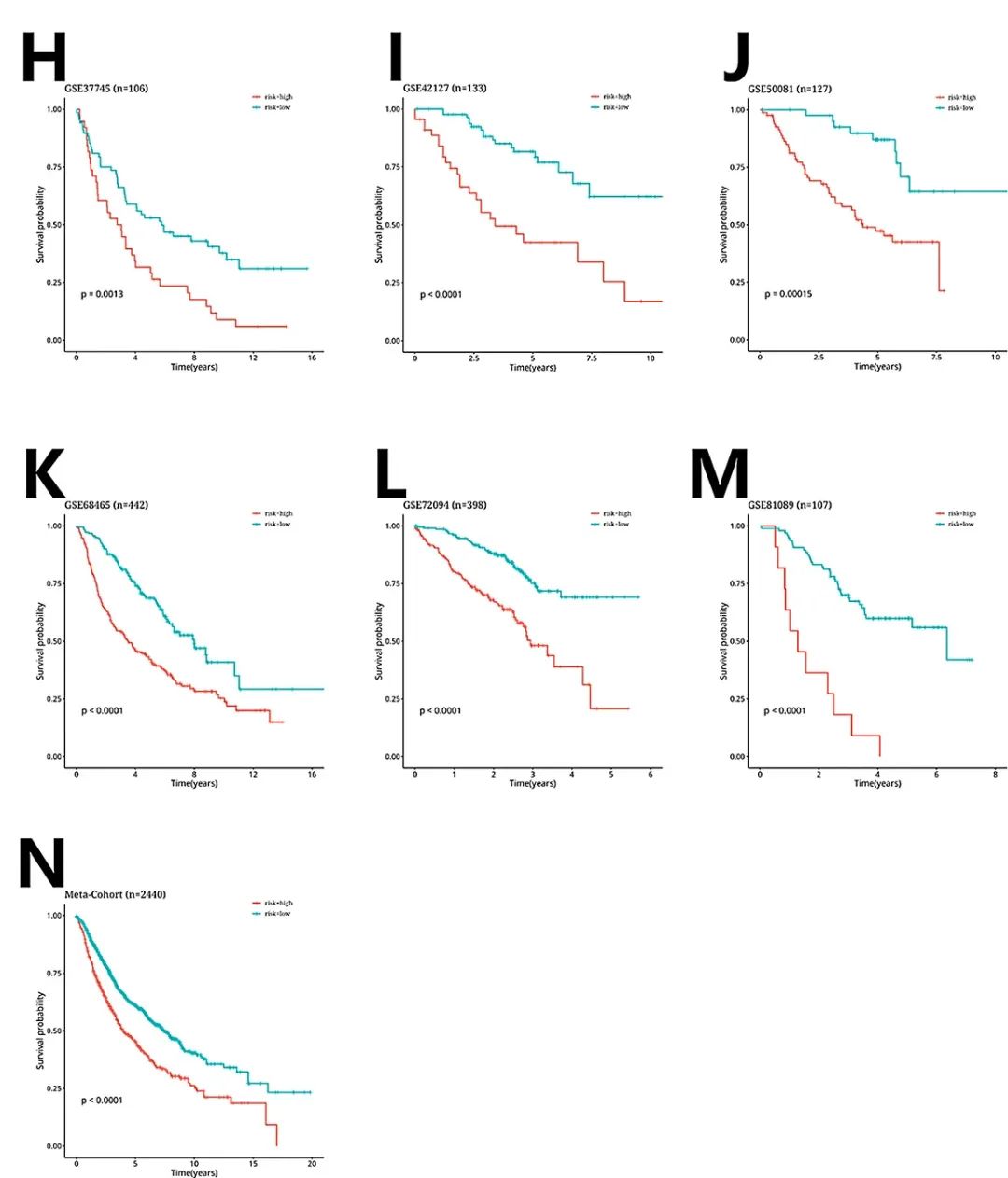
图3 基于机器学习的集成方法生成的风险模型构建结果
4. 高危组和低危组化疗和免疫治疗预测反应分析
本研究观察到化疗药物Bortezomib_1191, Docetaxel_1007, Sepantronium broide_1941, Vinblastine_1004的半最大抑制浓度(IC50)值在高危组和低危组之间存在显著差异(图4A-D)。此外,本研究使用TIDE算法进行分析显示,高危组的TIDE评分明显高于低危组(图4E)。生存分析显示,高危无应答组的预后明显差于高危应答组、低危无应答组和低危应答组(图4F)。条形图显示高危组和低危组中反应者和无反应者的比例,其中71.5%为无反应者,28.5%为反应者(图4G)。生存分析显示,高危组预后明显差于低危组(图4H)。IMvigor210数据集中反应者和无反应者比例的条形图显示,高风险组和低风险组之间的免疫反应组成差异极小(图4I)。最后,对风险评分与免疫检查点基因表达的相关热图分析显示,风险评分与CD276和PVR的表达呈正相关(图4J)。
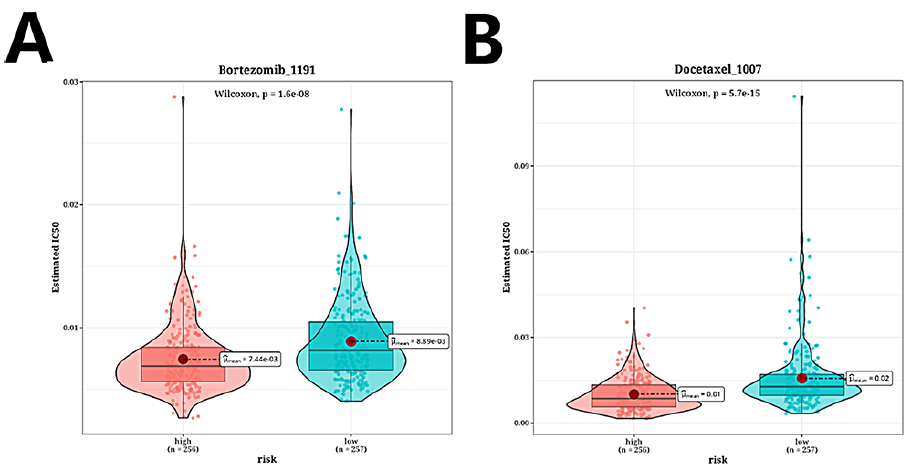
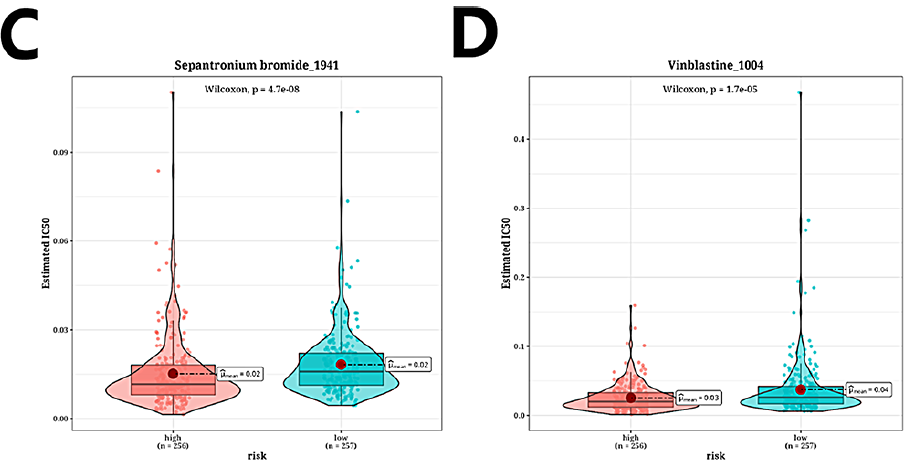
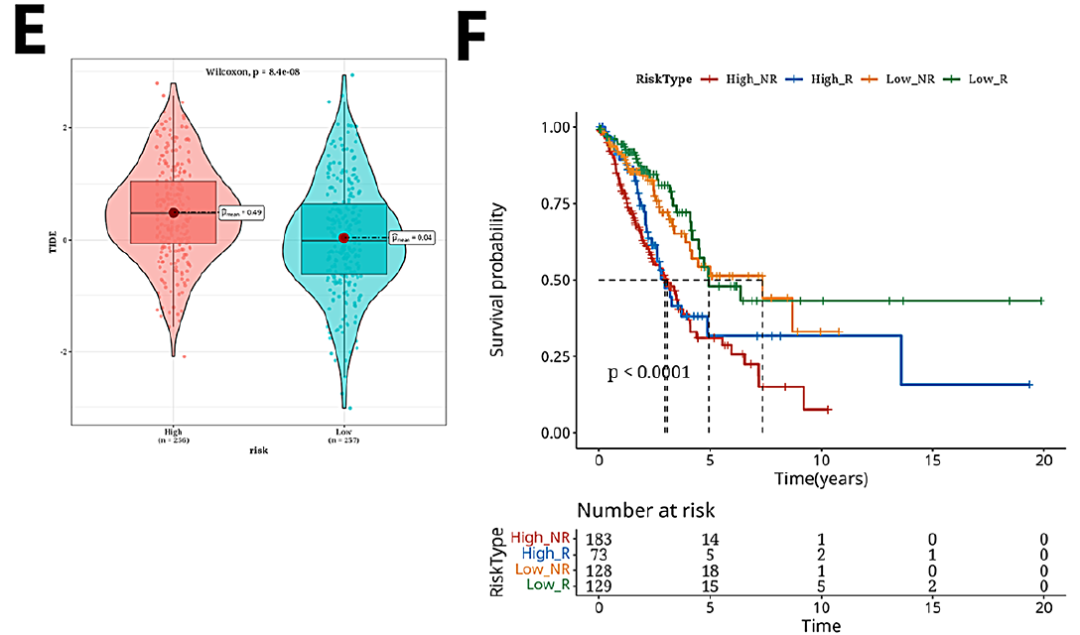
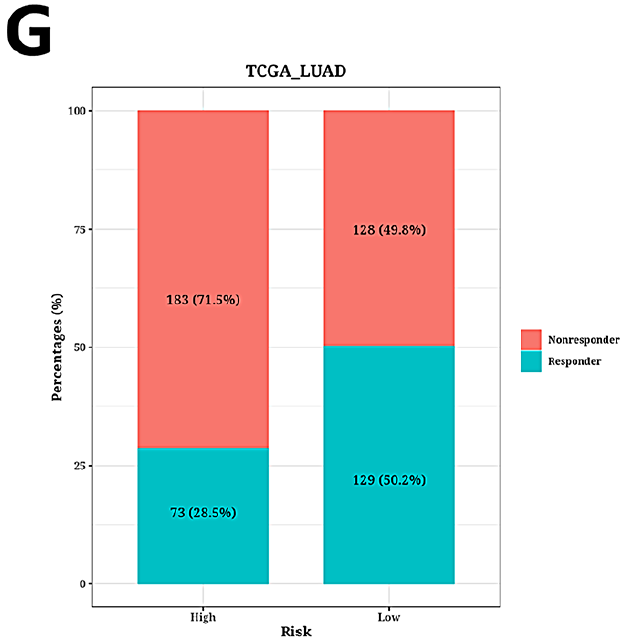
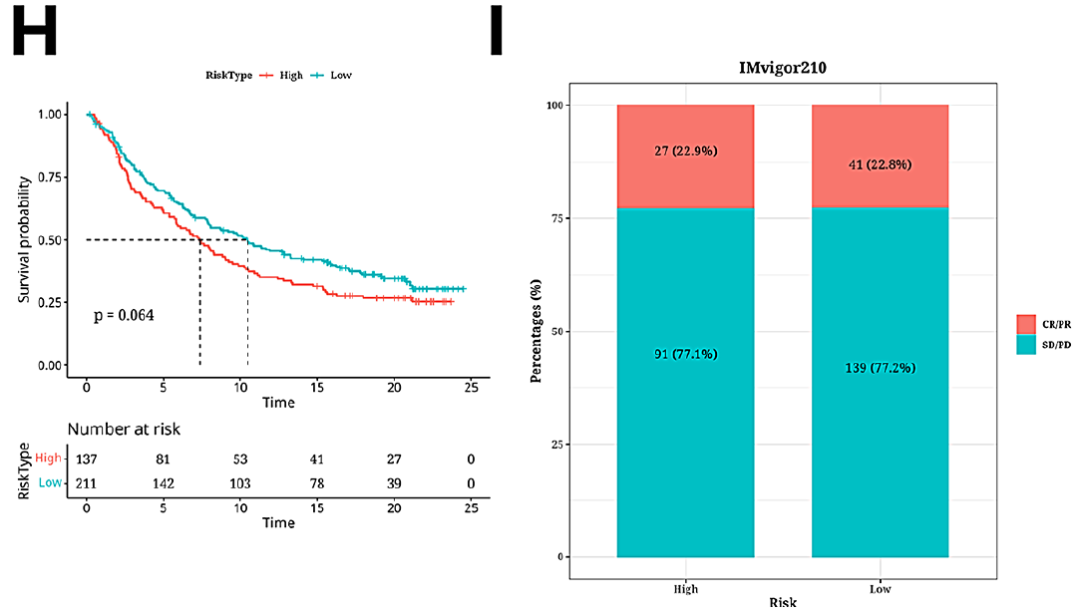

图4 高危人群免疫治疗/化疗反应预测分析结果
5. LC的GWAS数据分析和MR分析
肺癌GWAS数据的曼哈顿图显示,22条染色体上有多个显著的SNP位点,其中最突出的位点位于2号染色体上(图5A)。然后,本研究通过SMR软件使用eQTLGen和LC-GWAS数据进行基因共定位分析,确定了两个预后相关基因CDKN3和MYO1E(图5B-C)。本研究对Cox基因相关SNP位点的孟德尔随机化分析未发现间质性肺病(ebi-a-GCST90018643)和肺癌(ukb-a-54)之间存在显著关联。然而,rs1794002和rs244320等SNP位点与这两种情况都有显著的相关性(图5D-F)。
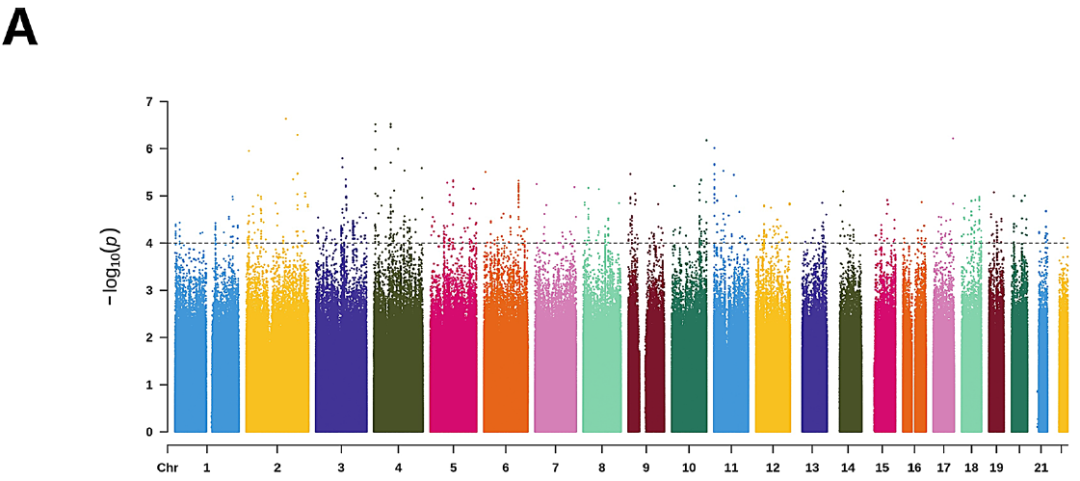
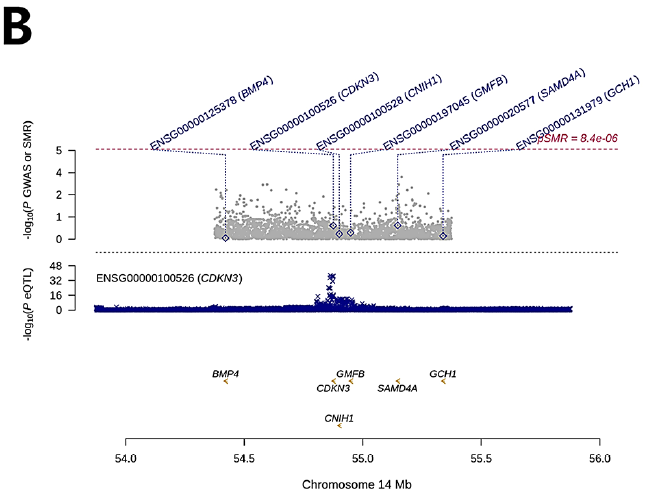
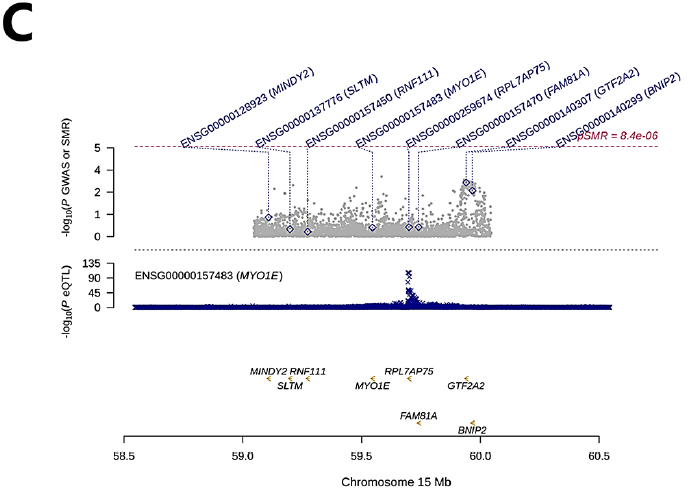
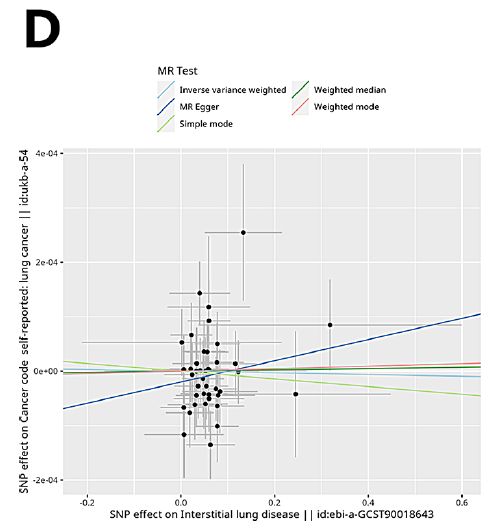
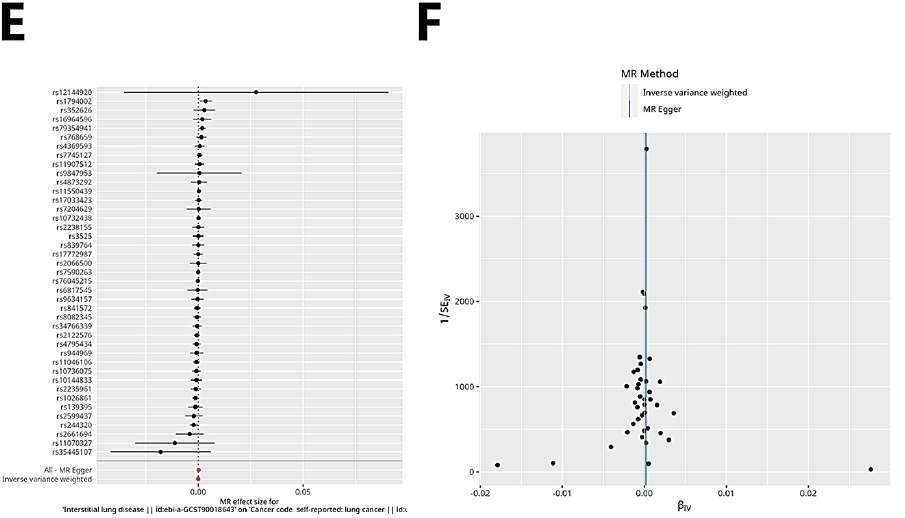
图5 遗传关联和MR分析结果
文章小结
看完这篇文章,有没有觉得文章思路如此新颖?本研究以线粒体相关基因为主要研究方向,妥妥滴紧跟国自然热点!其次,本研究通过多组学分析,揭示了肺腺癌预后和个性化治疗的线粒体基因特征,大量数据集的使用,是文章质量的关键!最后,文章通过机器学习+免疫微环境+全基因组关联研究等进行了深入分析,为线粒体相关基因在LUAD发病中的机制作用提供了额外的见解,给文章整体质量又进行了升华,文章直接纯生信拿下6分+!想发文章的小伙伴们,赶快抓住线粒体相关基因+多组学+机器学习的套路,赶紧大显身手! 关注馆长,无论是生信分析还是基础实验,各种创新性思路设计,馆长都可以帮你解忧哦~
馆长会持续为大家带来最新生信思路,也可以提供特色数据库构建、免费思路评估、付费生信分析等服务,对数据库构建和生信分析感兴趣的朋友可以咨询馆长哦!