特征重要性分析用于了解每个特征(变量或输入)对于做出预测的有用性或价值。目标是确定对模型输出影响最大的最重要的特征,它是机器学习中经常使用的一种方法。
为什么特征重要性分析很重要?
如果有一个包含数十个甚至数百个特征的数据集,每个特征都可能对你的机器学习模型的性能有所贡献。但是并不是所有的特征都是一样的。有些可能是冗余的或不相关的,这会增加建模的复杂性并可能导致过拟合。特征重要性分析可以识别并关注最具信息量的特征,从而带来以下几个优势:下面我们深入了解在Python中的一些特性重要性分析的方法。特征重要性分析方法
1、排列重要性 PermutationImportance该方法会随机排列每个特征的值,然后监控模型性能下降的程度。如果获得了更大的下降意味着特征更重要。from sklearn.datasets import load_breast_cancer from sklearn.ensemble import RandomForestClassifier from sklearn.inspection import permutation_importance from sklearn.model_selection import train_test_split import matplotlib.pyplot as plt
cancer = load_breast_cancer()
X_train, X_test, y_train, y_test = train_test_split(cancer.data, cancer.target, random_state=1)
rf = RandomForestClassifier(n_estimators=100, random_state=1) rf.fit(X_train, y_train)
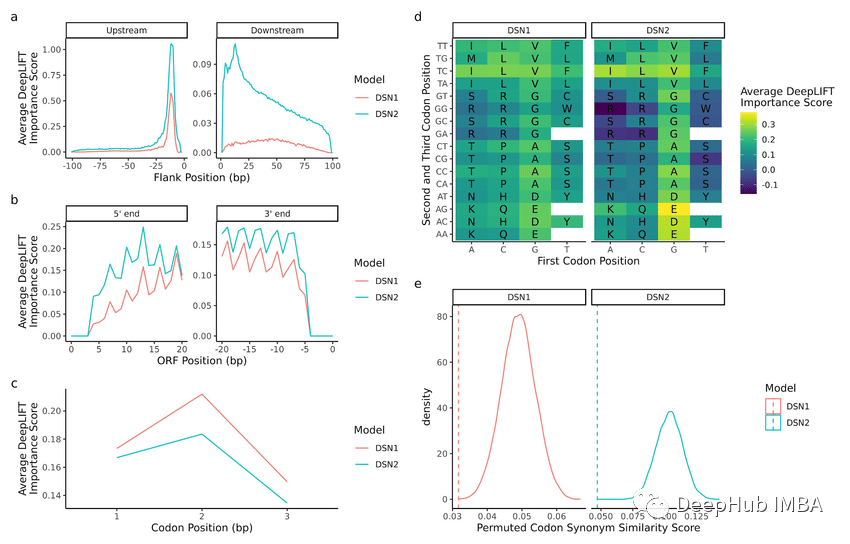
baseline = rf.score(X_test, y_test) result = permutation_importance(rf, X_test, y_test, n_repeats=10, random_state=1, scoring='accuracy')
importances = result.importances_mean
# Visualize permutation importances plt.bar(range(len(importances)), importances) plt.xlabel('Feature Index')
plt.ylabel('Permutation Importance') plt.show()
2、内置特征重要性(coef_或feature_importances_)一些模型,如线性回归和随机森林,可以直接输出特征重要性分数。这些显示了每个特征对最终预测的贡献。 from sklearn.datasets import load_breast_cancer from sklearn.ensemble import RandomForestClassifier
X, y = load_breast_cancer(return_X_y=True)
rf = RandomForestClassifier(n_estimators=100, random_state=1) rf.fit(X, y)
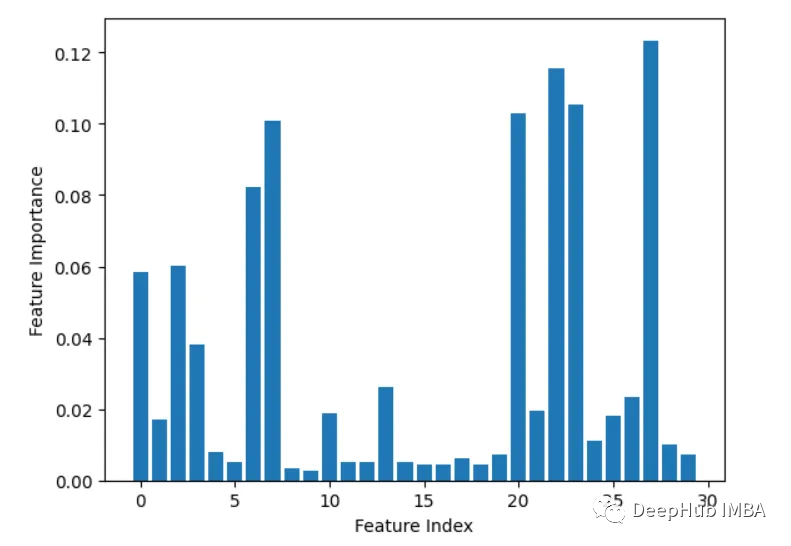
importances = rf.feature_importances_
# Plot importances plt.bar(range(X.shape[1]), importances) plt.xlabel('Feature Index') plt.ylabel('Feature Importance') plt.show()
from sklearn.datasets import load_breast_cancer from sklearn.model_selection import train_test_split from sklearn.ensemble import RandomForestClassifier from sklearn.metrics import accuracy_score import matplotlib.pyplot as plt import numpy as np
# Load sample data X, y = load_breast_cancer(return_X_y=True)
# Split data into train and test sets X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.3, random_state=1)
# Train a random forest model rf = RandomForestClassifier(n_estimators=100, random_state=1) rf.fit(X_train, y_train)
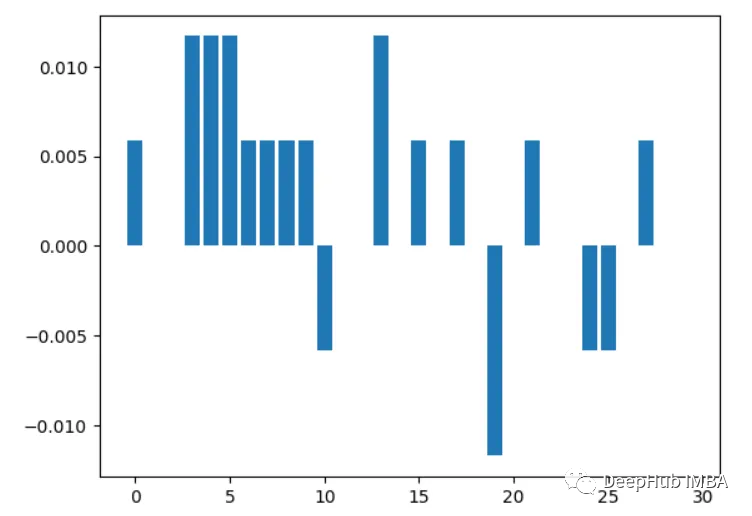
# Get baseline accuracy on test data base_acc = accuracy_score(y_test, rf.predict(X_test))
# Initialize empty list to store importances importances = []
# Iterate over all columns and remove one at a time for i in range(X_train.shape[1]): X_temp = np.delete(X_train, i, axis=1) rf.fit(X_temp, y_train) acc = accuracy_score(y_test, rf.predict(np.delete(X_test, i, axis=1))) importances.append(base_acc - acc)
# Plot importance scores plt.bar(range(len(importances)), importances) plt.show()
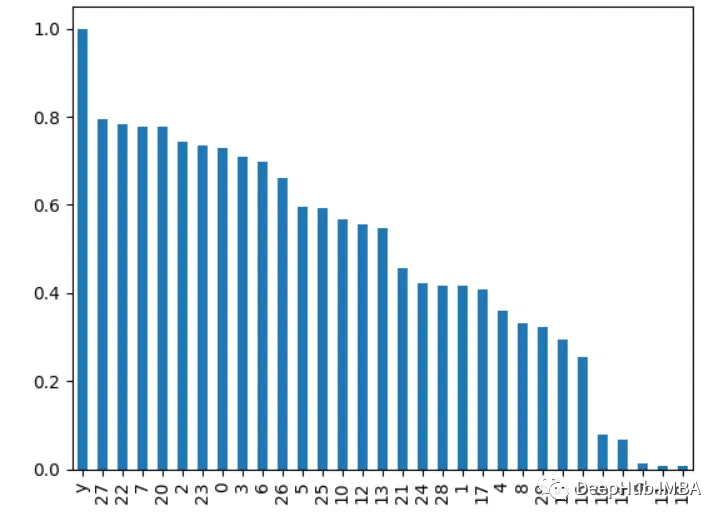
 计算各特征与目标变量之间的相关性。相关性越高的特征越重要。
计算各特征与目标变量之间的相关性。相关性越高的特征越重要。 import pandas as pd from sklearn.datasets import load_breast_cancer
X, y = load_breast_cancer(return_X_y=True) df = pd.DataFrame(X, columns=range(30)) df['y'] = y
correlations = df.corrwith(df.y).abs() correlations.sort_values(ascending=False, inplace=True)
correlations.plot.bar()
5、递归特征消除 Recursive Feature Elimination递归地删除特征并查看它如何影响模型性能。删除时会导致更大下降的特征更重要。 from sklearn.ensemble import RandomForestClassifier from sklearn.feature_selection import RFE import pandas as pd from sklearn.datasets import load_breast_cancer import matplotlib.pyplot as plt
X, y = load_breast_cancer(return_X_y=True) df = pd.DataFrame(X, columns=range(30)) df['y'] = y
rf = RandomForestClassifier()
rfe = RFE(rf, n_features_to_select=10) rfe.fit(X, y)
print(rfe.ranking_)输出为[6 4 11 12 7 11 18 21 8 16 10 3 15 14 19 17 20 13 11 11 12 9 11 5 11]
计算一个特性用于跨所有树拆分数据的次数。更多的分裂意味着更重要。 import xgboost as xgb import pandas as pd from sklearn.datasets import load_breast_cancer import matplotlib.pyplot as plt
X, y = load_breast_cancer(return_X_y=True) df = pd.DataFrame(X, columns=range(30)) df['y'] = y
model = xgb.XGBClassifier() model.fit(X, y)
importances = model.feature_importances_ importances = pd.Series(importances, index=range(X.shape[1])) importances.plot.bar()
对特征进行主成分分析,并查看每个主成分的解释方差比。在前几个组件上具有较高负载的特性更为重要。 from sklearn.decomposition import PCA import pandas as pd from sklearn.datasets import load_breast_cancer import matplotlib.pyplot as plt
X, y = load_breast_cancer(return_X_y=True) df = pd.DataFrame(X, columns=range(30)) df['y'] = y
pca = PCA() pca.fit(X)
plt.bar(range(pca.n_components_), pca.explained_variance_ratio_) plt.xlabel('PCA components') plt.ylabel('Explained Variance')
使用f_classif()获得每个特征的方差分析f值。f值越高,表明特征与目标的相关性越强。 from sklearn.feature_selection import f_classif import pandas as pd from sklearn.datasets import load_breast_cancer import matplotlib.pyplot as plt
X, y = load_breast_cancer(return_X_y=True) df = pd.DataFrame(X, columns=range(30)) df['y'] = y
fval = f_classif(X, y) fval = pd.Series(fval[0], index=range(X.shape[1])) fval.plot.bar()
使用chi2()获得每个特征的卡方统计信息。得分越高的特征越有可能独立于目标。 from sklearn.feature_selection import chi2 import pandas as pd from sklearn.datasets import load_breast_cancer import matplotlib.pyplot as plt
X, y = load_breast_cancer(return_X_y=True) df = pd.DataFrame(X, columns=range(30)) df['y'] = y
chi_scores = chi2(X, y) chi_scores = pd.Series(chi_scores[0], index=range(X.shape[1])) chi_scores.plot.bar()
为什么不同的方法会检测到不同的特征?
不同的特征重要性方法有时可以识别出不同的特征是最重要的,这是因为:像XGBOOST或者回国模型使用内置重要性来进行特征的重要性排列。线性模型倾向于线性关系、树模型倾向于接近根的特征。有的方法可以获取特征之间的相互左右,而有一些则不行,这就会导致结果的差异。使用不同的数据子集,重要性值可能在同一方法的不同运行中有所不同,这是因为数据差异决定的。通过调整超参数,如PCA组件或树深度,也会影响结果。所以不同的假设、偏差、数据处理和方法的可变性意味着它们并不总是在最重要的特征上保持一致。选择特征重要性分析方法的一些最佳实践
- 差异并不一定意味着有问题,检查差异的原因会对数据和模型有更深入的了解。
下载1:OpenCV-Contrib扩展模块中文版教程
在「小白学视觉」公众号后台回复:扩展模块中文教程,即可下载全网第一份OpenCV扩展模块教程中文版,涵盖扩展模块安装、SFM算法、立体视觉、目标跟踪、生物视觉、超分辨率处理等二十多章内容。在「小白学视觉」公众号后台回复:Python视觉实战项目,即可下载包括图像分割、口罩检测、车道线检测、车辆计数、添加眼线、车牌识别、字符识别、情绪检测、文本内容提取、面部识别等31个视觉实战项目,助力快速学校计算机视觉。在「小白学视觉」公众号后台回复:OpenCV实战项目20讲,即可下载含有20个基于OpenCV实现20个实战项目,实现OpenCV学习进阶。交流群
欢迎加入公众号读者群一起和同行交流,目前有SLAM、三维视觉、传感器、自动驾驶、计算摄影、检测、分割、识别、医学影像、GAN、算法竞赛等微信群(以后会逐渐细分),请扫描下面微信号加群,备注:”昵称+学校/公司+研究方向“,例如:”张三 + 上海交大 + 视觉SLAM“。请按照格式备注,否则不予通过。添加成功后会根据研究方向邀请进入相关微信群。请勿在群内发送广告,否则会请出群,谢谢理解~

