今天为大家分享的非肿瘤纯生信文章用了UKB数据库。如果您对临床公共数据库有兴趣,请为小骨点点关注,如果您需要定制化服务,欢迎扫码联系小骨~
文章标题:Data-driven identification of predictive riskbiomarkers for subgroups of osteoarthritis using interpretable machine learning
中文标题:使用可解释的机器学习对骨关节炎亚组的预测性风险生物标志物进行数据驱动识别
发表期刊:Nature Communications
发表时间:2024年4月
影响因子:14.7/Q1
骨关节炎 (OA) 是一种常见的慢性退行性关节疾病,估计有 5.28 亿人患有骨关节炎。从 1990 年到 2019 年,全球 OA 患病率增加了 48%,于人口老龄化和肥胖率的上升,预计还会进一步增加。提高我们对OA发病机制的理解,并制定适当的预测策略以解决未满足的需求至关重要。
研究思路
在这项回顾性研究中,我们开发了一种机器学习模型,用于预测个体风险并识别风险生物标志物,该模型在 OA 诊断前 5 年内出现。通过整合多模式患者数据,我们确定了具有不同风险生物标志物特征的 OA 亚组,经验证,该亚群对英国生物样本库的未见亚群有效,直至诊断前 11 年。该模型利用电子健康记录 (EHR)、临床生物标志物、自我报告的问卷数据、基因组学、蛋白质组学和代谢组学对可用个体子集的 ~20,000 名诊断患有 OA 的人的队列捕获了广泛的风险生物标志物景观。
结果分析
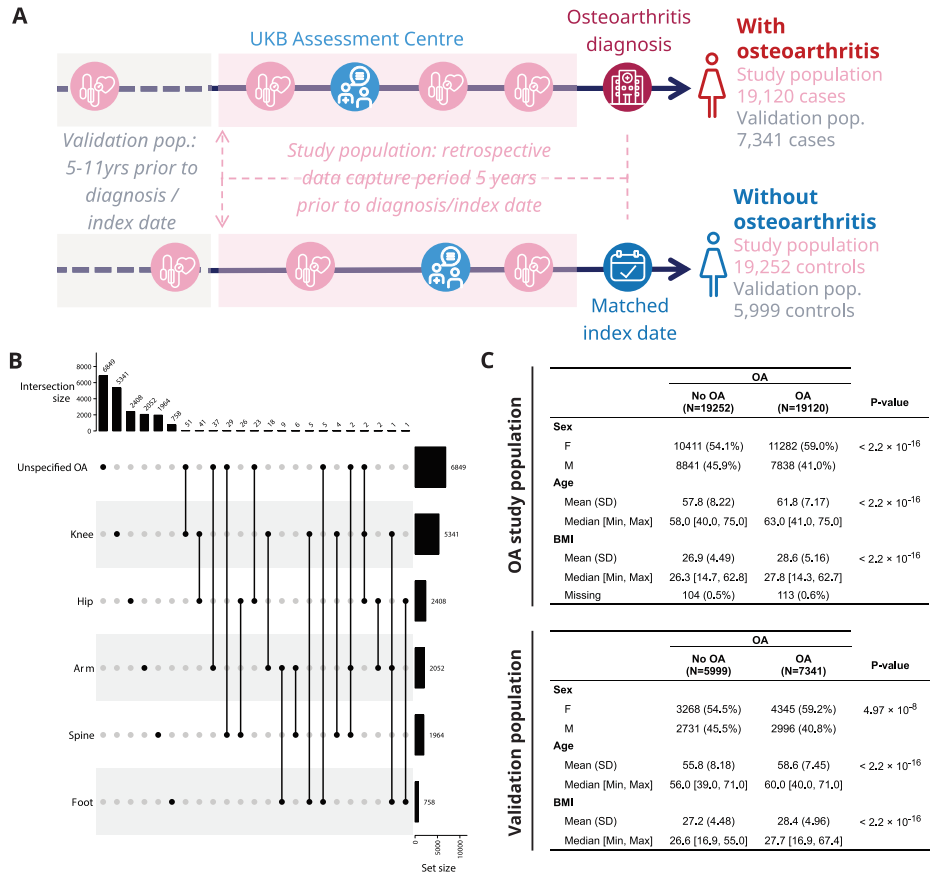
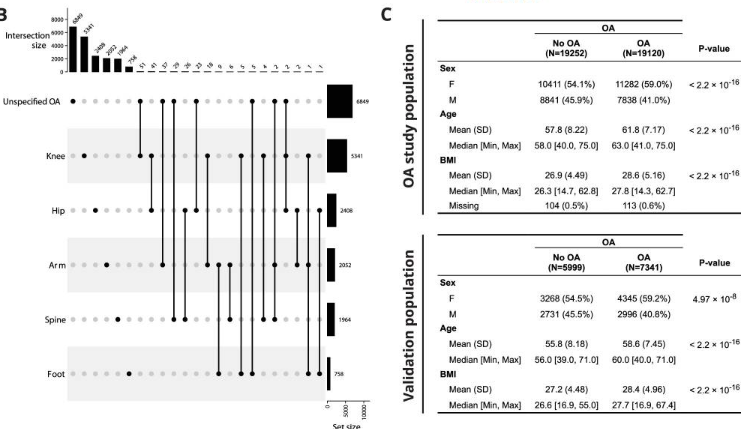
1. OA研究人群
英国生物样本库队列的 ~45% 的初级医疗保健数据可用,这使得被诊断患有 OA 的患者子集 (N = 67,772) 的纵向数据成为可能。确定了在可用的 EHR 研究期间从未被诊断患有 OA 的相同数量的对照参与者 (N = 67,772)。
2. 风险建模
在确定 OA 研究和验证人群后,处理了各种多模态纵向患者数据,以集成到 eXtreme Gradient Boosting (XGBoost) 机器学习模型中。XGBoost 模型经过训练,可以根据回顾性数据预测 OA 诊断的 5 年风险。XGBoost 模型整合了来自招募评估中心的临床、社会人口学、饮食、身体活动和生活方式数据,以及从 OA 诊断或匹配索引日期之前的可用数据中提取的 5 年纵向 EHR 数据的临床数据。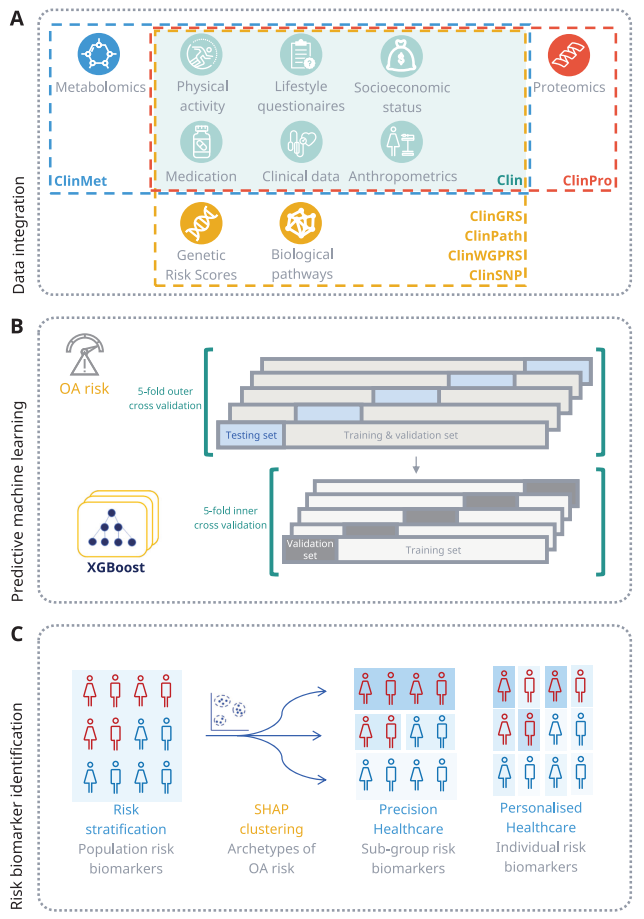
3. 根据 5 年多模式临床数据预测 OA
将回顾性纵向临床数据整合到 XGBoost 模型中,以预测 OA 诊断的 5 年风险(Clin 模型)。在测试集中以 5*5 交叉验证评估性能。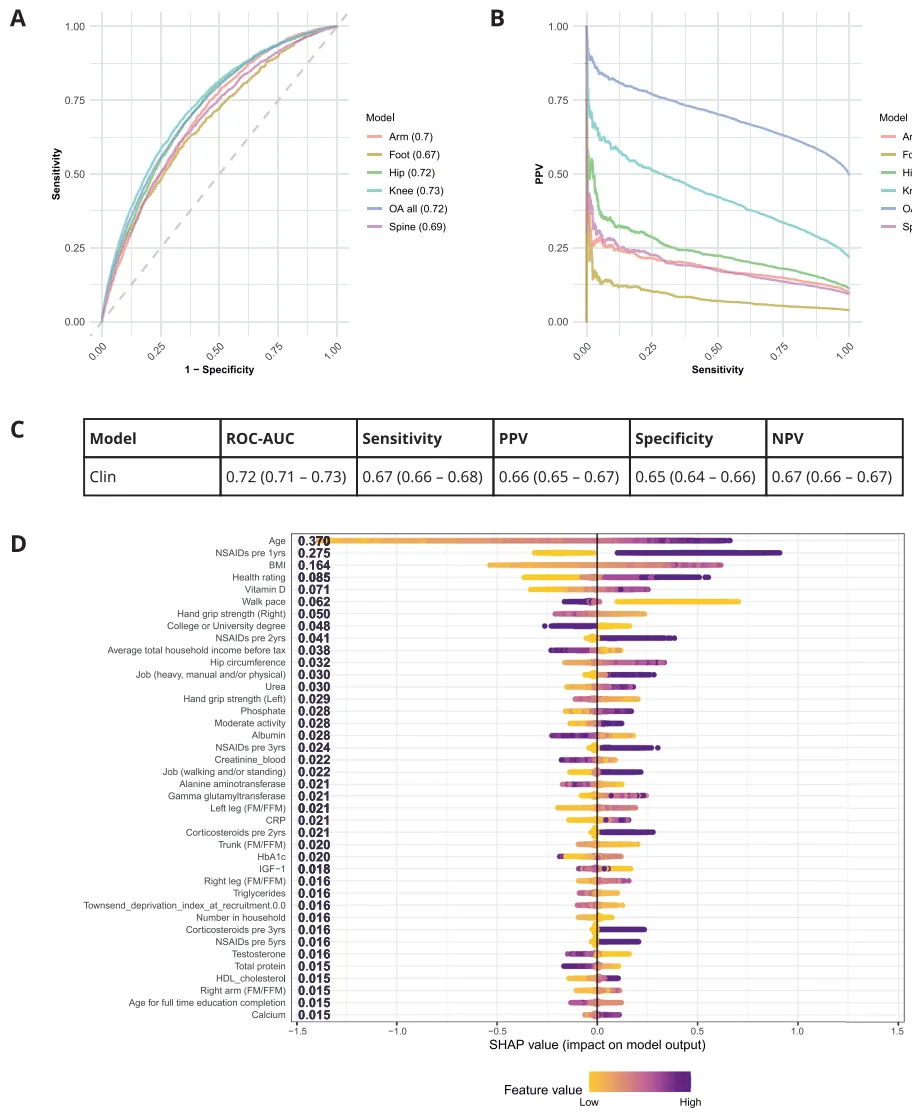
4. OA的精确子组
Clin 模型证实,OA 背后的生物学和环境危险因素在个体之间是异质的。我们试图捕捉这种异质性,并将患者分为具有不同风险生物标志物特征的亚组。因此,我们对所有个体中所有风险生物标志物的 SHAP 值进行了聚类,如 Clin 模型估计的那样。根据轮廓分数和预测指标对聚类分辨率进行了优化,识别出 14 个个体簇。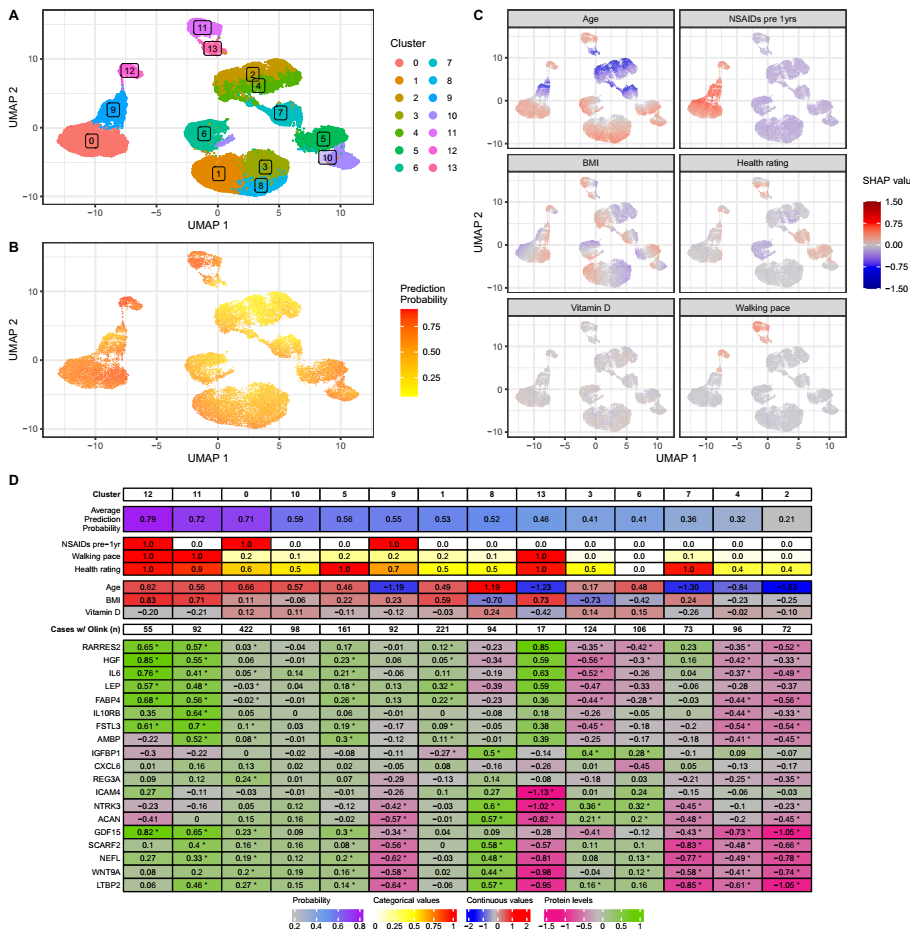
5. 个性化的OA风险生物标志物
使用 Clin 模型的 SHAP 值进行解释,可以量化个体患者数据对其预测的 OA 诊断风险的影响。我们使用瀑布图提取并可视化了个体 OA 风险概况,展示了个人 OA 风险生物标志物预测的积极和负面影响。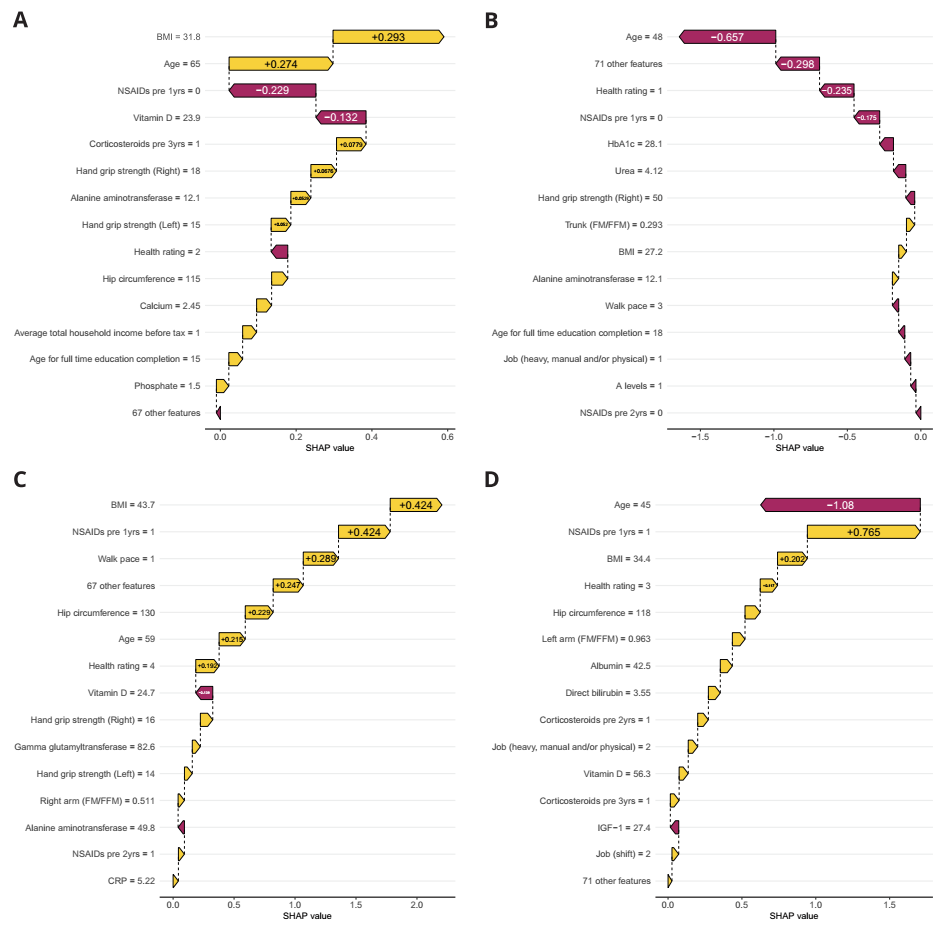
6. 多组学OA风险生物标志物
为了在临床预测模型(Clin模型)的背景下探索OA的分子风险生物标志物,我们将各种类型的组学数据与临床特征相结合,包括OA遗传学(ClinSNP、ClinWGPRS、ClinGRS和ClinPath模型)、代谢组学(ClinMet模型)和蛋白质组学数据(ClinPro模型),用于有这些数据的个体子集。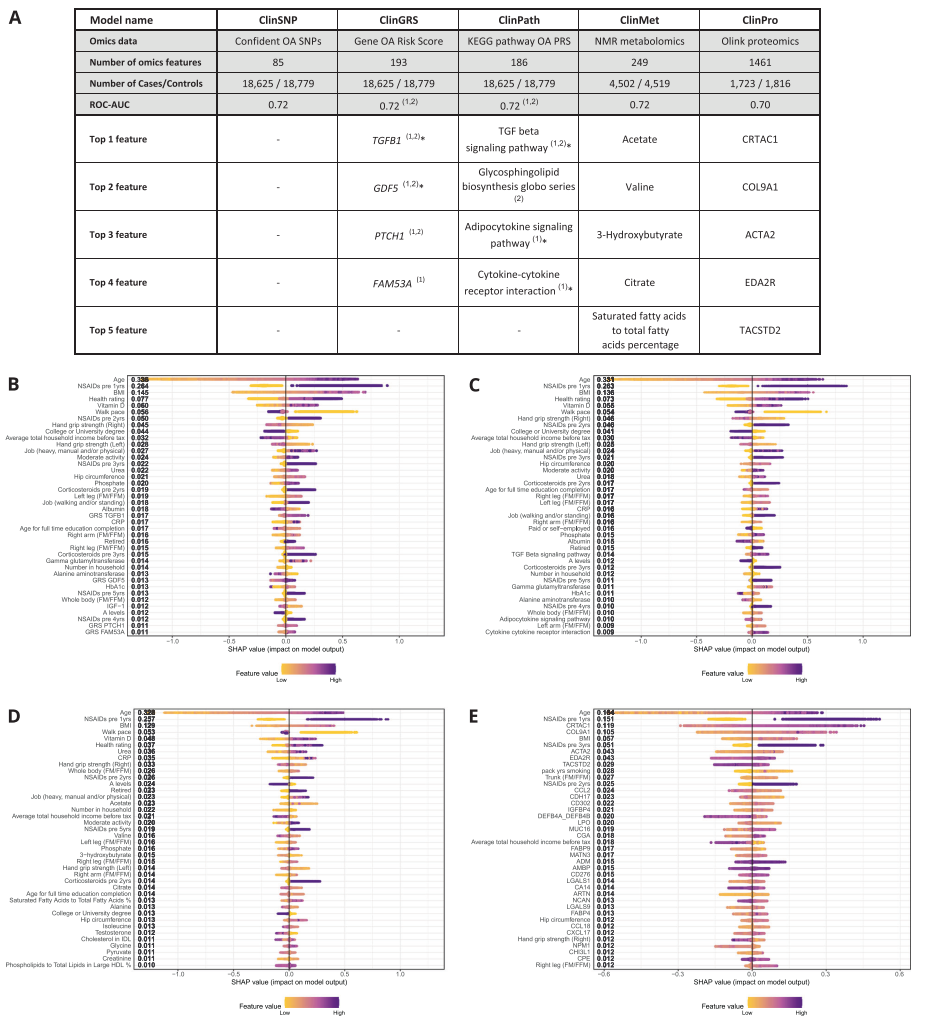
6. 关节间 OA 风险生物标志物异质性
为了进一步探索风险生物标志物在诊断为 OA 的不同关节中的影响,我们对被诊断患有 OA 的个体亚群重新训练了我们的 Clin 模型,这些个体在从临床 OA 诊断中确定的五个关节中的任何一个关节中。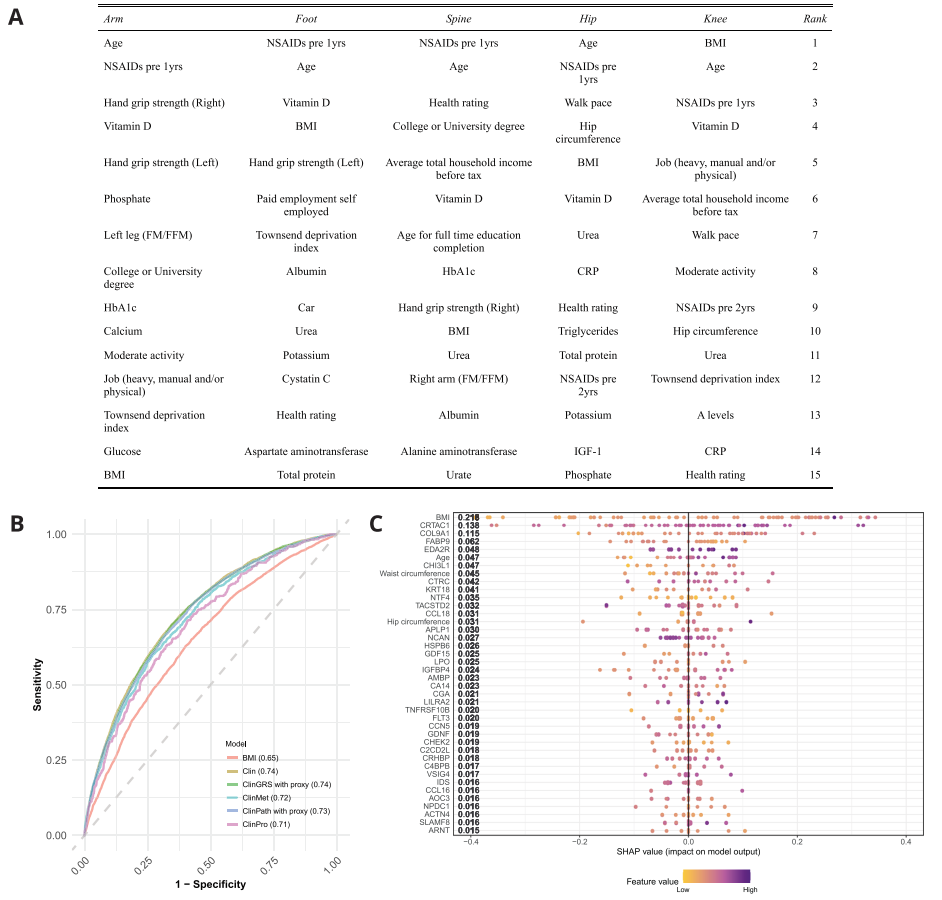
文章小结
该研究基于UKB数据库构建了OA机器学习模型并整合了组学特征,确定了OA特异性风险生物标志物,强调了潜在OA疾病生物学的预测重要性。这些发现可能会促进OA的早期筛查、预防和治疗,减少疾病的发病率和进展。没有思路,找不到创新方向,有任何问题都可以找小骨哦!