
今天我们将分享机器学习10大算法的下半部分,如果还没看过上半部分的可以点击下面图片查看上半部分。
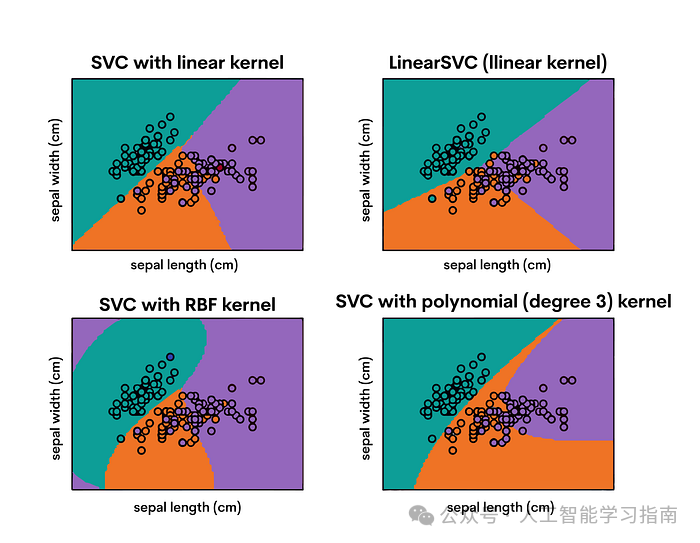
支持向量机(SVM)是强大且多功能的监督学习模型,广泛应用于分类和回归任务中,尤其擅长处理复杂数据集。
SVM 在高维空间中构建超平面(或一组超平面),以分隔不同的类别。
其核心目标是找到最优的边界(即超平面与每个类别中最近点之间的距离,这些点被称为支持向量),以此来区分各个类别。
评估指标
分类任务:准确率、精确率、召回率、F1分数。
回归任务:均方误差(MSE)、R平方。
使用Scikit-learn应用SVM
我们将SVM应用于乳腺癌数据集,专注于将肿瘤分类为良性或恶性。我们将训练SVM模型,并使用分类评估指标来评估其性能。
以下是我们将遵循的步骤:
创建并训练SVM模型:使用默认设置创建一个支持向量机(SVM)模型。SVM以其能够构建超平面(或在更高维空间中构建多个超平面)的能力而著称,这些超平面以尽可能宽的边距分隔不同类别。
预测:使用训练好的SVM模型对测试数据集的类别标签进行预测。模型通过判断每个数据点位于超平面的哪一侧来实现这一点。
评估:将模型的预测结果与测试集的实际标签进行比较,以评估其性能。
代码实现:
from sklearn.svm import SVC
breast_cancer = load_breast_cancer()X, y = breast_cancer.data, breast_cancer.target
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42)
svm_model = SVC()svm_model.fit(X_train, y_train)
y_pred_svm = svm_model.predict(X_test)
accuracy_svm = accuracy_score(y_test, y_pred_svm)precision_svm = precision_score(y_test, y_pred_svm, average='macro')recall_svm = recall_score(y_test, y_pred_svm, average='macro')f1_svm = f1_score(y_test, y_pred_svm, average='macro')
accuracy_svm, precision_svm, recall_svm, f1_svm
print("Accuracy:", accuracy_svm)print("Precision:", precision_svm)print("Recall:", recall_svm)print("F1 Score:", f1_svm)
输出结果:

这些结果表明,SVM模型在乳腺癌数据集上表现极为出色。其高准确率、精确率、召回率和F1分数充分证明了该模型在区分良性和恶性肿瘤方面的有效性。
在医疗诊断中,精确率和召回率之间的平衡尤为重要,因为假阳性和假阴性都可能带来严重的后果。
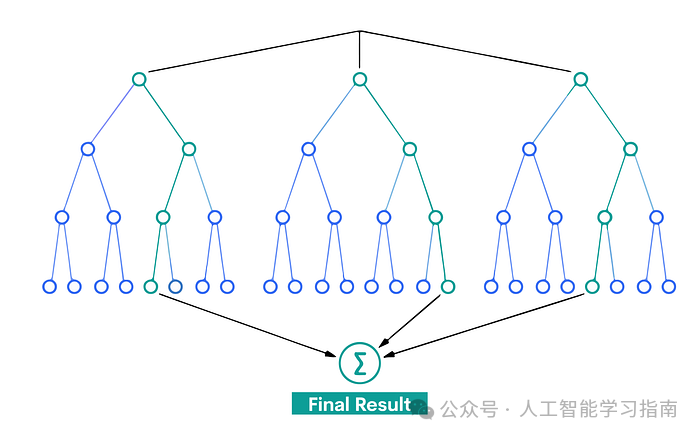
随机森林是一种常用于回归和分类的集成学习技术,为了提供更可靠、更准确的预测,它构建了多棵决策树并将它们的结果进行融合。
在随机森林中,每棵树都会做出一个预测,对于分类任务,模型的预测结果属于获得最多投票的类别;
对于回归任务,则取不同树输出的平均值作为最终结果。
评估指标:
分类:准确率、精确率、召回率、F1分数。
回归:均方误差(MSE)、R平方。
使用Scikit-learn应用随机森林
我们将随机森林应用于乳腺癌数据集,以分类肿瘤为良性或恶性。我们将训练随机森林模型,并使用分类评估指标来评估其性能。
以下是步骤:
代码实现:
from sklearn.ensemble import RandomForestClassifier
breast_cancer = load_breast_cancer()X, y = breast_cancer.data, breast_cancer.target
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42)
rf_model = RandomForestClassifier(random_state=42)rf_model.fit(X_train, y_train)
y_pred_rf = rf_model.predict(X_test)
accuracy_rf = accuracy_score(y_test, y_pred_rf)precision_rf = precision_score(y_test, y_pred_rf, average='macro')recall_rf = recall_score(y_test, y_pred_rf, average='macro')f1_rf = f1_score(y_test, y_pred_rf, average='macro')
print("Accuracy:", accuracy)print("Precision:", precision)print("Recall:", recall)print("F1 Score:", f1)
输出结果:

这些结果表明,随机森林模型在乳腺癌数据集上表现优异,所有关键指标得分均很高。
高精确率和高召回率表明,该模型在准确识别良性和恶性肿瘤方面非常有效,同时能够平衡地减少假阳性和假阴性的发生。
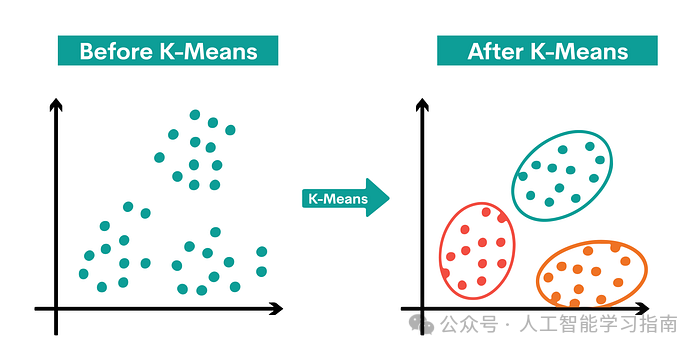
K-均值聚类是一种无监督学习算法,用于将数据分组为“K”个簇。
算法首先确定K个质心,然后将每个数据点分配给最近的簇,目标是最小化质心的大小(即簇内数据点到质心的距离之和)。
算法通过将数据点分配给簇,使得数据点与簇质心之间的平方距离之和达到最小。
簇内数据点的同质性随着簇内方差的减小而增加。
评估指标:
使用Scikit-learn进行应用
我们将使用鸢尾花(Iris)数据集进行K-均值聚类。任务是根据鸢尾花的花卉测量数据将其分组为不同的簇。
我们将训练模型,将植物分配给不同的簇,并评估聚类效果。
代码实现:
from sklearn.datasets import load_irisfrom sklearn.cluster import KMeansfrom sklearn.metrics import silhouette_score
iris = load_iris()X = iris.data
kmeans = KMeans(n_clusters=3, random_state=42)kmeans.fit(X)
y_pred_clusters = kmeans.predict(X)
inertia = kmeans.inertia_silhouette = silhouette_score(X, y_pred_clusters)
print("Inertia:", inertia)print("Silhouette:", silhouette)
输出结果:

这些指标表明,K-均值算法在聚类鸢尾花数据集方面表现相当不错,尽管在簇的紧凑性和分离度方面仍有改进空间。
主成分分析(PCA)是实现降维的一种有效方法。它通过将数据转换到一个新的坐标系中,减少变量的数量,同时尽可能保留原始数据的主要变化信息。
PCA旨在找到能够最大化数据中方差的主要成分或轴。第一个主成分捕获最多的方差,第二个主成分(与第一个正交)捕获次多的方差,依此类推。
评估指标:
解释方差:表示每个主成分捕获了多少数据中的方差。
总解释方差:所选主成分累计解释的方差总和。
使用Scikit-learn进行应用:
我们将对乳腺癌数据集应用PCA,该数据集包含了从乳腺肿瘤细针穿刺(FNA)数字图片中提取的特征。
我们的目标是在最小化数据集维度的同时,保留尽可能多的信息。
以下是我们将遵循的步骤:
应用PCA:我们初始化PCA,设置n_components=2,表示我们的目标是将数据集降至二维。这一选择通常用于可视化目的或作为其他算法的预处理步骤。我们将PCA拟合到数据X上。在此过程中,PCA会识别出对数据中方差贡献最大的轴(即主成分)。
转换数据:使用PCA的transform方法将降维应用到X上。这将生成一个新的数据集X_pca,其中每个数据点现在都用两个主成分来表示。
评估PCA转换效果:我们通过查看每个主成分的解释方差来评估PCA转换的效果。这告诉我们每个主成分捕获了数据总方差中的多少比例。
通过计算两个主成分的解释方差之和,我们得到总解释方差,这为我们提供了一个整体衡量标准,即在降维过程中保留了多少信息。
代码实现:
from sklearn.datasets import load_breast_cancerfrom sklearn.decomposition import PCAimport numpy as np
breast_cancer = load_breast_cancer()X = breast_cancer.data
pca = PCA(n_components=2) pca.fit(X)
X_pca = pca.transform(X)
explained_variance = pca.explained_variance_ratio_
total_explained_variance = np.sum(explained_variance)
print("Explained variance:", explained_variance)print("Total Explained Variance:", total_explained_variance)
输出结果:

现在,我们来评估结果。
解释方差:
第一主成分:98.20%
第二主成分:1.62%
总解释方差:99.82%
这些结果表明,通过仅将数据集降维到两个主成分,我们已经捕获了数据集中约99.82%的总方差。
其中,仅第一主成分就占据了显著的大部分方差,这表明它捕获了数据集中大部分的关键信息。
梯度提升是一种先进的机器学习技术,它通过顺序构建多个弱预测模型(通常是决策树)来实现。
每个新模型都逐渐最小化整个系统的损失函数(误差)。
它涉及三个主要组成部分:
一个加法模型,用于添加弱学习器以最小化损失函数;
一个需要优化的损失函数;
一个需要生成预测的弱学习器。
每一棵新树都会修正前面树所犯的错误。
评估指标:
对于分类问题:准确率、精确率、召回率、F1分数。
对于回归问题:均方误差(MSE)、R平方。
使用Scikit-learn应用:我们将使用糖尿病数据集进行梯度提升。
我们的目标是基于各种特征来预测糖尿病的进展,我们将训练一个梯度提升模型并评估其性能。
以下是我们将遵循的步骤:
加载糖尿病数据集:糖尿病数据集包含了多种特征,包括年龄、性别、身体质量指数、平均血压以及六项血液血清指标。目标变量是在基线后一年内对疾病发展的定量评估。
创建并训练梯度提升模型:我们初始化一个梯度提升回归器。梯度提升允许优化任何可微分的损失函数,并以逐步向前的方式构建加性模型。我们在训练数据上训练(拟合)这个模型。在这一步中,模型学习基于特征来预测糖尿病的进展。
预测:我们使用训练好的梯度提升模型对测试数据中的疾病进展进行预测。这一步涉及将模型应用于未见过的数据,以评估其预测能力。
评估:我们使用两个关键指标来评估模型的性能:
均方误差(MSE):该指标计算错误平方的平均值。它是评估估计量质量的一个指标;值越接近零表示质量越高。
R平方:该统计量基于模型解释的总结果方差的百分比,给出了模型复制观测结果好坏的指示。
代码实现:
from sklearn.datasets import load_diabetesfrom sklearn.ensemble import GradientBoostingRegressorfrom sklearn.metrics import mean_squared_error, r2_score
diabetes = load_diabetes()X, y = diabetes.data, diabetes.target
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42)
gb_model = GradientBoostingRegressor(random_state=42)gb_model.fit(X_train, y_train)
y_pred_gb = gb_model.predict(X_test)
mse_gb = mean_squared_error(y_test, y_pred_gb)r2_gb = r2_score(y_test, y_pred_gb)
print("MSE:", mse_gb)print("R2 score:", r2_gb)
输出结果:

这些结果表明,梯度提升模型在预测糖尿病进展方面具有中等程度的准确性。
R平方值为0.45,表明模型解释了目标变量中近45%的方差,这对于如此复杂的任务来说是一个不错的表现。
MSE为我们提供了观测到的实际结果与模型预测结果之间平均平方差的一个概念。
如果大家想更进一步学习机器学习、深度学习、神经网络技术的可以看看以下文章(文章中提到的资料都打包好了,都可以直接添加小助手获取)
机器学习算法的上半部分结束了,大家觉得这篇文章有帮助的话记得分享给你的朋友、同学、闺蜜、敌蜜、死党!