
今天给同学们分享一篇生信文章“Effects of Lipid Metabolism-Related Genes PTGIS and HRASLS on Phenotype, Prognosis, and Tumor Immunity in Lung Squamous Cell Carcinoma”,这篇文章发表在Oxid Med Cell Longev期刊上,影响因子为7.31。

脂质代谢相关差异表达基因的鉴定和突变景观
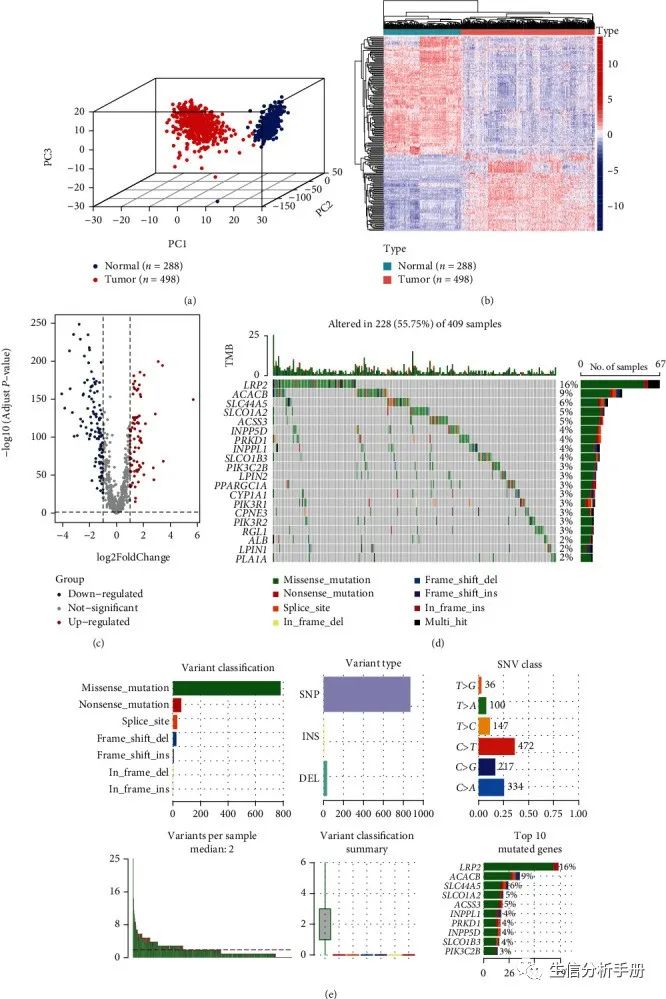
该研究的工作流程图如图1所示。3D PCA图显示TCGA数据集中的498个LUSC样本和GTEx数据集中的288个正常肺组织样本明显独立(图2(a))。通过使用筛选阈值为|log2FC| > 1和p值 < 0.05,鉴定出了191个与脂质代谢相关的差异表达基因(DEGs),其中包括116个下调基因和75个上调基因(图2(b)和2(c))。然后,作者总结了LUSC中191个DEGs的体细胞突变发生率。在409个LUSC患者中,228个(55.75%)发现了遗传突变(图2(d)和2(e))。图2(d)显示了LUSC中具有前20个突变率的DEGs的突变景观。错义突变是最高分类的变异类型。单核苷酸多态性(SNP)是最常见的突变类型,C>T是具有最高发生率的单核苷酸变异(SNV)类型(图2(e))。
差异表达基因的富集分析
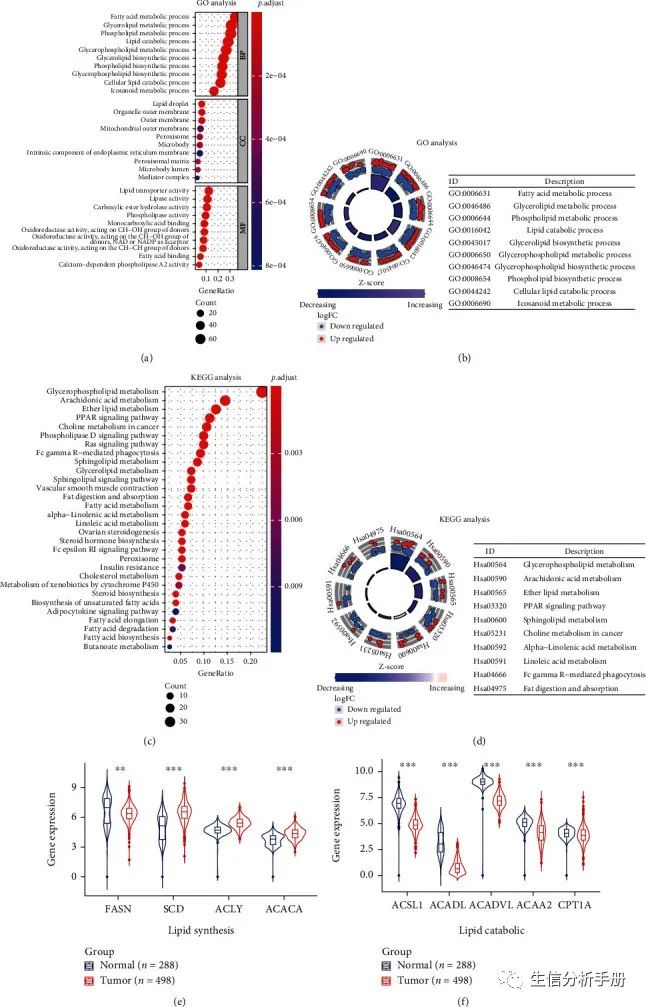
GO分析显示,这191个差异表达基因主要参与了脂肪酸代谢过程、甘油脂代谢过程和磷脂代谢过程(图3(a)和3(b))。KEGG分析富集的通路包括甘油磷脂代谢、花生四烯酸代谢和醚脂代谢(图3(c)和3(d))。此外,与正常肺组织相比,LUSC中的脂质合成(图3(e))、脂质分解(图3(f))和脂质摄取(附图1)的代表性基因表达存在显著差异。这些结果表明了脂质代谢在LUSC中的重要性。
鉴定与TCGA队列中脂质代谢相关的预后基因
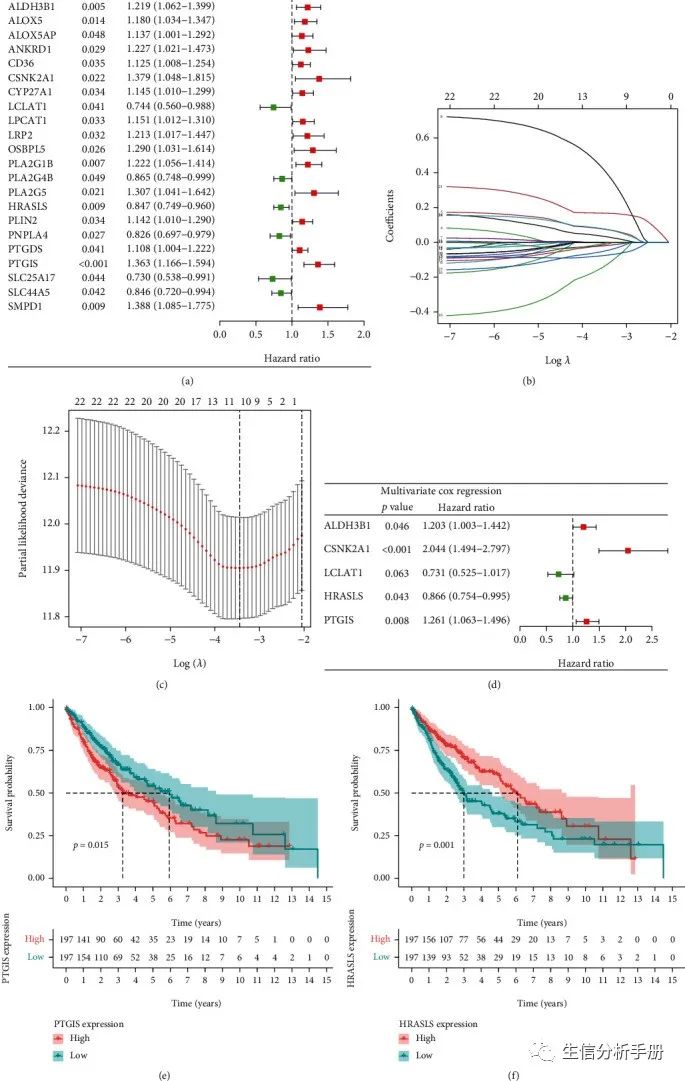
作者使用单变量Cox回归、LASSO回归和多变量Cox回归分析筛选与LUSC预后相关的DEGs。通过单变量Cox回归分析筛选出与LUSC预后相关的24个LMRGs(p值<0.05)(图4(a))。然后,这些基因参与了LASSO回归分析,并确定了具有最佳λ值的10个基因(图4(b)和4(c))。基于LASSO回归分析产生的基因,通过多变量Cox回归分析确定了5个独立的LUSC预后基因(图4(d))。Kaplan-Meier生存分析检查了5个独立预后基因的表达与LUSC的OS之间的关系(图4(e)和4(f)以及附图2),其中PTGIS的高表达(图4(e))和HRASLS的低表达(图4(f))与LUSC的不良预后相关。
在细胞系和组织中验证PTGIS和HRASLS的表达
RT-qPCR显示PTGIS在LUSC细胞系和组织中的表达较低,而HRASLS较高(图5(a)和5(c))。WB分析显示LUSC细胞系(NCI-H226和SK-MES-1)中PTGIS的蛋白水平显著低于肺上皮细胞系(BEAS-2B),而HRASLS显著高(图5(b))。接下来,作者使用免疫组化(IHC)检测LUSC和配对的肿瘤邻近正常肺组织中PTGIS和HRASLS的蛋白表达,发现PTGIS的蛋白水平在LUSC中较低,而HRASLS较高(图5(d))。
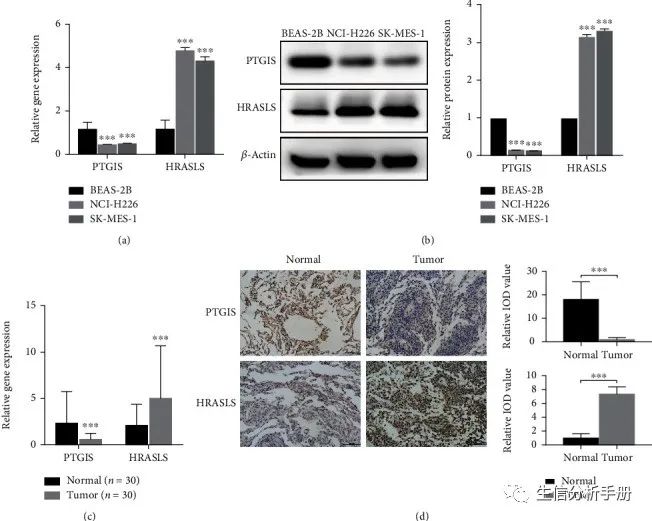
PTGIS和HRASLS对LUSC的体外增殖、迁移和侵袭产生影响
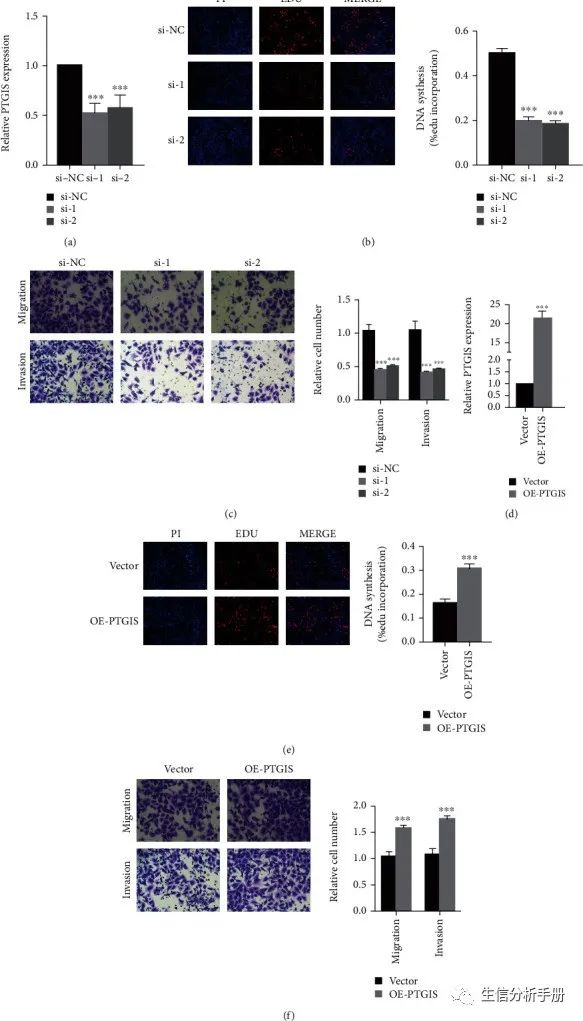
为了阐明PTGIS和HRASLS在LUSC中的作用,作者在LUSC细胞系(SK-MES-1)中进行了细胞实验。使用siRNA使PTGIS和HRASLS减少导致PTGIS(图6(a))和HRASLS(图7(a))水平显著下降。PTGIS敲除显著抑制了LUSC细胞的增殖、迁移和侵袭(图6(b)和6(c)),而HRASLS敲除显著促进了LUSC细胞的增殖、迁移和侵袭(图7(b)和7(c))。PTGIS和HRASLS表达质粒导致PTGIS(图6(d))和HRASLS(图7(d))水平显著过表达。PTGIS过表达促进了LUSC细胞的增殖、迁移和侵袭(图6(e)和6(f)),而HRASLS过表达抑制了LUSC细胞的增殖、迁移和侵袭(图7(e)和7(f))。综上所述,这些结果表明PTGIS和HRASLS在LUSC表型中起着重要作用。

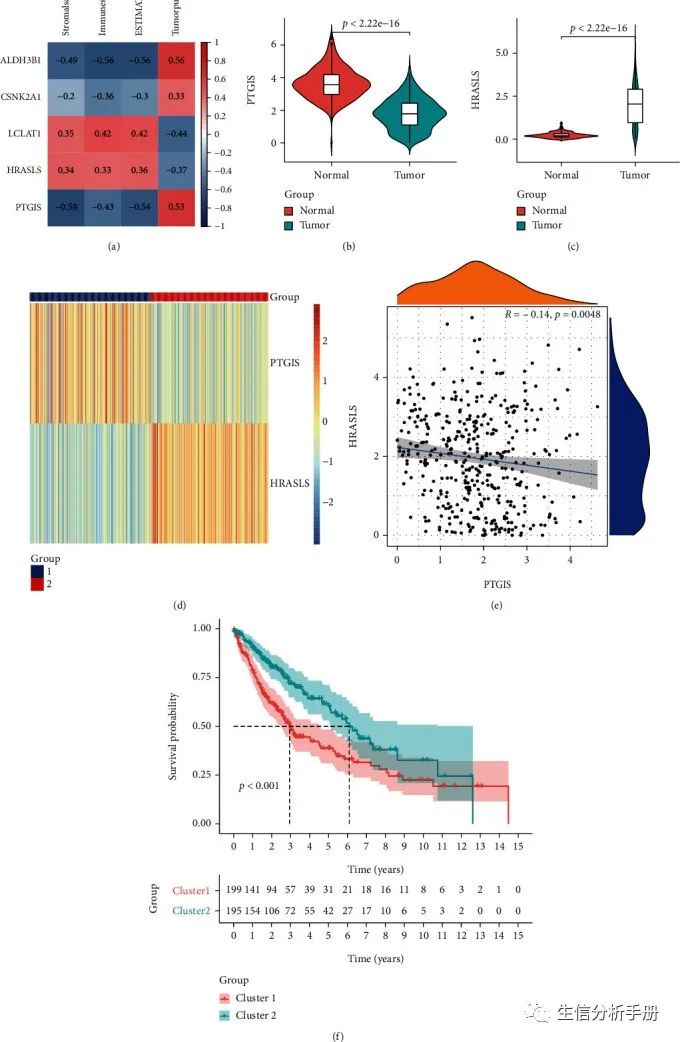
基于PTGIS和HRASLS的LUSC一致性聚类
为了探索脂质代谢在LUSC肿瘤免疫中的作用,作者评估了独立预后基因的表达与ESTIMATE指数在LUSC中的相关性(图8(a))。考虑到与OS和ESTIMATE指数的相关性,作者在后续分析中包括了PTGIS和HRASLS。作者比较了TCGA队列中这两个基因的表达,并发现肿瘤组织中PTGIS的表达低于正常组织(图8(b)),而HRASLS的表达高于正常组织(图8(c))。接下来,作者根据PTGIS和HRASLS的表达分布对394例LUSC患者进行了一致性聚类,并将样本分为两个亚型(图8(d)和补充图3)。一致性聚类热图显示,在亚型1(n = 199)中,PTGIS的表达高而HRASLS的表达低,而在亚型2(n = 195)中,PTGIS的表达低而HRASLS的表达高。由于这两个基因在两个亚型中呈相反趋势,作者研究了PTGIS和HRASLS之间的Spearman相关性,并发现它们之间存在弱的负相关(R = -0.14,p = 0.0048)(图8(e))。作者比较了两个亚型之间的临床特征差异,发现亚型1的pT、pN和pTNM分期高于亚型2(表3)。此外,亚型2的预后明显优于亚型1(图8(f))。这些结果表明PTGIS和HRASLS将TCGA队列分为具有不同特征的两个分子亚型。
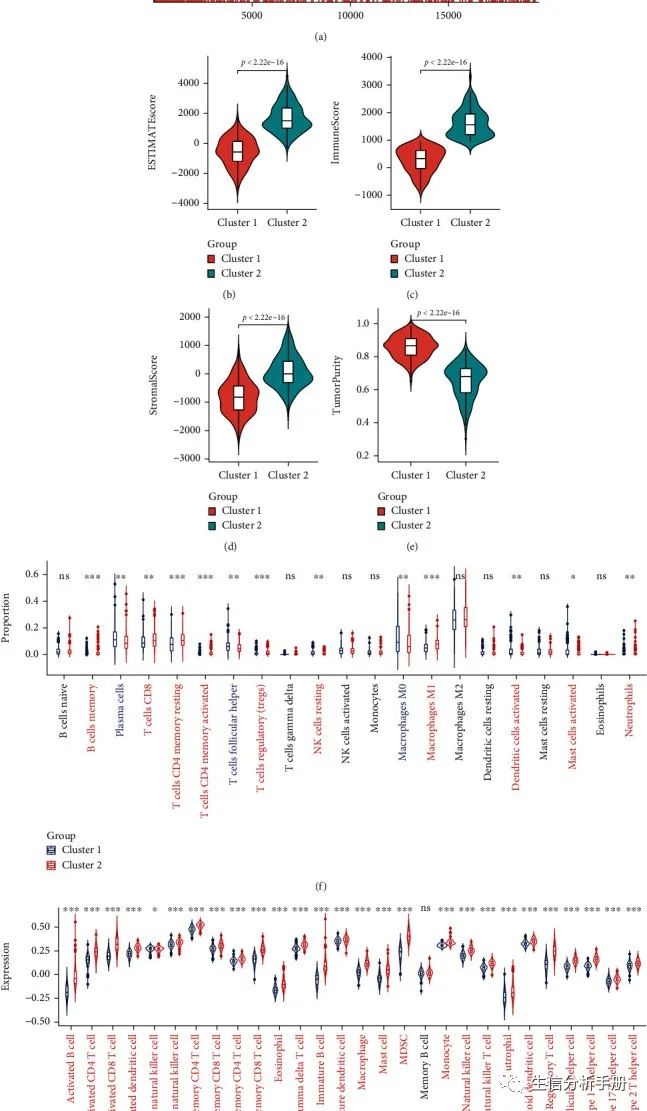
两个群集之间免疫特性的比较
为了了解两个聚类之间的功能富集差异,作者将两个聚类的所有差异表达基因(DEGs)包括在GSEA分析中。在MSigDB Collection(c5.all.v7.4.symbols.gmt)的富集分析中,作者发现了许多与免疫相关的重要通路,包括免疫应答的激活、急性炎症反应和适应性免疫应答,而这些通路在聚类2中得到了增强(图9(a))。然后,作者使用ESTIMATE、CIBERSORT和ssGSEA比较了两个聚类的免疫浸润情况。ESTIMATE显示聚类2的ESTIMATEScore、ImmuneScore和StromalScore较高(图9(b)–9(d)),但TumorPurity较低(图9(e))。22种免疫细胞的浸润热图显示,在LUSC中,M2巨噬细胞的比例明显高于其他类型的免疫细胞(附图4)。CIBERSORT显示聚类2中CD8 T细胞和M1巨噬细胞的比例较高(图9(f))。ssGSEA显示聚类2中27种免疫细胞的表达较高(如激活的B细胞、激活的CD4 T细胞、激活的CD8 T细胞和自然杀伤细胞)(图9(g))。这些结果显示,亚型2的免疫细胞浸润比亚型1更强。
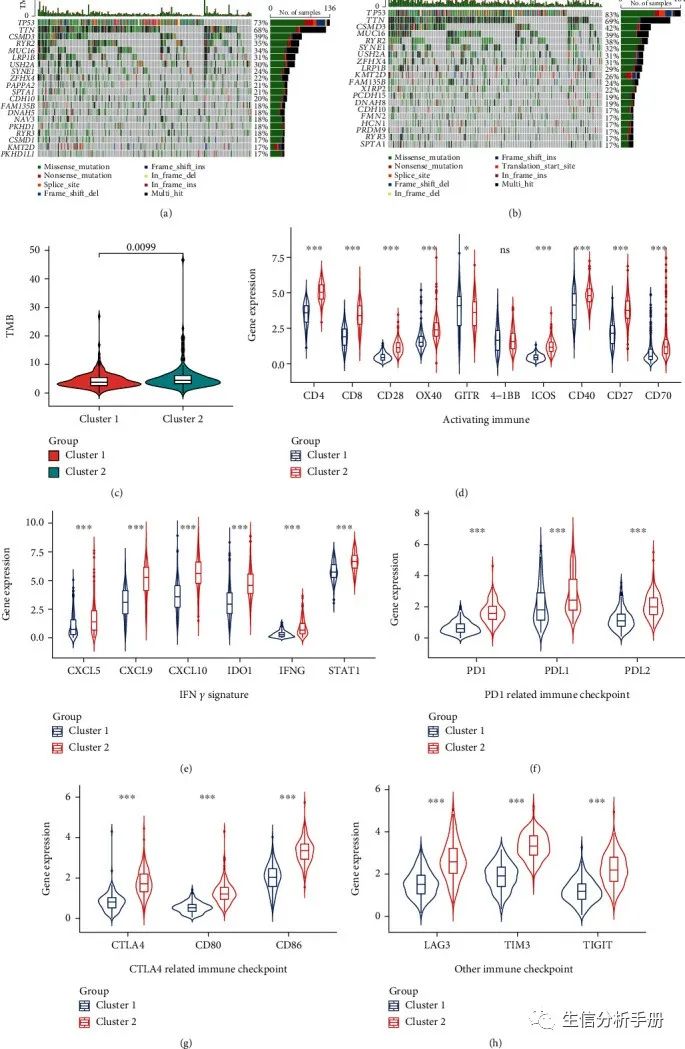
两个亚型之间基因突变的比较和免疫疗法敏感性评估
TMB水平影响肿瘤免疫浸润和免疫治疗的疗效[10]。因此,作者评估了这两个亚型的体细胞突变。图10(a)和10(b)展示了这两个亚型的突变景观,而亚型2的TMB水平高于亚型1(图10(c))。这表明亚型2在肿瘤免疫浸润方面可能更丰富,对肿瘤免疫治疗更敏感。因此,作者首先比较了这两个亚型中常见免疫调节标志物的表达,并发现亚型2中免疫激活和INF γ信号通路标志物的表达水平明显高于亚型1(图10(d)和10(e))。然后,作者比较了这两个亚型之间的免疫检查点的表达,以评估LUSC对免疫治疗的敏感性,并发现亚型2中与PD1相关的免疫检查点(PD1、PDL1和PDL2)的表达(图10(f)),与CTLA4相关的免疫检查点(CTLA4、CD80和CD86)的表达(图10(g)),以及其他报道的免疫检查点(LAG3、TIM3和TIGIT)的表达(图10(h))均显著高于亚型1。
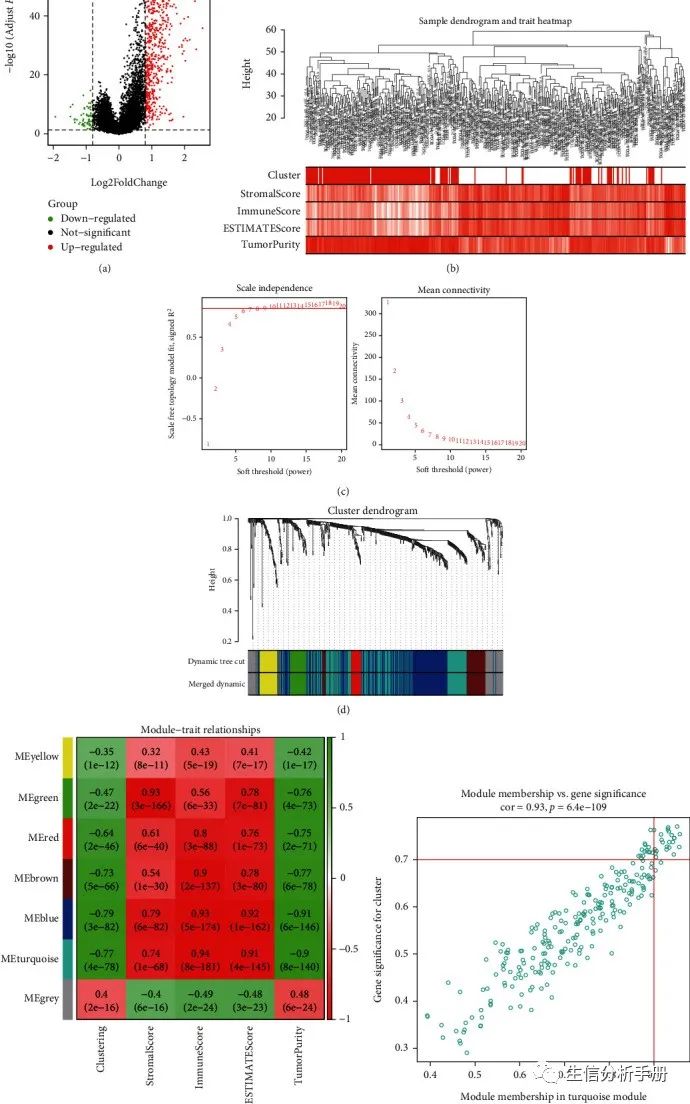
在加权基因共表达网络分析(WGCNA)中鉴定与聚类和免疫相关的模块基因
通过筛选阈值 |log2FC| > 0.8 和 p 值 < 0.05,作者鉴定出763个差异表达基因(包括697个上调基因和66个下调基因)(图11(a))。包含394个LUSC样本和763个差异表达基因的表达矩阵被纳入WGCNA分析。样本聚类分析排除了8个异常样本,保留了386个样本(附图5)。聚类和免疫浸润评分的热图如图11(b)所示。作者选择9作为最佳软阈值(图11(c)),每个共表达网络模块的最小基因数为30,得到了7个网络表达模块(图11(d))。为了确定与聚类和免疫相关的模块,作者分析了模块与特征之间的相关性(图11(e))。蓝绿色模块与聚类(r = -0.77, p = 4 × 10 −78 )和免疫(r = 0.94, p = 8 × 10 −181 )有很强的相关性,因此将其作为后续研究的对象。最后,基于 |MM| > 0.9 和 |GS| > 0 的条件。7,作者从蓝绿色共表达网络模块(图11(f))中获得了28个关键基因(CD53,SLA,ARHGAP15,SASH3,EVI2B,GIMAP4,SELPLG,CYTH4,PTPRC,NCKAP1L,WAS,RCSD1,IL10RA,CD74,IRF8,TNFRSF1B,BTK,SNX20,APBB1IP,CD37,DOCK2,CD4,GIMAP7,LILRB1,IL16,DOCK8,CORO1A和BIN2)。
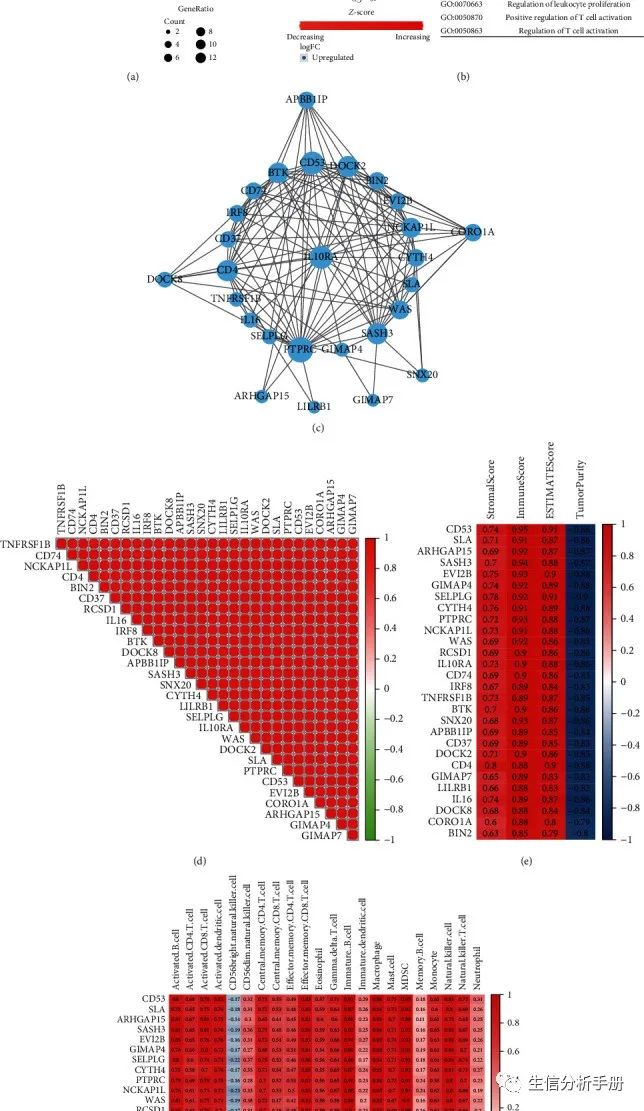
关键基因的富集分析及其与肿瘤免疫浸润的关系
作者对通过WGCNA获得的28个关键基因进行了GO富集分析,发现最重要的GO项目是免疫细胞的激活和增殖(图12(a)),并且这些过程得到了增强(图12(b))。PPI分析显示IL10RA位于关键基因相互作用网络中(图12(c)),这些基因之间存在强烈的正相关关系(图12(d))。关键基因与肿瘤免疫浸润分析(ESTIMATE和ssGSEA)的Spearman相关分析表明,所有关键基因与肿瘤免疫密切相关(图12(e)和12(f))。
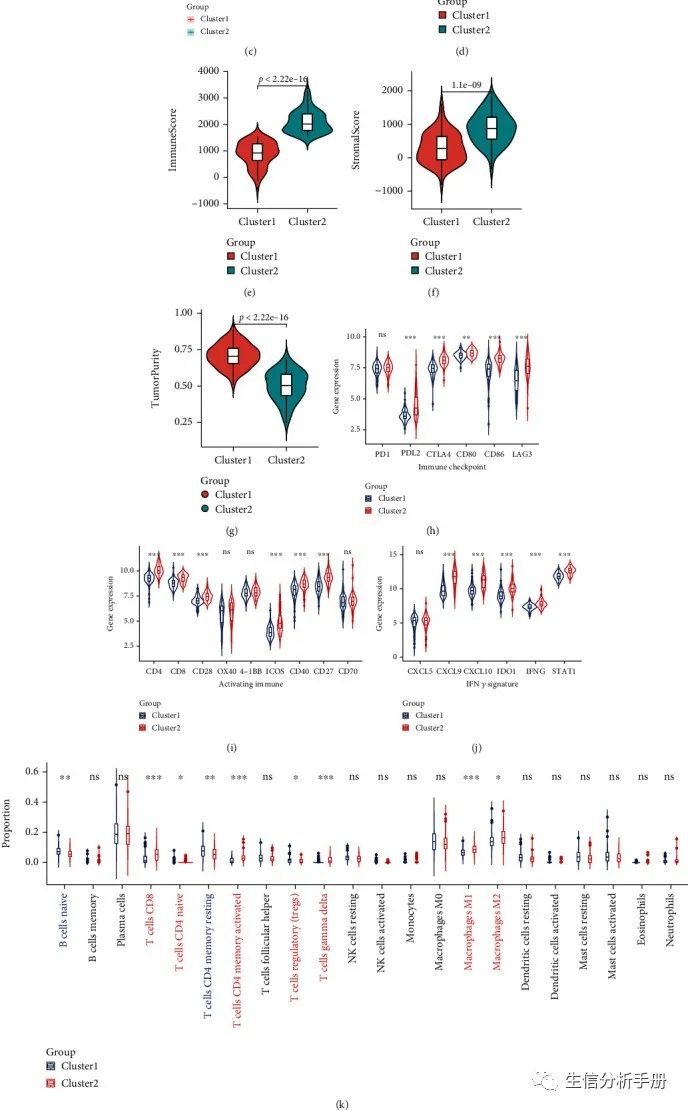
GEO验证PTGIS和HRASLS在评估LUSC的预后和肿瘤免疫中的作用
作者使用与TCGA队列相同的共识聚类方法将GSE4573队列分为两个亚型,并发现PTGIS和HRASLS在这两个亚型中的表达分布与TCGA队列中的类似(图13(a))。临床病理数据的比较显示,亚型1的pN和pTNM分期高于亚型2(表4)。Spearman相关分析显示,PTGIS和HRASLS的表达水平呈负相关,与TCGA队列相似(r = -0.21,p = 0.018)(图13(b))。生存分析显示,在LUSC中,PTGIS的高表达和HRASLS的低表达与不良预后相关(补充图6),亚型2的OS率明显高于亚型1(图13(c))。作者使用与TCGA队列相同的分析方法比较了两个亚型之间的肿瘤免疫浸润程度,包括ESTIMATE(图13(d)–13(g)),免疫调节标志物的表达(图13(h)–13(j)),CIBERSORT(图13(k))和ssGSEA(图13(l))。毫不奇怪,亚型2的免疫系统更为活跃。
作者确定了与LUSC的预后和肿瘤免疫相关的LMRGs PTGIS和HRASLS。在体外实验中,PTGIS和HRASLS影响了LUSC的增殖、迁移和侵袭能力。基于PTGIS和HRASLS表达分布的共识聚类,TCGA和GSE4573队列中的LUSC患者被分为两个亚型。亚型2具有更好的预后、更强的免疫浸润、更高的免疫检查点表达和更高的TMB水平,相比之下,亚型1则较差。因此,作者的研究表明,PTGIS和HRASLS在指导LUSC的免疫治疗方面具有潜在的临床价值,同时也是预后和肿瘤免疫的生物标志物。
往期推荐