|
| 江江包今天刷文献时发现一篇工作量很丰满的生信文章,不过它只发了4.4分,江江包顿时觉得有点亏。不过想到最新影响因子发布后几乎所有杂志的影响因子又下降了一波,因此江江包查看了下文章发表时的影响因子,果不其然,当时的影响因子是6分,这么想来也没有太亏。忘记说啦,这篇文章投中的杂志是Computational and Structural Biotechnology Journal。这是一本OA期刊,版面费3230美元,从投稿到接受的平均时间为3个月。收稿范围由四常规部分、智能医院部分、纳米科学与先进材料部分、量子生物学和生物光子学部分组成: 江江包只截取生信友好的部分来给大家看下,手里有类似的稿件也可考虑下这本二区杂志(下面的内容是翻译过后的)。下面还是一起来看看这篇生信文章吧,手里已经有成稿的朋友可以和这篇文章的工作量对比一下,心里就大概有个数了(ps:江江包这里有各种生信方案和分析技术,想要做生信文献复现欢迎来call~) |
|
定制生信分析
云服务器租赁 加好友备注“66”领取试用 |
|
| 题目:单细胞和批量分析的综合免疫基因组学分析揭示了肺腺癌中的新型肿瘤抗原和亚型特异性治疗剂杂志:Computational and Structural Biotechnology Journal |
研究背景近年来,具有良好安全性和功能特性的mRNA疫苗在癌症免疫治疗中获得了显著的发展势头。然而,肺腺癌(LUAD)的稳定的免疫分子亚型和LUADmRNA疫苗开发所需的新肿瘤抗原仍然难以捉摸。因此,迫切需要一种新的方法来识别适合的LUAD亚型和潜在的肿瘤抗原。研究思路利用癌症基因组图谱(TCGA)、基因型组织表达(GTEx)和基因表达综合数据库(GEO)检索基因表达数据。通过单细胞和组织块转录组分析,使用七种机器学习(ML)算法开发了肺腺癌免疫学多组学分类(LIMOC)系统。随后,应用了一系列方法来识别新的肿瘤抗原。 |
|
主要结果研究通过应用七种聚类算法构建了LIMOC系统,鉴定出三种免疫亚型:LIMOC1、LIMOC2和LIMOC3。LIMOC2亚型的患者显示出更短的中位总生存期,并且在不同的独立队列中,LIMOC2始终与最差的临床结果相关。此外,与其他研究团队报告的分子分类的关联分析进一步验证了LIMOC系统分类的准确性。 |
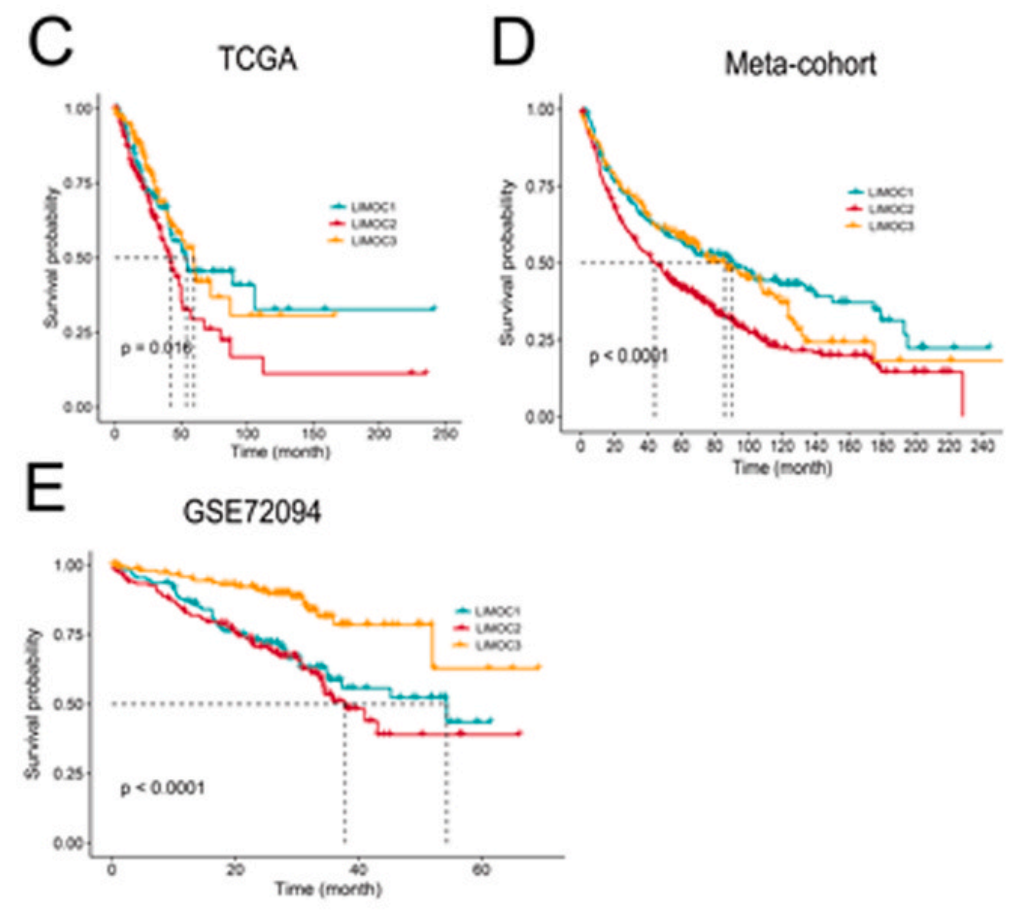
图1 在LUAD队列中识别LIMOC系统,并将其与其他最先进的LUAD亚型进行比较(C)TCGA中三种LIMOCs的不同总体生存结果。(D)元队列中三种LIMOCs的不同总体生存结果。(E)GSE72094中三种LIMOCs的不同总体生存结果。(G-K)分别来自(G)Song的研究、(H)Wei的研究、(I)Deng的研究、(J)Li的研究以及(K)Thorsson的研究的不同总体生存结果。 |
| 鉴于其与免疫治疗疗效已建立的联系,评估了三个亚型中的肿瘤突变负担(TMB)。发现LIMOC2亚型具有更高的肿瘤突变负担(TMB),并且与APOBEC特征相关的突变更多。特别是,LIMOC2在包括TP53在内的多个频繁改变的基因中显示出最多的突变。TP53的排除对LIMOC分类有显著影响,表明其在分类中的重要性。此外,LIMOC2还表现出更高的染色体不稳定性,特别是8q24.21区域的拷贝数增加,而Ch9p21.3区域则表现出特异性删除。这些发现进一步支持了LIMOC2亚型可能对免疫治疗有较差反应的假设。 |
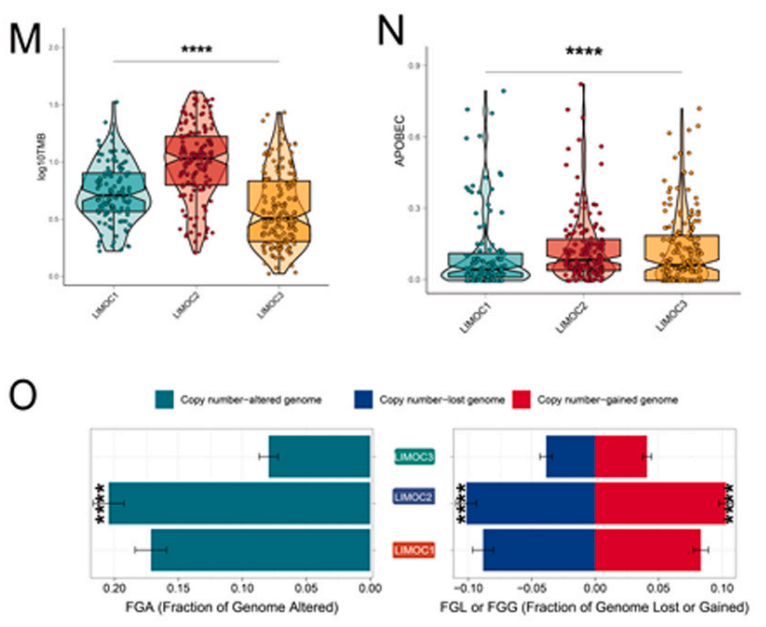
图1 在LUAD队列中识别LIMOC系统,并将其与其他最先进的LUAD亚型进行比较(M-N)展示了TMB(M)和APOBEC(N)对特定变化的贡献。(O)分数基因组变化(FGA)和增/减(FGA/FGG)的分布。 |
| 评估了68个染色质重塑调控子和23个其他调控子的活动,以识别亚型之间的潜在差异。差异活动分析揭示了三个亚型之间不同的调控模式。发现LIMOC2亚型的调控与人类Fox基因家族有关,而LIMOC3亚型则受AR和ESR2激活的影响。通过致癌途径的分析,发现LIMOC2激活了细胞周期和PI3K途径,可能影响DNA损伤反应和细胞转移。此外,LIMOC2还与NRF2途径的激活有关,这可能促进了免疫冷微环境的形成。代谢途径分析显示,三个亚型在代谢重编程上各有特点,这可能对他们的分子特征和临床结果有重要影响。 |
图2 TCGA队列中LIMOC系统中调节子和通路的差异活性 |
|
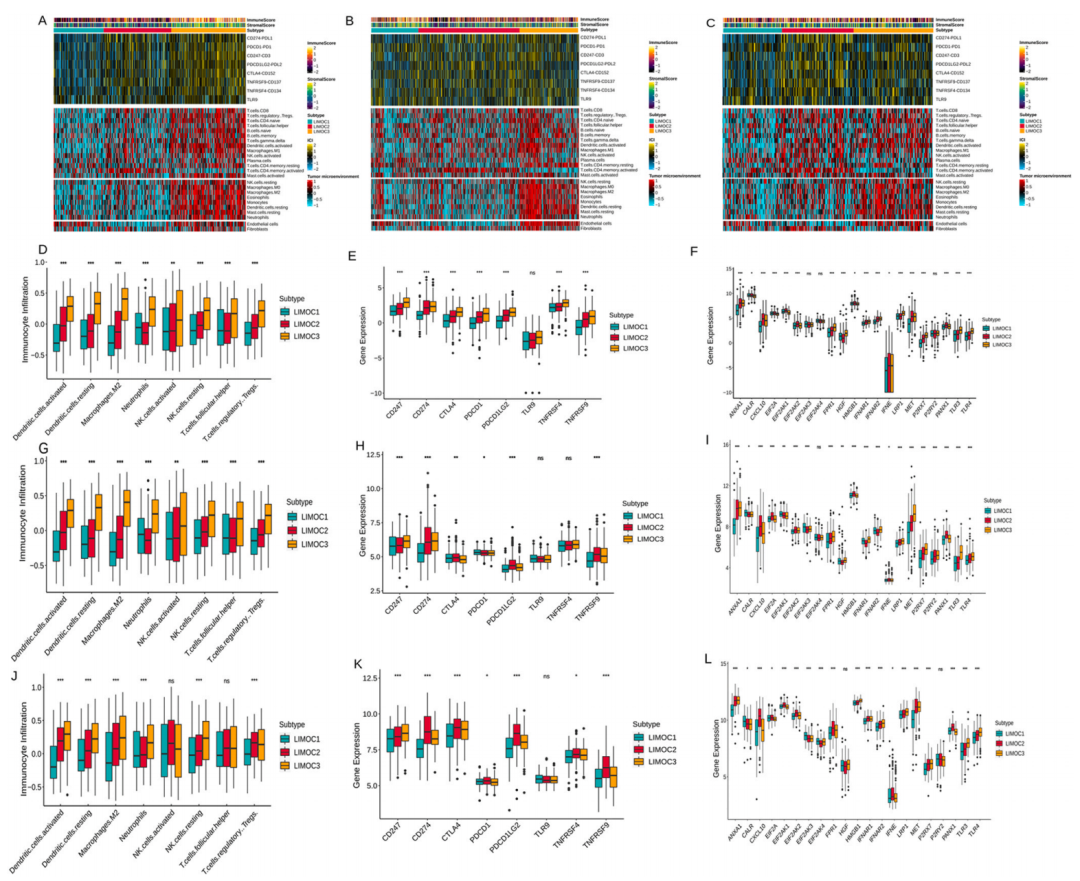
LUAD的三种亚型在临床特征、免疫细胞丰度和分子表达上各有不同。LIMOC2亚型主要影响老年男性,且总生存期较短。该亚型的免疫细胞特征为活化记忆CD4+T细胞数量增多。相比之下,LIMOC3亚型的免疫细胞特征为多种免疫细胞类型丰度增加,特别是M2型巨噬细胞、中性粒细胞和Tregs。此外,LIMOC3亚型的免疫检查点(ICPs)表达水平较高,而LIMOC2亚型的免疫原性细胞死亡(ICD)调节因子表达水平上调,这可能影响其对免疫治疗的响应。 |
|
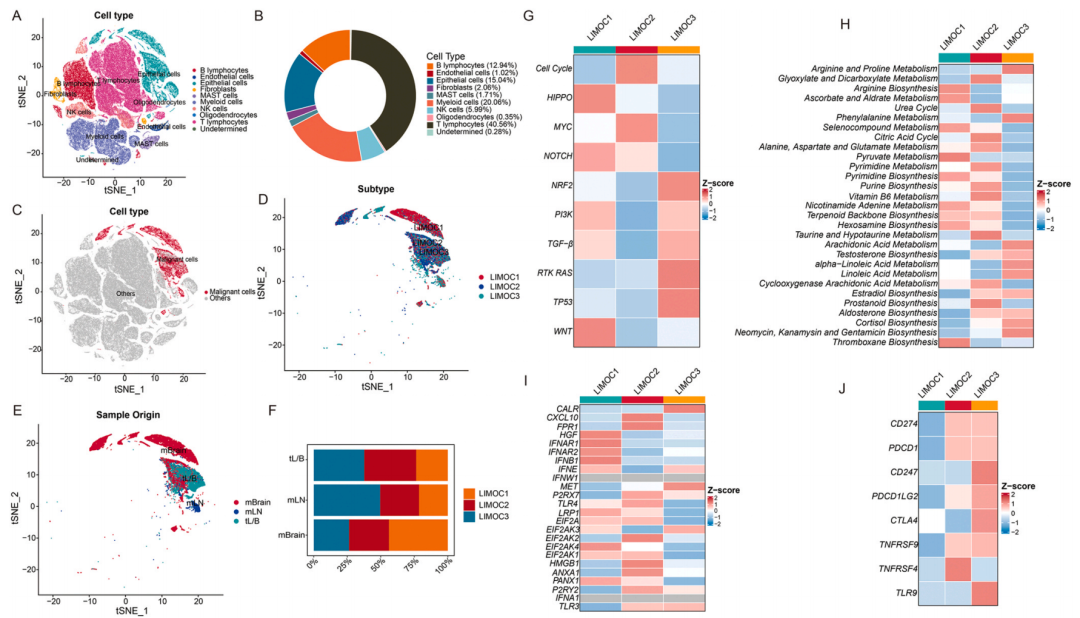
| 通过单细胞RNA测序分析,研究者揭示了LUAD三种亚型(LIMOC1、LIMOC2和LIMOC3)在单细胞水平上的分子特征。LIMOC2亚型中细胞周期途径激活,而LIMOC1和LIMOC3亚型中TGF-β途径激活。此外,LIMOC3亚型的免疫检查点(ICPs)表达强烈,而LIMOC2亚型中多种免疫原性细胞死亡(ICD)调节因子上调,反映了不同亚型的免疫微环境和代谢重编程的差异。这些发现为理解LUAD的肿瘤异质性及开发亚型特异性治疗策略提供了重要信息。 |
|
| 通过综合分析TCGA和GTEx数据库中的数据,研究者筛选出三个有潜力成为LUADmRNA疫苗靶点的肿瘤抗原候选基因:CHIT1、LILRA4和MEP1A。这些基因在LUAD中表达上调、频繁突变和扩增,并与患者的总生存期和无进展生存期显著相关。在蛋白水平上,这些抗原在LUAD组织中的表达水平高于邻近正常组织,且siRNA敲低这些基因能够抑制LUAD细胞系A549的增殖。这些结果表明,针对这些抗原的mRNA疫苗可能有效诱导抗肿瘤免疫反应。 |

图5 识别LUAD中的潜在肿瘤抗原,并分析正常组织与肿瘤组织中肿瘤抗原(CHIT1、LILRA4和MEP1A)的表达差异 |
| 7. LIMOC2亚型中APC标记与三种抗原之间的关系 LIMOC2亚型的LUAD患者具有较高的肿瘤突变负担和其他可能促进肿瘤抗原产生和APCs肿瘤微环境渗透的因素。研究发现,LIMOC2亚型中ICPs表达较低而ICD调节因子表达升高,这表明LIMOC2亚型可能对mRNA疫苗有良好的响应。特别是,CHIT1和LILRA4与APCs的共刺激分子CD40、CD80和CD86的表达水平呈正相关,这支持了针对这些抗原的mRNA疫苗可能在LIMOC2亚型中特别有效的观点。尽管MEP1A与某些APC标记物也表现出一定的相关性,但CHIT1和LILRA4与APC标记物的相关性更强。 |
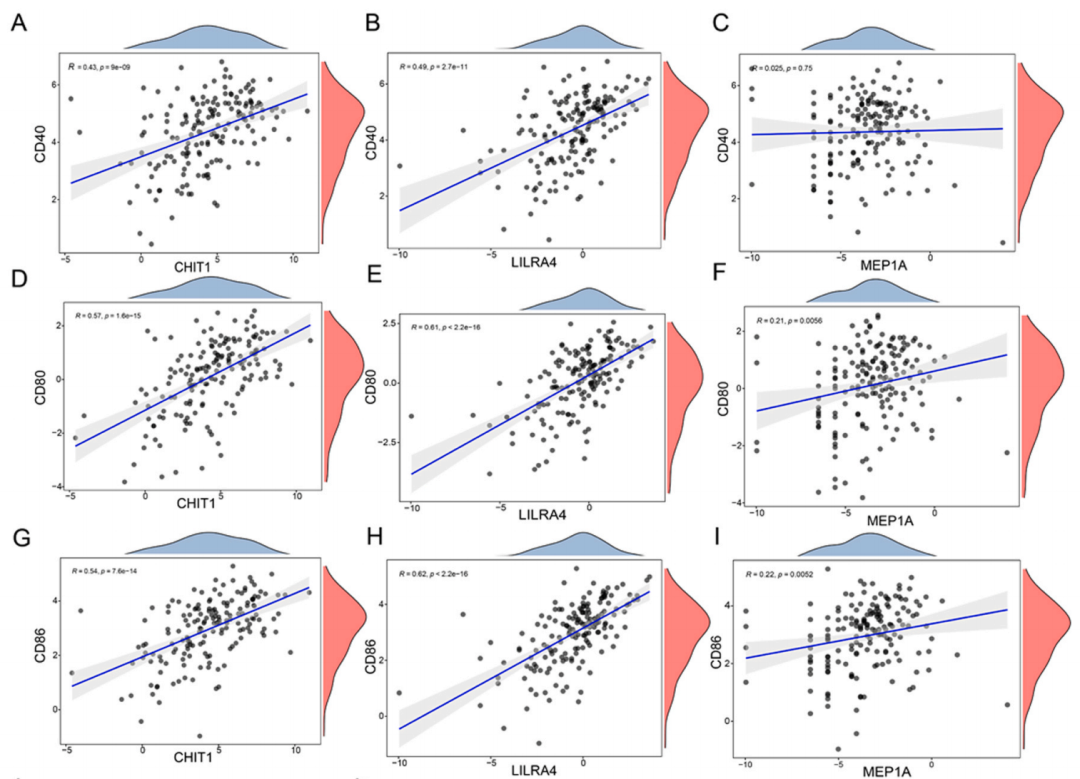
图6 LUAD中的APC标志物和三种抗原表达水平与LIMOC2亚型的相关性(A-C)分别展示了CD40与CHIT1(A)、LILRA4(B)、MEP1A(C)表达水平之间的相关性。
(D-F)分别展示了CD80与CHIT1(D)、LILRA4(E)、MEP1A(F)表达水平之间的相关性。(G-I)展示了CD86与CHIT1(G)、LILRA4(H)、MEP1A(I)表达水平之间的相关性。 |
| 对LUAD中三种候选肿瘤抗原CHIT1、LILRA4和MEP1A的潜在生物学功能进行了分析。通过相关性分析和途径富集分析,发现这些抗原与免疫相关的生物学过程密切相关。CHIT1主要与Th17细胞分化、趋化因子信号通路和细胞因子相互作用相关;LILRA4与免疫效应过程的调节、细胞因子产生的正向调节和T细胞激活相关;MEP1A则与B细胞受体信号通路相关。这些发现为开发针对这些抗原的mRNA疫苗提供了理论依据,表明它们在免疫反应中的潜在作用,可能有助于设计有效的肿瘤免疫治疗策略。 |
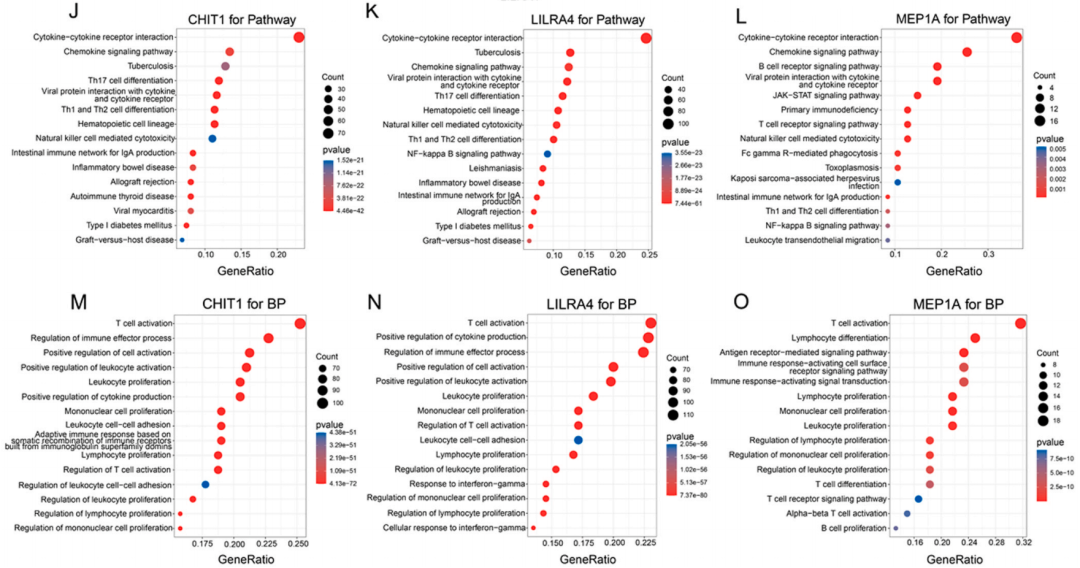
(J-L)分别对CHIT1(J)、LILRA4(K)、MEP1A(L)相关的类别进行了KEGG分析。(M-O)分别对CHIT1(M)、LILRA4(N)、MEP1A(O)相关的类别进行了GO分析。 |
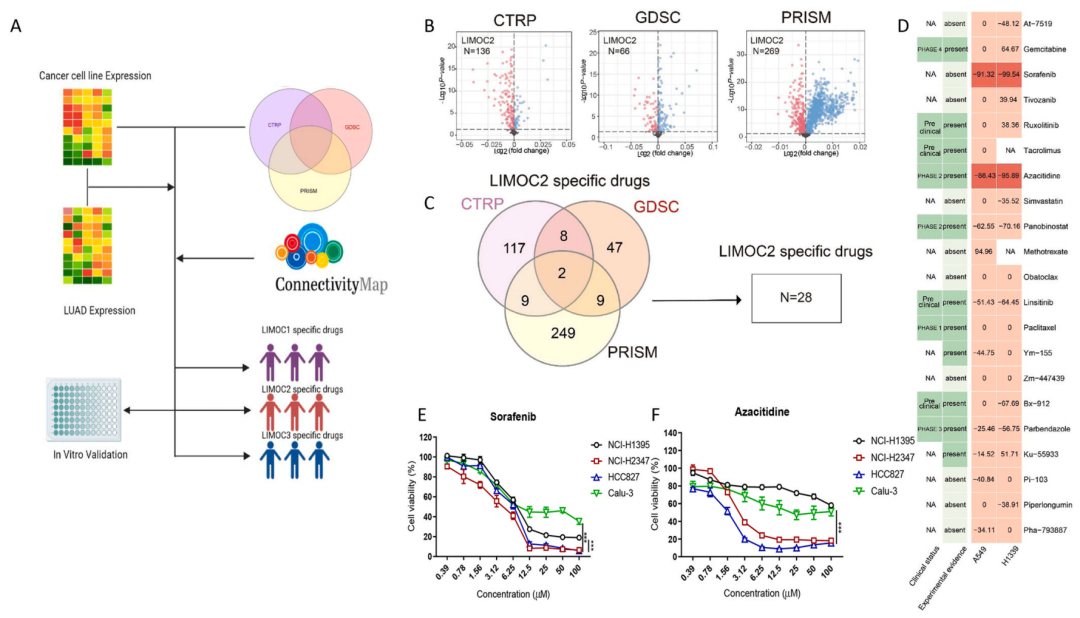
| 通过综合分析不同数据集的药物反应数据,筛选出可能对该亚型有效的治疗药物。通过pRRophetic软件包的calcPhenotype功能评估药物敏感性,结合limma软件包的敏感性分析,鉴定出多个潜在的LIMOC2特异性药物。进一步通过文献调研和CMap数据库分析,缩小了候选药物范围,并特别关注了Sorafenib和Azacitidine两种化合物。体外实验结果支持了这两种药物作为LIMOC2患者潜在治疗剂的有效性,且这两种药物在以往的研究中也显示出了治疗潜力。 |
|
| (B)在每个数据集(CTRP、GDSC和PRISM)中鉴定的LIMOC2特异性药物(Wilcoxon秩和检验,P<0.05)。(D)根据多个来源的证据鉴定最有前途的亚型特异性药物。(E-F)使用细胞活性测定在4种LUAD细胞系中验证Sorafenib(E)和Azacitidine(F)药物反应的三个亚型之间的差异。 |
文章小结这篇文章生信和实验结合不错,工作量还是蛮多的。不过可能在选题上不够新颖(这个方向有点卷),肺腺癌分型的研究有很多了,做出创新比较难,不过作者除了肿瘤分型研究,落脚点还在新型候选抗原筛选上并加入了亚型特异性治疗药物的筛选验证,一波小连招将创新度拉高了不少。所以同样是烂大街的肿瘤分型研究,思路不同,落脚点不同也能做出新的东西的!好啦,想做生信分析却苦无技术的朋友们可以call江江包哦~专业技术团队竭诚为您服务! |