作者:梅菜
编辑:李宝珠,三羊
上海国家应用数学中心(上海交通大学分中心)俞章盛课题组、王宇光课题组与合作者,共同开发了深度学习系统 IGI-DL,通过组织病理学图像,为没有空间转录组数据的癌症患者预测肿瘤微环境信息,从而实现精确的癌症预后。
世界卫生组织 2022 年发布的报告指出,癌症等非传染性疾病 (NCDs)) 已超过传染病,成为「全球头号杀手」。中国国家癌症中心发布的最新数据显示,2022 年中国约有 482.47 万新发癌症病例和 257.42 万新发癌症死亡病例。
很长一段时间里,人们「闻癌色变」,但其实癌症作为一种慢性病,1/3 是可以预防的,1/3 是可以通过早发现、早诊断、早治疗达到治愈的,1/3 不可治愈,但通过适当治疗可以控制、获得较好的生活质量并延长生存期。其中,预防主要是通过提高自身免疫力、定期体检等途径,关注个人健康。而在确诊癌症后,预后分析十分重要。
癌症预后是指预测癌症患者的可能病程和结局,预后分析有助于提高癌症患者的生存机会。过去,研究人员基于空间转录组学 (ST) 技术,从空间基因表达的角度对肿瘤微环境 (TME) 进行表征,从而区分癌症患者的不同预后亚组。然而,ST 的高昂成本和长实验周期阻碍了其应用于大规模癌症患者队列进行生存预测。与之相比,组织学图像具有成本效益,在临床环境中易于获取,并且能够提供有关肿瘤形态的丰富信息,是分子水平 TME 分析的更好替代品,可实现更精确的癌症预后。
近日,上海国家应用数学中心(上海交通大学分中心)俞章盛课题组(生命科学技术学院/医学院临床研究中心),王宇光课题组(自然科学研究院/数学科学学院)与合作者在 Cell Reports Medicine 发表了题为「Harnessing TME depicted by histological images to improve cancer prognosis through a deep learning system」的论文。该研究开发了一个深度学习系统,可以通过组织病理学图像,为没有空间转录组数据的癌症患者预测肿瘤微环境信息,从而实现精确的癌症预后。
研究亮点:
* 通过组织病理学图像为没有 ST 数据的癌症患者预测 TME 信息
* 以 IGI-DL 为特征的 TME 提高了癌症生存预测的准确性
* 大幅拓展了基因空间表达信息在大型生物医学病理图像公共数据库的使用
论文地址:
https://www.cell.com/cell-reports-medicine/fulltext/S2666-3791(24)00205-2
关注公众号,后台回复「肿瘤微环境」获取完整 PDF
数据集:评估 3 种实体瘤类型组织样本
本研究为了评估模型在结直肠癌 (CRC)、乳腺癌和皮肤鳞状细胞癌 (cSCC) 这三种不同实体瘤类型组织样本上的性能,使用了 3 种不同的数据集。
对于结直肠癌,研究人员使用了来自上海交通大学医学院附属瑞金医院 10 名 CRC 患者的 10 个 ST 数据集的 41,492 个点,这些数据集由 10× Visium 测序,作为留一患者 (leave-one-patient-out) 验证集,如下表所示。
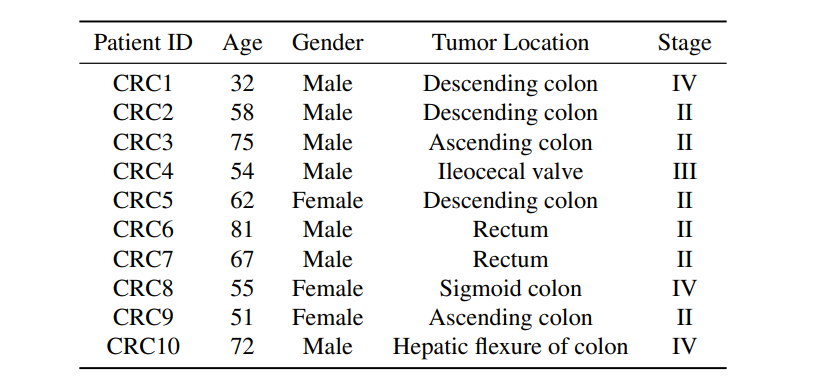
内部 CRC 数据集的临床特征
对于乳腺癌,研究人员使用了来自 27 名患者的 92 个组织样本的 34,678 个点,这些样本通过传统的 ST 技术进行了测序,作为留一患者验证集,如下表所示。
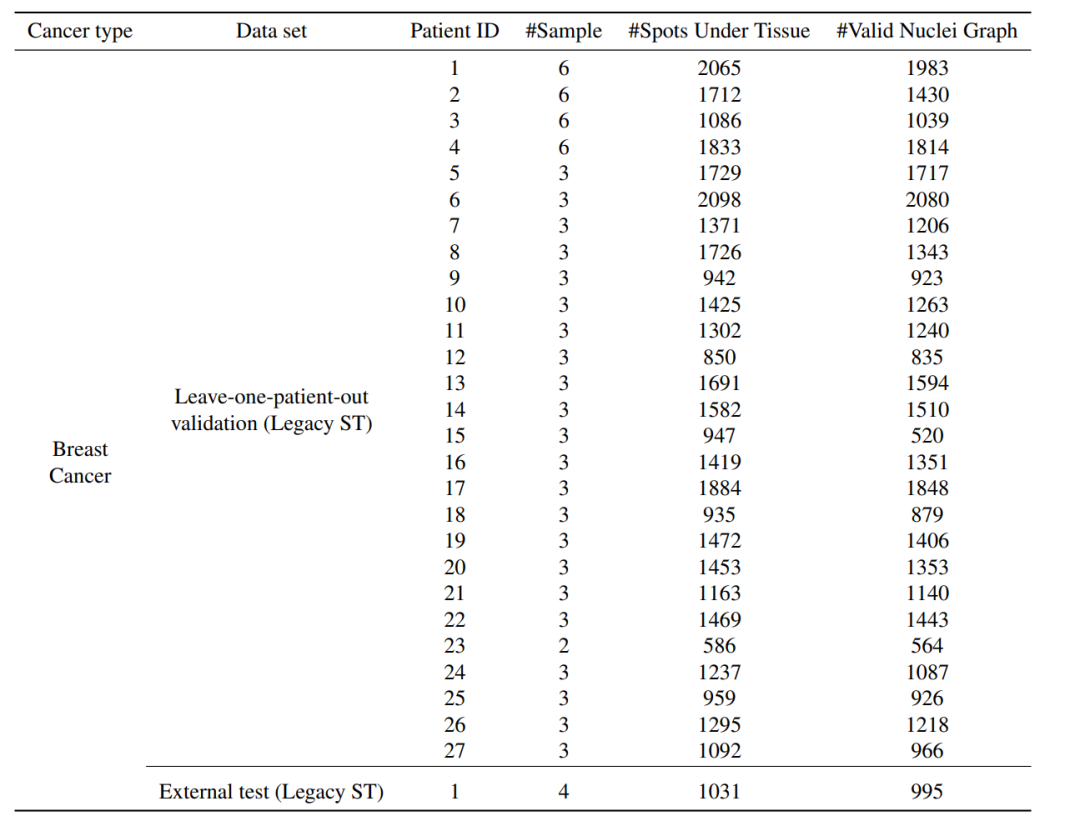
乳腺癌空间转录组数据集汇总
对于皮肤鳞状细胞癌,研究人员使用了来自 4 名患者的 12 个组织样本的 4,353个 点,这些样本通过传统的 ST 技术进行了测序,作为留一患者验证集,如下表所示。
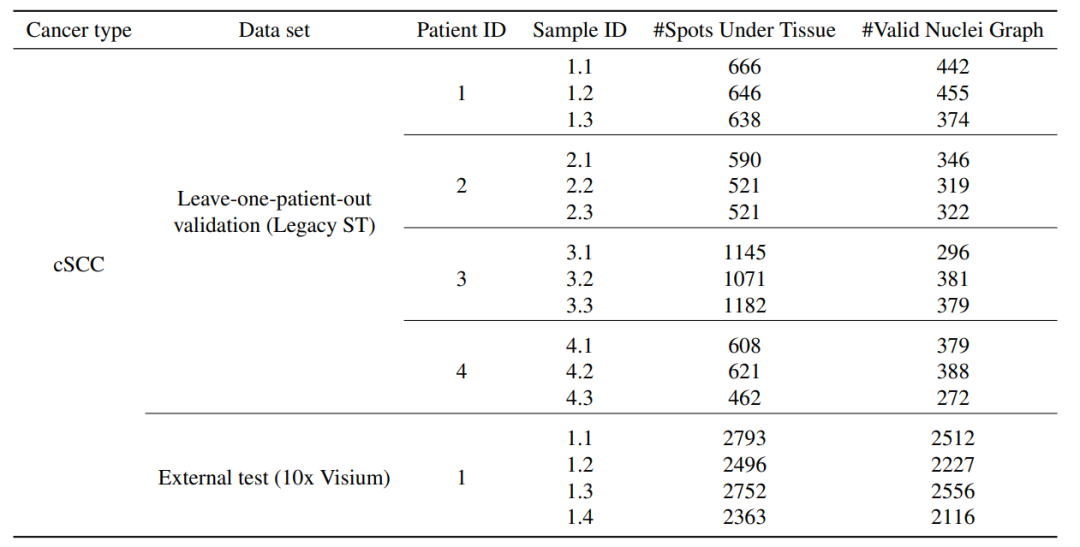
cSCC 空间转录组数据集汇总
模型架构:新型深度学习系统改善癌症预后
在本研究中,研究人员开发了一个深度学习系统,可以利用组织学图像描绘的 TME 改善癌症预后。
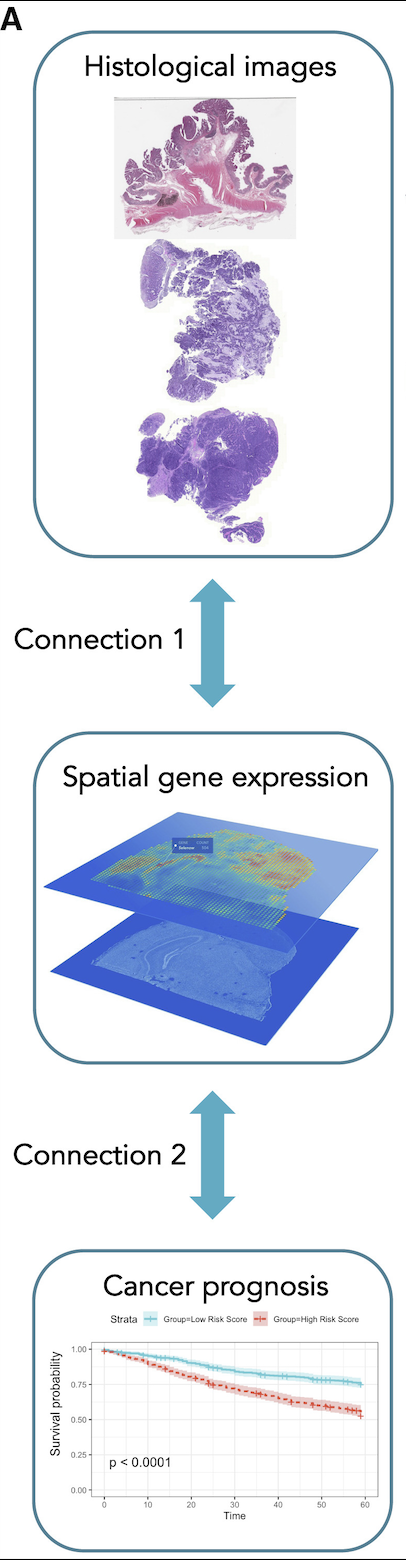
该深度学习系统包含两部分
该系统包含两个部分:
第一部分(上图中 Connection 1)是基于 integrated graph 和图深度学习的模型 (integrated graph and image deep learning, IGI-DL),该模型采用卷积神经网络和图神经网络将 H&E 染色组织学图像 (histological images) 投射到基因表达空间 (gene expression space)。
第二部分(上图中 Connection 2),研究人员在癌症基因组图谱 (TCGA) 数据集中的结直肠癌队列和乳腺癌队列中,使用 super-patch graph 和 IGI-DL 预测的空间基因表达 (spatial gene expression) 作为节点特征 (node features),进行了预后预测,然后在外部测试集 MCO-CRC (Molecular and Cellular Oncology colorectal cancer) 中进行了验证。
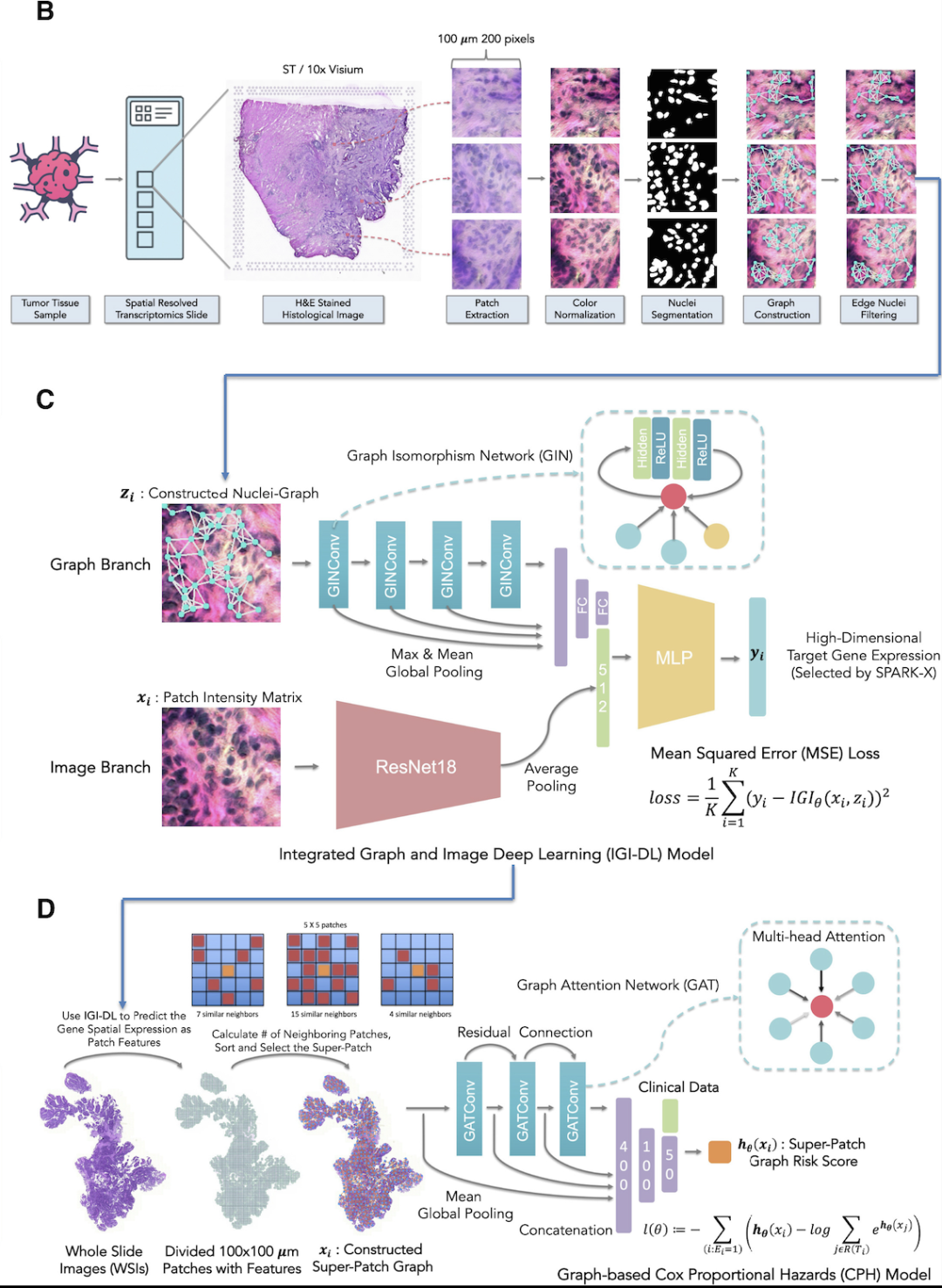
深度学习系统的工作流程
具体而言,该系统的构建包括 3 个步骤:H&E 染色组织学图像预处理、空间基因表达预测模型和基于预测的空间基因表达 super-patch graph 生存模型。
*H&E 染色组织学图像 (H&E-stained histological image) 预处理:
首先根据每个点的坐标,将每个 H&E 染色的组织学图像分割成多个非重叠的 200×200 像素的 patches,分辨率为 0.5 μm/pixel;*空间基因表达预测模型:
对于每个 patch,研究人员建立了一个 Nuclei-Graph,其中由 Hover-Net24 分割的每个细胞核表示为一个节点 (node),并且每个细胞核对之间的距离决定了是否存在边缘 (edge) 连接。根据上图 C 所示的架构,研究人员使用 IGI-DL 模型来预测组织学图像中每个点的目标基因表达。*基于预测的空间基因表达的 super-patch graph 生存模型:为了进一步基于由空间基因表达勾画的 TME 进行预后预测,研究人员从每个患有癌症的患者的 H&E 染色全切片成像 (whole-slide imaging, WSI) 中构建了一个 super-patch graph,然后利用构建的 super-patch graph 和临床特征作为输入,构建了基于图的生存预测模型。研究结果:IGI-DL 模型综合表现出色
总体来看,本研究构建的 IGI-DL 模型集成了卷积神经网络和图神经网络的优势,充分利用组织病理学图像中的像素强度和结构特征,实现更准确的基因空间表达水平预测。模型在结直肠癌、乳腺癌和皮肤鳞状细胞癌这 3 种类型实体瘤中均表现出色,与 5 种现有方法相比平均相关系数提升了 0.171。
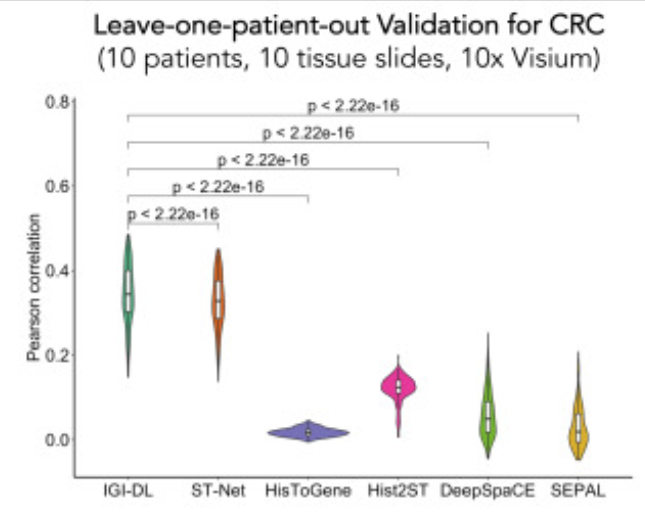
IGI-DL在CRC中空间基因表达的预测性能和可视化
对于结直肠癌,研究人员将 IGI-DL 预测的 179 个基因的 Pearson 相关性与 5 个 SOTA 模型进行比较,IGI-DL 在 10 名保留患者中实现了 0.343 的平均 Pearson 相关性,明显优于其他模型,平均增加了 0.233,如上图所示。
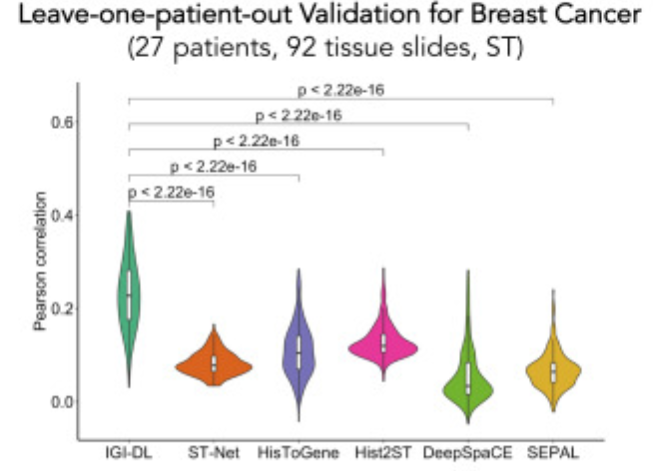
IGI-DL在乳腺癌中空间基因表达的预测性能和可视化
对于乳腺癌,研究人员将 IGI-DL 预测的 187 个基因的 Pearson 相关性与之前的模型进行比较,IGI-DL 在 27 名保留患者中实现了 0.231 的平均相关性。如上图所示,IGI-DL 模型优于所有 SOTA 模型,平均提高了 0.142。
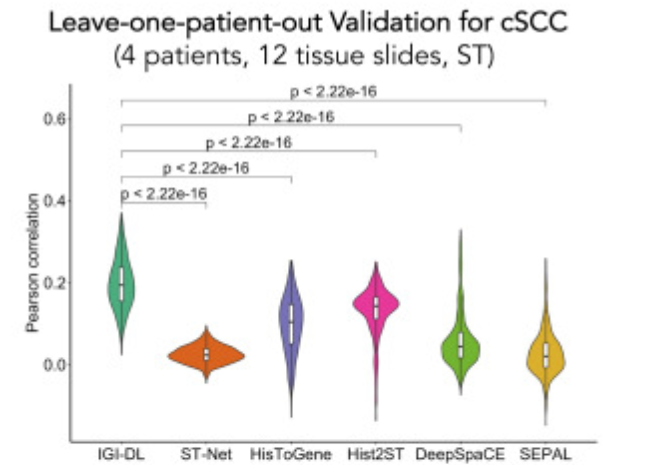
IGI-DL在cSCC中空间基因表达的预测性能和可视化
对于皮肤鳞状细胞癌,研究人员将 IGI-DL 预测的 487 个基因的 Pearson 相关性与之前的模型进行了比较,IGI-DL 在 4 名保留患者中实现了 0.198 的平均相关性,在所有模型中具有最好的性能,比其他 SOTA 模型的平均性能提高了 0.131,如上图所示。
在跨平台和跨癌种性能方面,如上述实验,对于不同癌症类型的内部验证和外部测试集,最佳的 SOTA 模型并不固定,
但 IGI-DL 模型的性能始终优于其他模型,平均改进幅度为 0.171,展现出良好的跨平台泛化能力。
更进一步,研究人员还调查了 IGI-DL 的跨癌种预测性能,其在结直肠癌上训练的模型在皮肤鳞状细胞癌的内部验证和外部测试集上表现良好,平均相关性分别为 0.204 和 0.143。然而,大多数跨癌种预测性能较训练和测试单一癌症类型时要低。这一结果表明,肿瘤区域的空间基因表达具有一定的癌症特异性,跨癌种预测存在固有的困难。
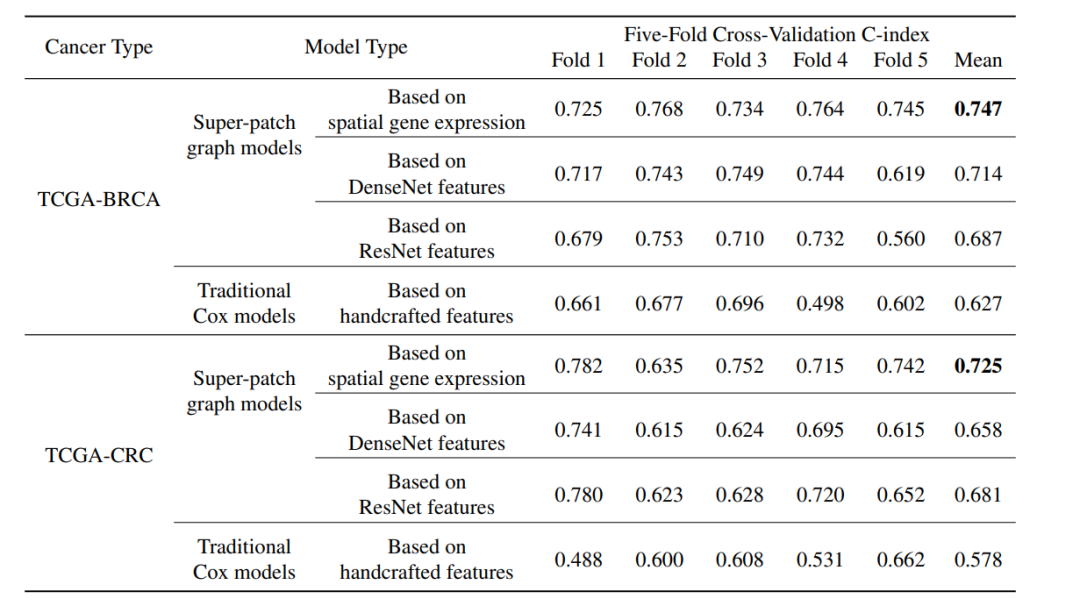
TCGA 乳腺癌和结肠癌不同生存模型的预测性能
针对预后预测性能,在癌症基因组图谱乳腺癌 (TCGA-BRCA) 队列中,基于以空间基因表达为节点特征的 super-patch graph 生存模型在 5 倍交叉验证中可以达到 0.747 的平均一致性指数 (C-index);在癌症基因组图谱结直肠癌 (TCGA-CRC) 队列中,该生存模型在 5 倍交叉验证 C-index 为 0.725,优于其它预后模型,如上图所示。
该生存预后模型对于早期患者 (I 期和 II 期) 的预后预测同样保持精度优势,预测得到的风险评分可以作为所有阶段患者和早期患者的独立预后指标。在包含一千多名患者数据的外部测试集 MCO-CRC 中,生存预后模型保持稳定的优势,具有泛化能力。
乳腺癌与胰腺癌先行:借力 AI 改善预后
在癌症的诊疗过程中,癌症预后分析可以有效避免过度治疗及医疗资源的浪费,为医务人员及家属进行医疗决策提供科学依据,已成为近年癌症研究的热门方向。
为了改善乳腺癌预后,2020 年,Salesforce 公司研究人员与南加州大学 Lawrence J. Ellison 研究所的临床医生合作,推出了机器学习系统 ReceptorNet,其算法可以通过低成本且易获取的组织图像来预测激素受体的状态——这是临床医生在为乳腺癌患者决定合适的治疗路径时的一个重要的生物标记。该系统的准确率达到了 92%。

2024 年 2 月,来自美国肯塔基大学、澳门科技大学、澳门大学、广州医科大学第一附属医院的研究人员采用神经网络模型,建立了一个精准的预后评分系统——MIRS (metastasis and immunogenomic risk score),用于肿瘤转移与免疫基因组风险评分,提供了一项几乎普遍适用于乳腺癌患者的预测工具,为乳腺癌人群的治疗选择提供了新方向。(点击查看详细报道:剑指全球第一大癌症,中国学者建立乳腺癌预后评分系统 MIRS)
此外,胰腺癌作为消化道常见的恶性肿瘤之一,确诊后的五年生存率不超过 10%。提高患者生存率的一个关键环节是准确预测患者的预后风险,以便设计针对性的治疗方案。组织病理是肿瘤科的常规检查,可在微观层面解析肿瘤特性,是评估肿瘤进展风险的重要方法。但由于切片尺寸极大、组织成分复杂,评估结果容易受主观因素影响。

2023 年,来自南京信息工程大学、人工智能学院智慧医疗研究院的研究团队,发布了题为「基于多任务和注意力的胰腺癌全切片图像多组织分割模型」 的研究论文,研究了胰腺癌病理切片 8 种类别的组织分割,通过引入注意力机制并设计分层共享的多任务结构,利用相关辅助任务显著提升模型性能。
该研究提出的模型在上海长海医院的数据集上进行训练与测试,并在 TCGA 公开数据集上进行外部验证,在内部测试集上 F1 分数均高于 0.97,在外部验证集上 F1 分数均高于 0.92,且泛化性能显著优于基线方法。
值得强调的是,AI 并不能替代病理学专家,而是作为一种辅助诊断技术,为病理诊断带来更多便利,进一步提高病理医师工作效率。从长远趋势来看,AI 在数字化生物标志物检测、医疗图像分析、病程预测等方面,还有较大的发展空间。
参考资料:
1.https://news.un.org/zh/story/2018/09/1017602
2.https://mp.weixin.qq.com/s/VE68FKL6kwpO1IFsbR-LVA
3.https://ins.sjtu.edu.cn/articles/286
4.https://www.cdstm.cn/theme/khsj/khzx/khcb/202012/t20201214_1039028.html