在 R 编程语言中,使用 table() 函数可以创建列联表(contingency table),也称为频数表或交叉表。列联表用于显示两个或多个分类变量之间的关系,它显示了每个组合的计数(频数)。在列联表中,行代表一个变量的水平(类别),列代表另一个变量的水平(类别),交叉点的值表示两个变量对应水平的组合出现的次数。
我们做单细胞转录组数据分析的时候尤其是喜欢使用这个函数,比如我们的多个样品整合后细分到亚群,然后在R的gplots包的balloonplot函数对table后的列联表的可视化效果如下所示:
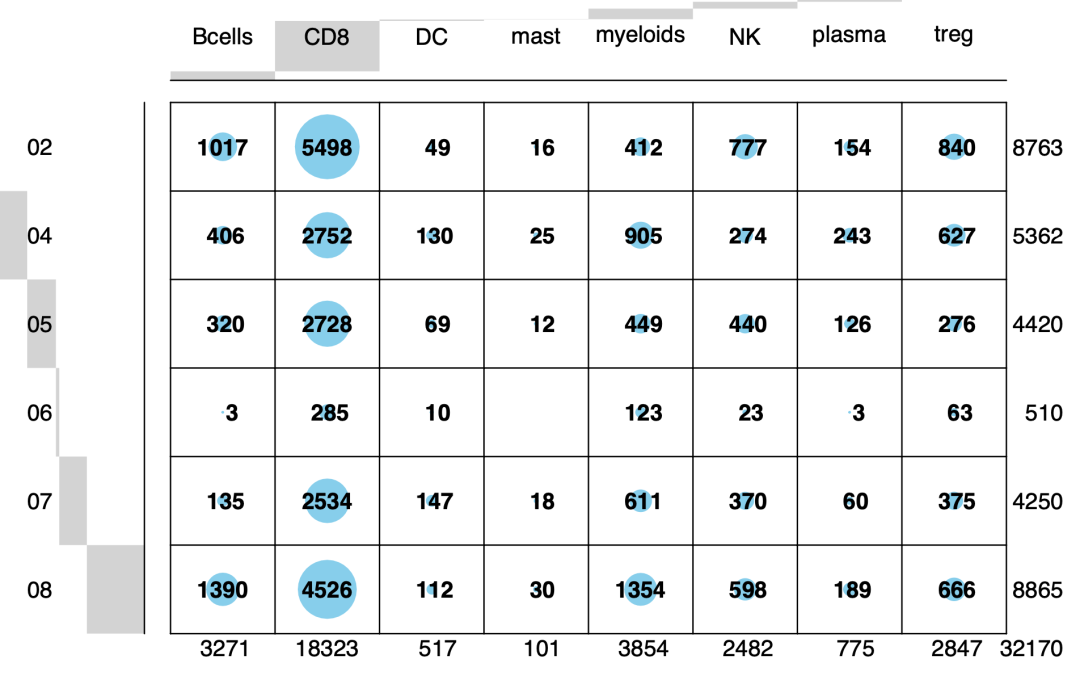
R的gplots包的balloonplot函数对table后的列联表的可视化效果从上面的列联表可以看到06的这个样品其实是有点惨淡,它整体就细胞数量偏少。当然了,上面的是绝对数量的展现,也可以展现细胞比例,那个是后话。目前学员们感兴趣的如何在Python编程语言里面实现这个过程,首先是需要把R里面的数据导出来:
load('phe.Rdata')
colnames(phe)
write.csv(phe[,c(1,16)],file = 'phe.csv')
gplots::balloonplot(table(phe$celltype,phe$orig.ident))
然后在Python里面,使用代码读取上面的 phe.csv文件后,进行统计可视化:
import pandas as pd
import seaborn as sns
import matplotlib.pyplot as plt
import pandas as pd
# 读取 CSV 文件
df = pd.read_csv('phe.csv' )
# 打印前几行数据
print(df.head())
df = pd.DataFrame(df)
# 使用 Seaborn 的heatmap绘制交叉表
cross_tab = pd.crosstab(df['celltype'], df['orig.ident'])
sns.heatmap(cross_tab, annot=True, cmap="YlGnBu", fmt='g')
plt.xlabel('Orig Ident')
plt.ylabel('Cell Type')
plt.title('Cross-tabulation of Cell Type and Orig Ident')
plt.show()
可以看到,效果如下所示:
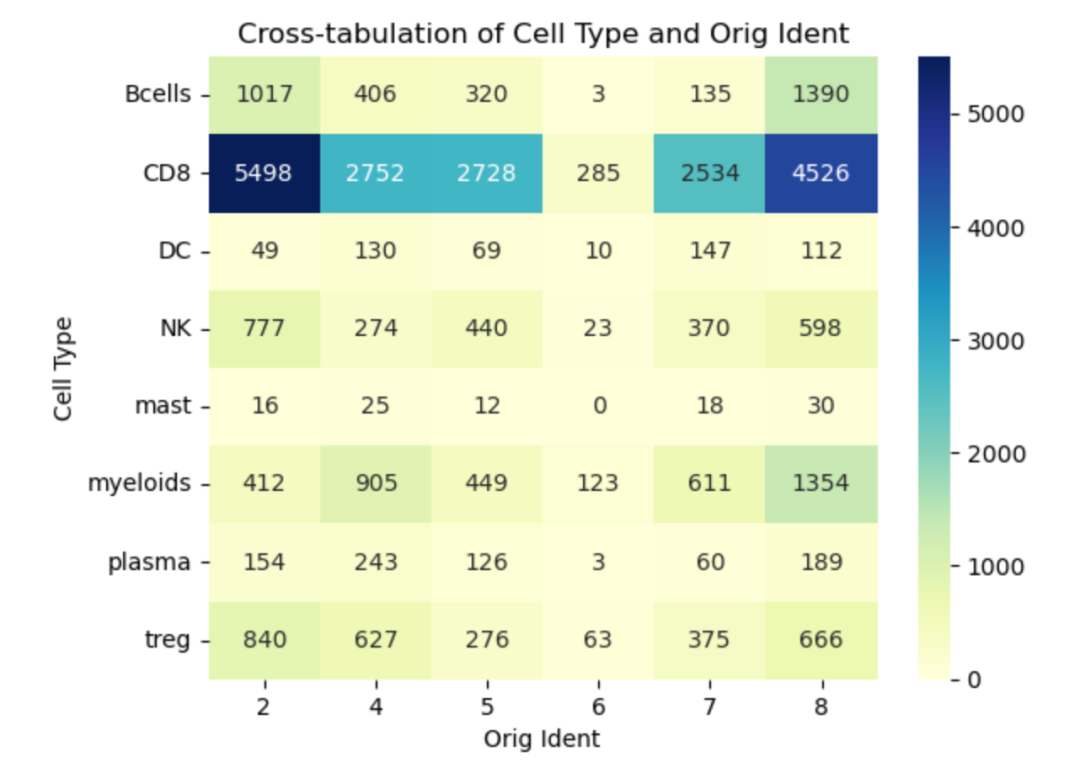
Python的列联表文末友情宣传
强烈建议你推荐给身边的博士后以及年轻生物学PI,多一点数据认知,让他们的科研上一个台阶: