
MIT James J. Collins等研究人员开发新策略,解释基于图神经网络(graph
neural networks, GNN)的深度学习模型对潜在抗生素小分子的预测,挖掘能解释预测结果的分子 “亚结构(substructures)” ;从而,不是简单筛选孤立的的潜在抗生素小分子,而是挖掘新结构类型抗生素[1], [2]。
该策略的关键是:1. 使用更 “理解” 原子关联的图神经网络架构;2. 大量(39,312)且多样的小分子训练数据集;3. 图形搜索算法(graph search algorithms)解析潜在支撑其活性的 “亚结构”[2]。
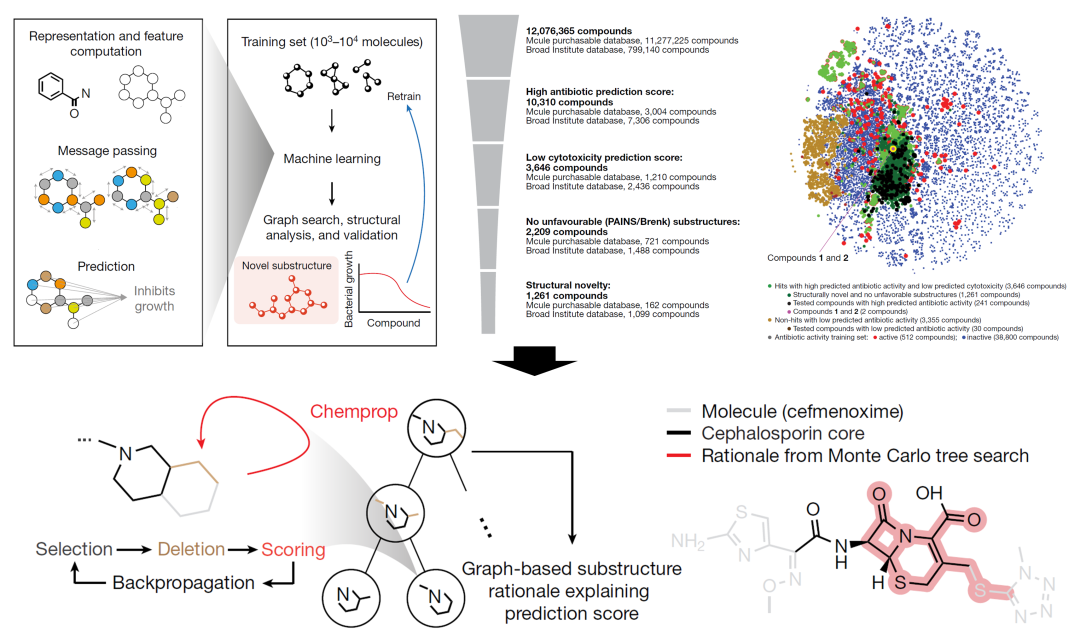 可解释的深度学习模型帮助发现新结构类型的抗生素[2]。
可解释的深度学习模型帮助发现新结构类型的抗生素[2]。
研究人员进一步分析该方法筛选的新类型抗生素潜在工作机制,以及在体(小鼠)对耐药菌的效果[2]。
该项工作2023年12月21日发表在nature[2]。
Comment(s):
该策略或许还能用于筛选抗癌药;毕竟,根据文中数据,不同类型细胞对小分子的毒性反应差异比较大[2]。
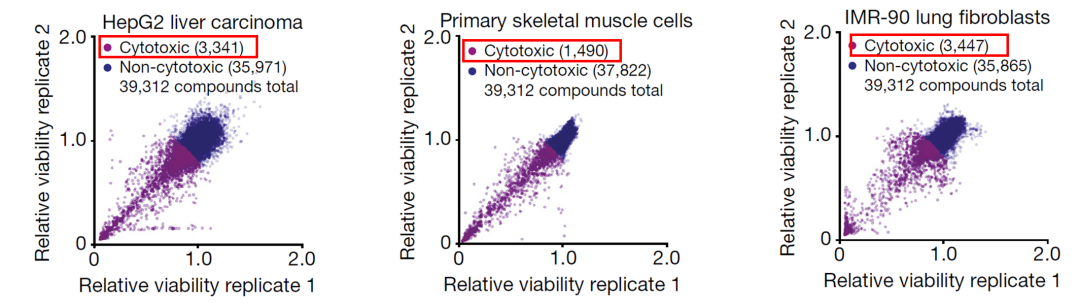 不同类型细胞对小分子的毒性反应有较大差异[2]。
不同类型细胞对小分子的毒性反应有较大差异[2]。
 https://scholar.google.com/citations?user=te5DjPQAAAAJ&hl=en[1] J. M. Stokes et al.,
“A Deep Learning Approach to Antibiotic Discovery,” Cell, vol. 180, no.
4, pp. 688-702.e13, 2020, doi: 10.1016/j.cell.2020.01.021.[2] F. Wong et al., “Discovery
of a structural class of antibiotics with explainable deep learning,” Nature,
no. January 2022, Dec. 2023, doi: 10.1038/s41586-023-06887-8.https://www.nature.com/articles/s41586-023-06887-8
https://scholar.google.com/citations?user=te5DjPQAAAAJ&hl=en[1] J. M. Stokes et al.,
“A Deep Learning Approach to Antibiotic Discovery,” Cell, vol. 180, no.
4, pp. 688-702.e13, 2020, doi: 10.1016/j.cell.2020.01.021.[2] F. Wong et al., “Discovery
of a structural class of antibiotics with explainable deep learning,” Nature,
no. January 2022, Dec. 2023, doi: 10.1038/s41586-023-06887-8.https://www.nature.com/articles/s41586-023-06887-8