将 ScienceAI 设为星标
第一时间掌握
新鲜的 AI for Science 资讯

空间转录组学 (ST) 技术可检测单个细胞中的 mRNA 表达,同时保留其二维 (2D) 空间坐标,使研究人员能够研究转录组在组织中的空间分布;然而,对多个 ST 切片进行联合分析并将它们对齐以构建组织的三维 (3D) 堆栈仍然是一个挑战。
近日,来自中国科学技术大学、合肥综合性国家科学中心和北京生命科学研究所(NIBS)的研究团队,提出了用于 ST 数据分析的深度学习空间架构表征 (SPACEL)。SPACEL 包含三个模块——Spoint、Splane 和 Scube——涵盖了 ST 数据的三个分析任务。
使用来自各种组织的模拟和真实 ST 数据集和 ST 技术与 19 种最先进的方法进行比较,表明 SPACEL 在细胞类型反卷积、空间域识别和 3D 对齐方面优于其他方法,从而展示了 SPACEL 作为 ST 数据处理和分析的有价值的集成工具包。
该研究以《SPACEL: deep learning-based characterization of spatial transcriptome architectures》为题,于 2023 年 11 月 22 日发布在《Nature Communications》上。

空间转录组学 (ST) 技术使研究人员原则上能够检测组织学切片中整个转录组的空间分布,从而大大提高了我们对器官结构和疾病微环境的理解。实验 ST 技术有两大类,可以 i) 以单细胞分辨率检测部分转录组的表达,或 ii) 在没有单细胞分辨率的情况下检测整个转录组。
目前 ST 技术快速发展和应用的趋势表明,在可预见的未来,科学和医学研究中将产生大量来自不同组织(和条件)的 ST 切片。因此,迫切需要能够快速有效地实现多个 ST 切片数据的集成分析的计算工具。
准确识别跨多个切片的功能空间域并重建组织的 3D 结构,为各种实际应用中的重大生物学发现提供了宝贵的机会。总体而言,对于具有重大缺陷的 ST 数据集来说,构建组织的堆叠 3D 对齐仍然是一个巨大的挑战。
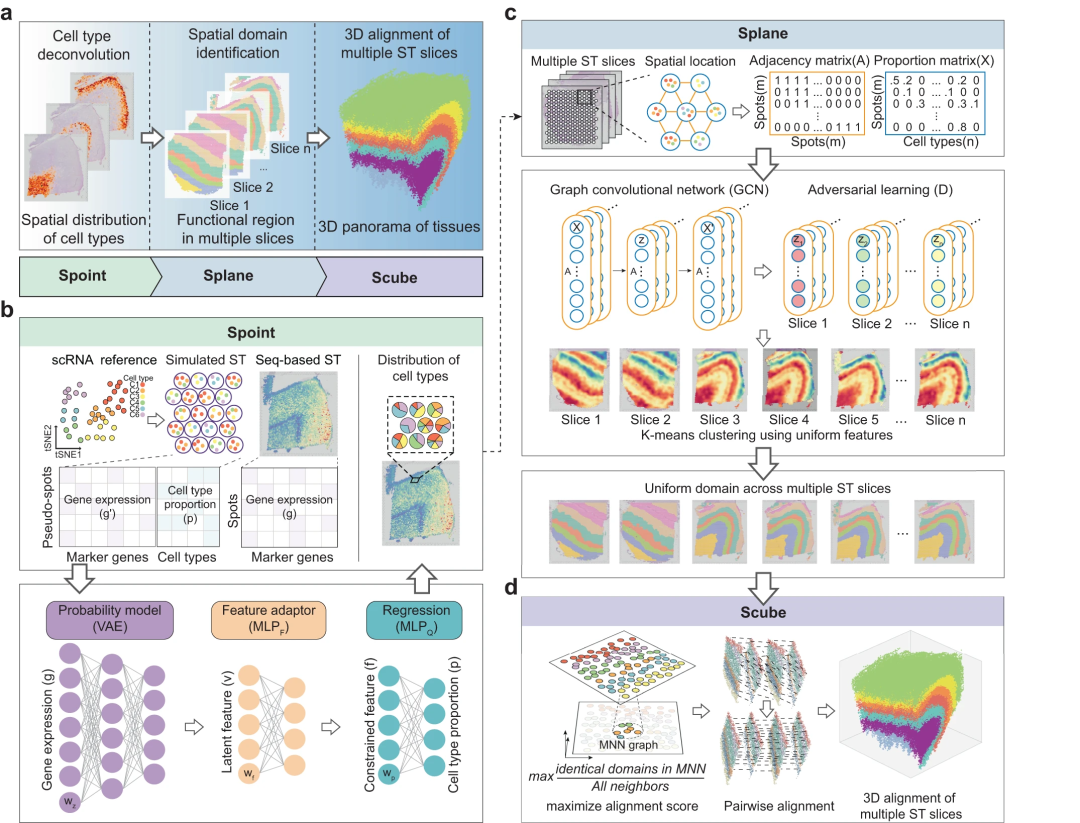
在此,研究人员开发了 SPACEL,一个基于深度学习的工具包。SPACEL 包含三个模块:Spoint 嵌入了带有概率模型的多层感知器,用于对单个 ST 切片中每个点的细胞类型组成进行解卷积;Splane 采用图卷积网络方法和对抗性学习算法来识别跨多个 ST 切片在转录组和空间上一致的空间域;Scube 自动转换连续切片的空间坐标系并将它们堆叠在一起以构建组织的 3D 结构。
为了确保 SPACEL 的稳健性,研究人员进行了广泛的实验来评估其在各种超参数设置下的性能。结果表明,与其他最先进的方法相比,Spoint、Splane 和 Scube 对超参数变化表现出卓越的稳健性,强调 SPACEL 在不同实验设置中提供可靠且一致的结果的有效性。
研究人员应用 SPACEL 分析了 11 个 ST 数据集,其中包括使用 10X Visium、STARmap、MERFISH、Stereo-seq 和 Spatial Transcriptomics 技术获取的 156 个切片。SPACEL 在三个检查的分析任务中均优于其他最先进的方法,因此代表了用于 ST 数据处理和分析的有价值的集成工具包。
在 11 种反卷积方法中,Spoint 产生了最高的平均 PCC/SSIM 值(= 0.73/0.69),以及最低的平均 RMSE/JSD 值(=0.05/0.41)。此外,应用基准研究中定义的准确度得分(AS)来评估每种方法的性能:Spoint 的平均 AS( = 0.93)明显高于其他方法(AS = 0.24-0.82)。
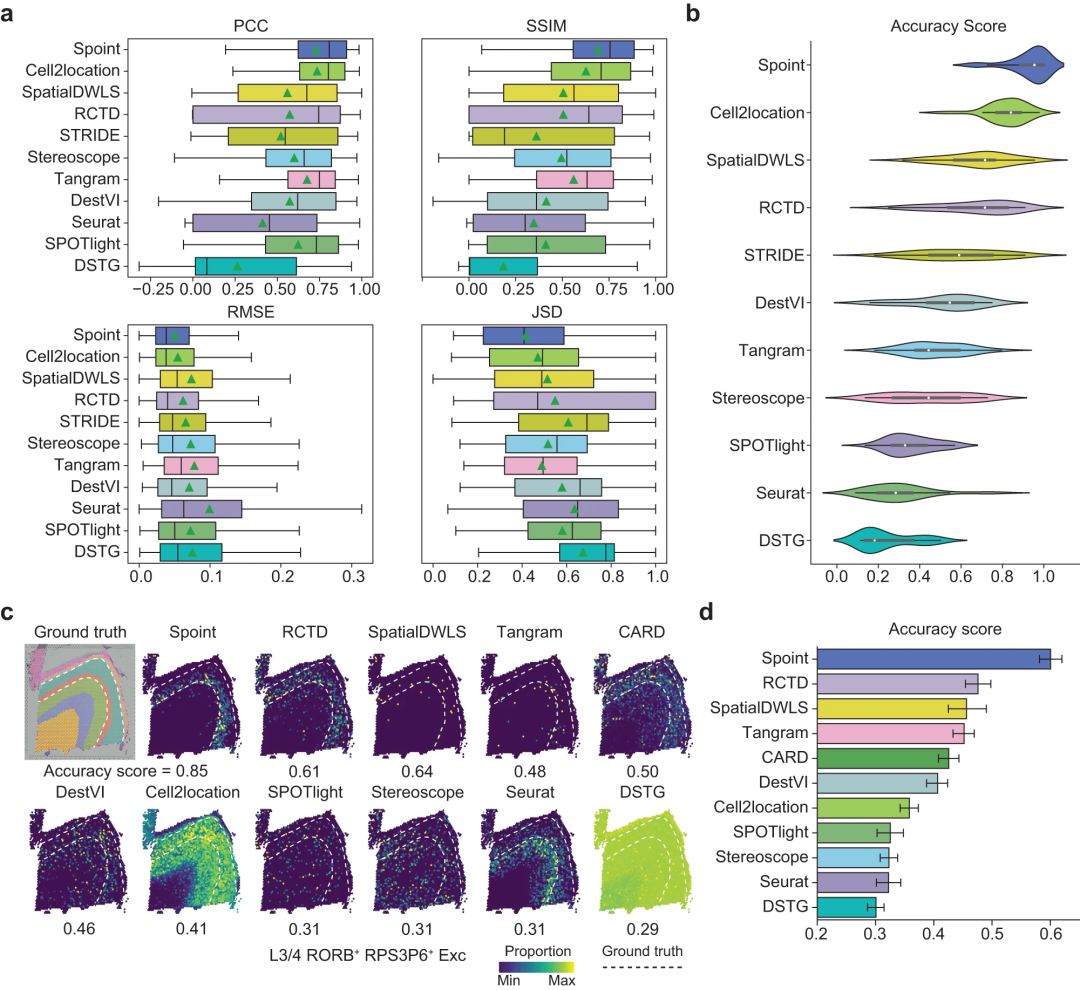
图 2:使用 Spoint 和其他反卷积方法对点进行细胞类型反卷积。(来源:论文)在使用 Spoint 对细胞类型进行解卷积后,应用 Splane 来识别上述 DLPFC 数据集 ST 切片的空间域。研究表明联合分析方案提高了 Splane 空间识别的精度。
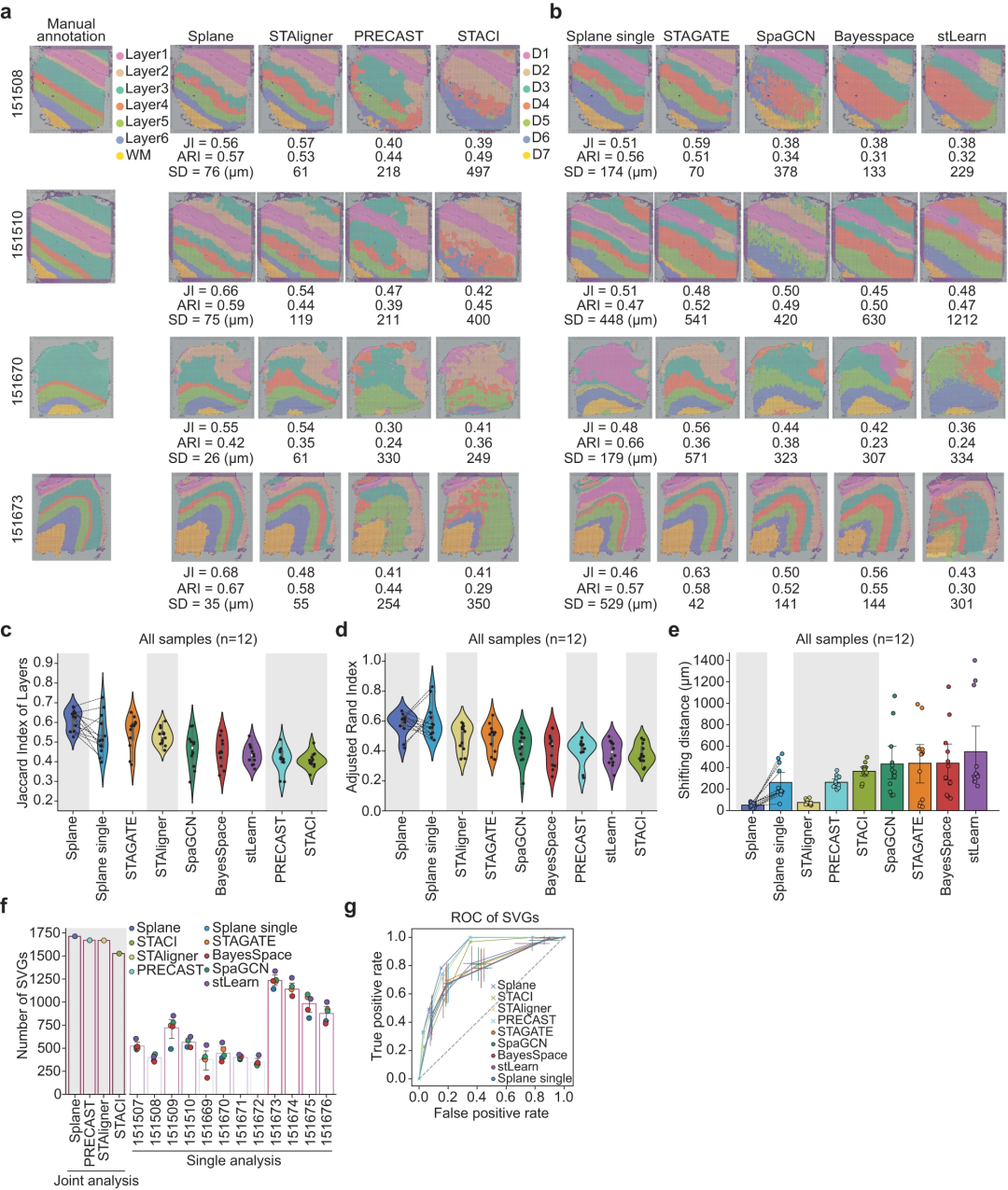
图 3:从 DLPFC 的 12 个 10X Visium 切片中识别空间域。(来源:论文)紧接着测试了 Splane 在识别疾病切片空间域方面的性能。研究结果证明了 Splane 在肿瘤系统的多个切片中识别一致的肿瘤区域和边界的能力。
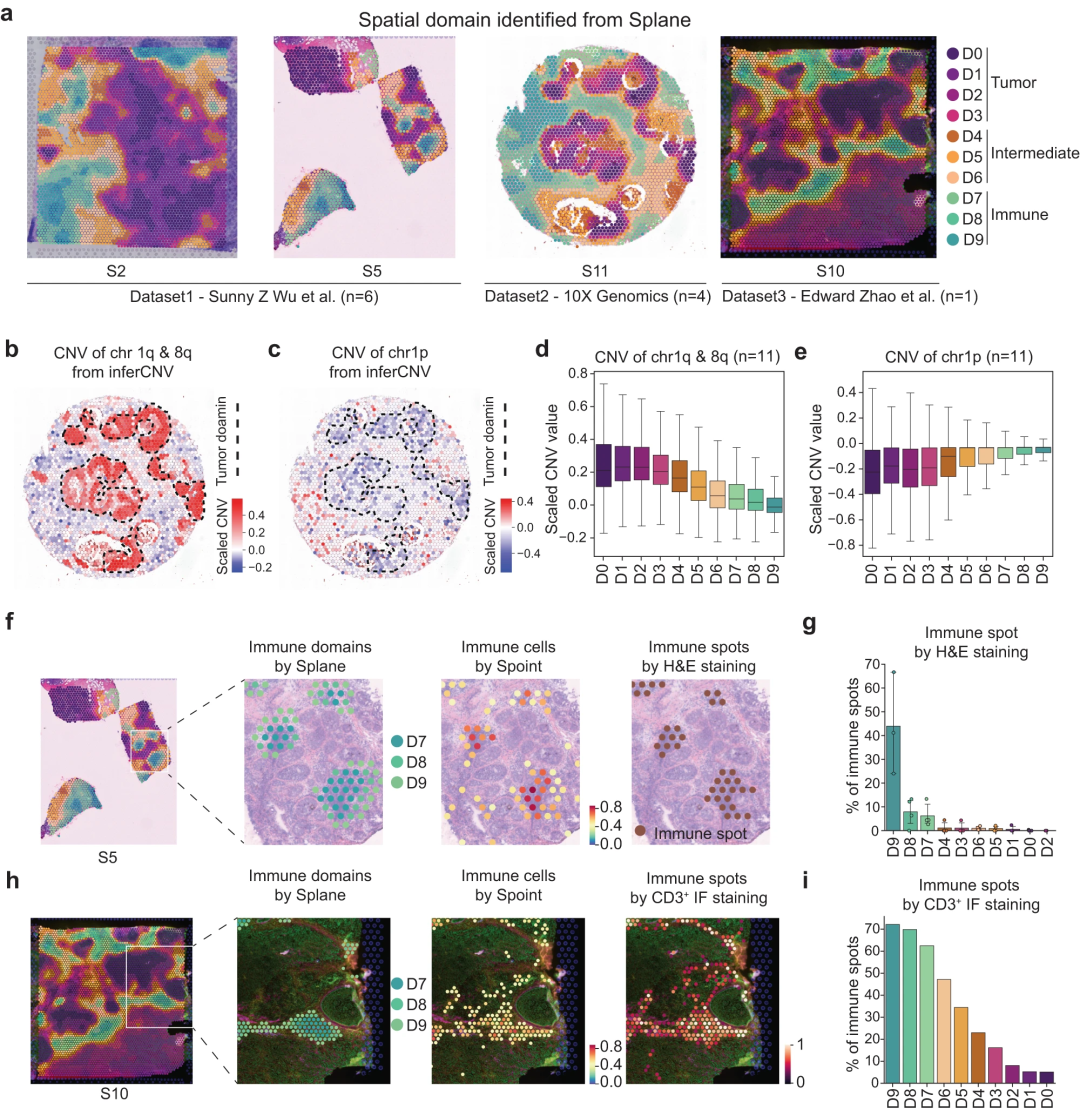
图 4:来自三个不同数据集的 11 个乳腺癌 ST 切片的联合分析。(来源:论文)研究人员还开发了 SPACEL 的 Scube 模块来构建和研究给定组织的 3D 架构。SPACEL 的 Scube 模块在模拟(STARmap)和真实(MERFISH 和 Stereo-seq)数据集的对齐和 3D 架构构建方面均优于 STAligner 和 PASTE。
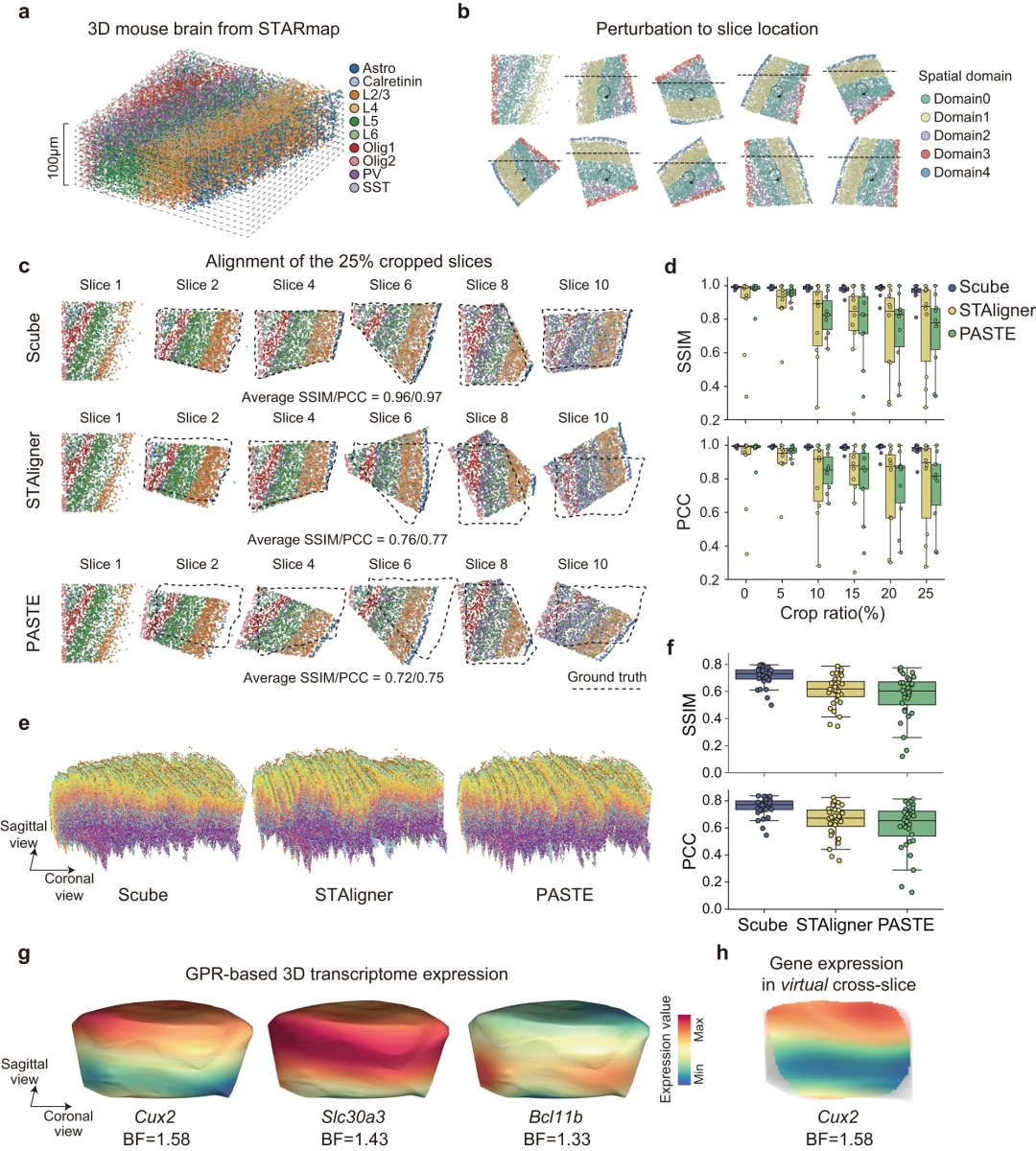
图 5:小鼠大脑连续 ST 切片的 3D 对齐。(来源:论文)为了突出 SPACEL 的集成性质,研究人员应用完全集成的工作流程来分析小鼠全脑 ST 数据。研究表明,SPACEL 可作为分析多个 ST 切片的有效集成工具包。
论文链接:https://www.nature.com/articles/s41467-023-43220-3
人工智能 × [ 生物 神经科学 数学 物理 化学 材料 ]
「ScienceAI」关注人工智能与其他前沿技术及基础科学的交叉研究与融合发展。
欢迎关
注标星,并点击右下角点赞和在看。
点击阅读原文,加入专业从业者社区,以获得更多交流合作机会及服务。