单分子定位显微镜Single-molecule localization microscopy (SMLM) 彻底改变了生物成像,将传统显微镜的空间分辨率提高了一个数量级。然而,单分子定位显微镜SMLM技术需要长的采集时间,通常为几分钟,以产生单个超分辨图像,因其依赖于数千个记录帧上的许多定位累积。因此,在高时间分辨率下,单分子定位显微镜SMLM观测动态的能力,一直受到限制。近日,以色列理工学院(Technion-Israel Institute of Technology) Alon Saguy,Yoav Shechtman等,在Nature Methods上发文,提出了一种基于深度学习的超时空分辨率重建方法DBlink。DBlink的输入是单分子定位显微镜SMLM数据的记录视频,输出是超时空分辨率视频重建。
利用用卷积神经网络与双向长短期记忆网络架构相结合,旨在捕捉不同输入帧之间的长期相关性。DBlink性能验证在模拟细丝和线粒体样结构、在受控运动条件时实验单分子定位显微镜SMLM数据上,以及在活细胞动态SMLM。DBlink时空插值构成了活细胞动态过程超分辨率成像的重要进展之一。
 DBlink: dynamic localization microscopy in super spatiotemporal resolution via deep learning.
DBlink: dynamic localization microscopy in super spatiotemporal resolution via deep learning.
DBlink:基于深度学习,实现了超时空分辨率的动态定位显微镜。
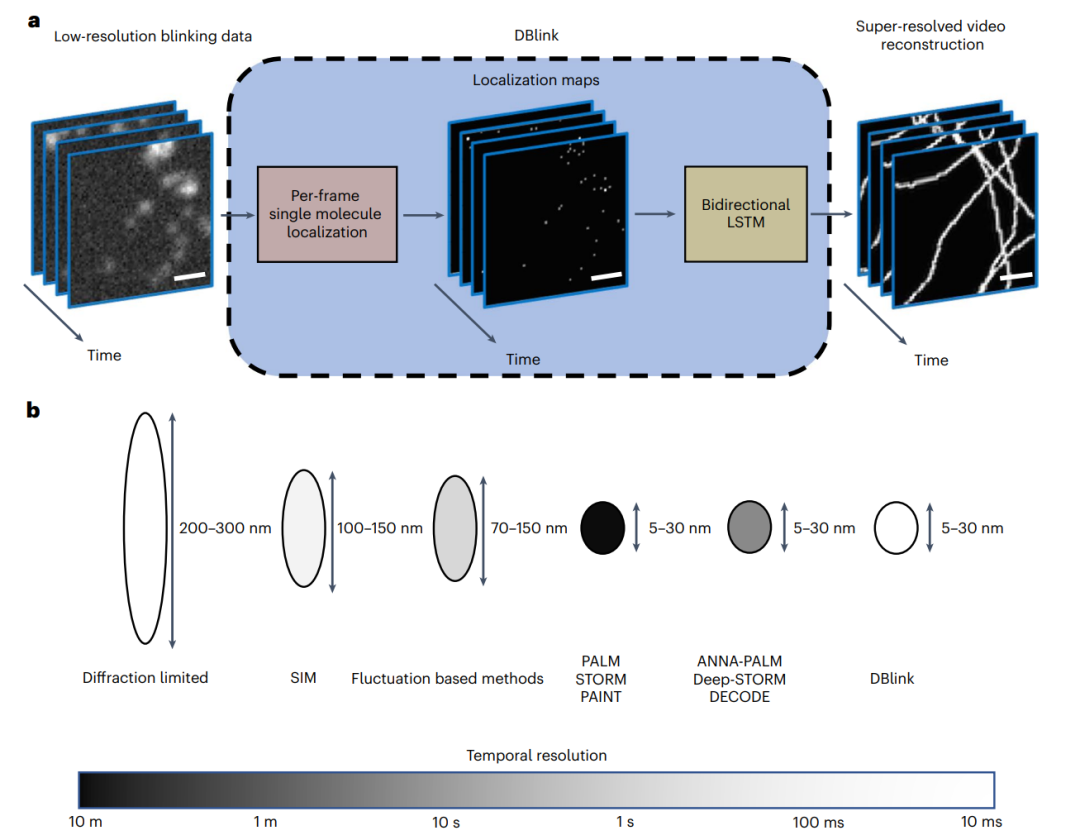 图1:DBlink概念。
图1:DBlink概念。
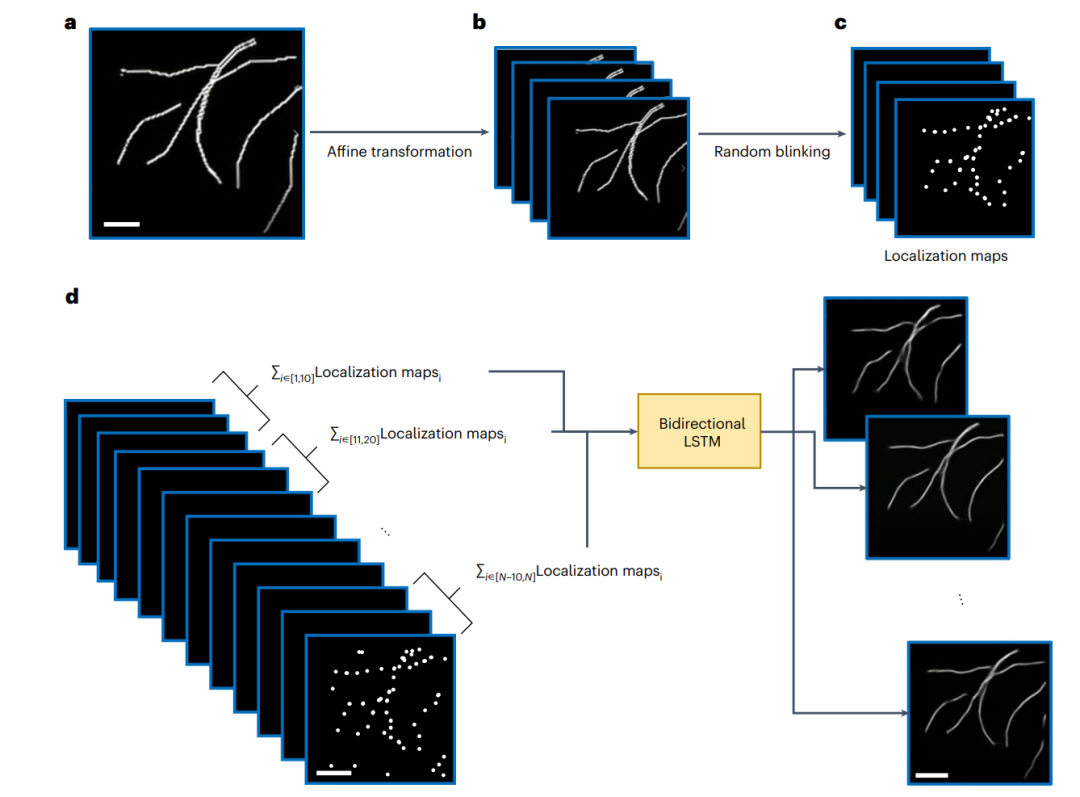 图2:模拟纤维丝数据的生成和分析。
图2:模拟纤维丝数据的生成和分析。
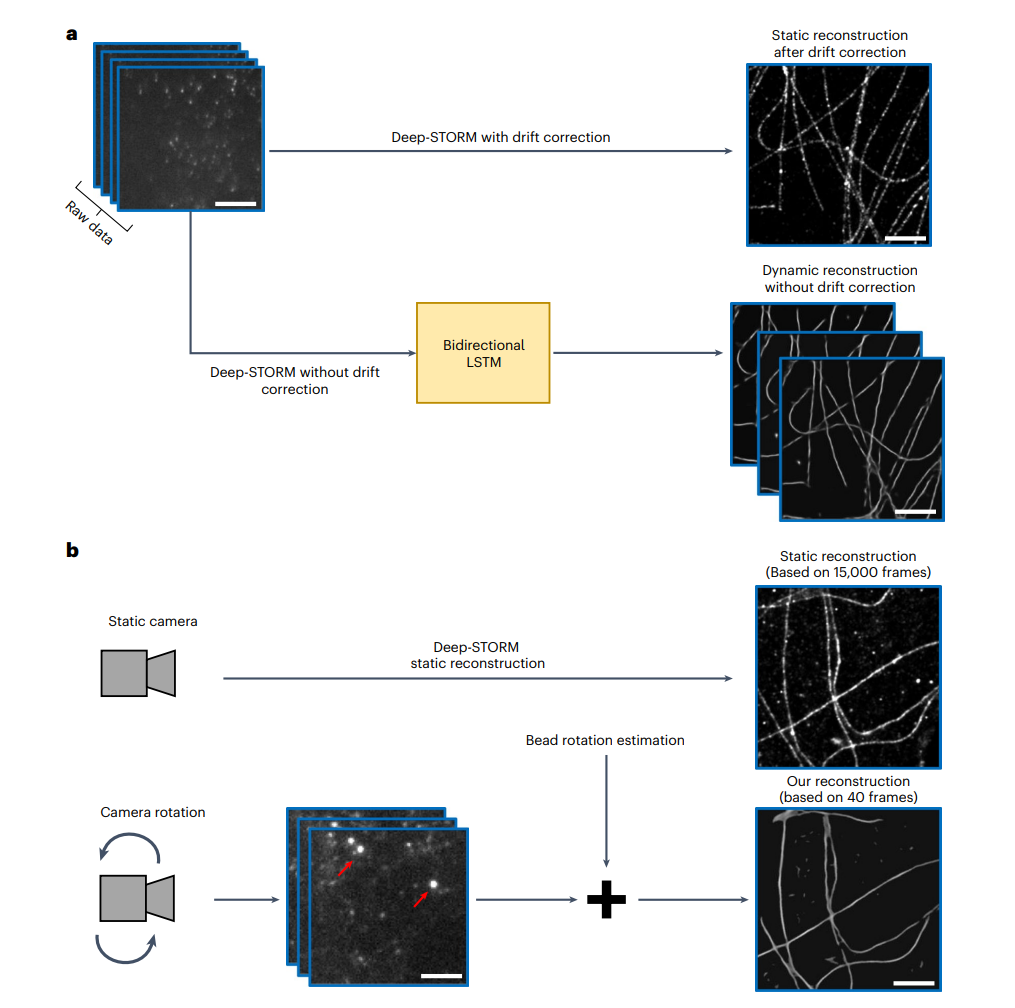 图3:全局运动期间的静态结构重建。
图3:全局运动期间的静态结构重建。
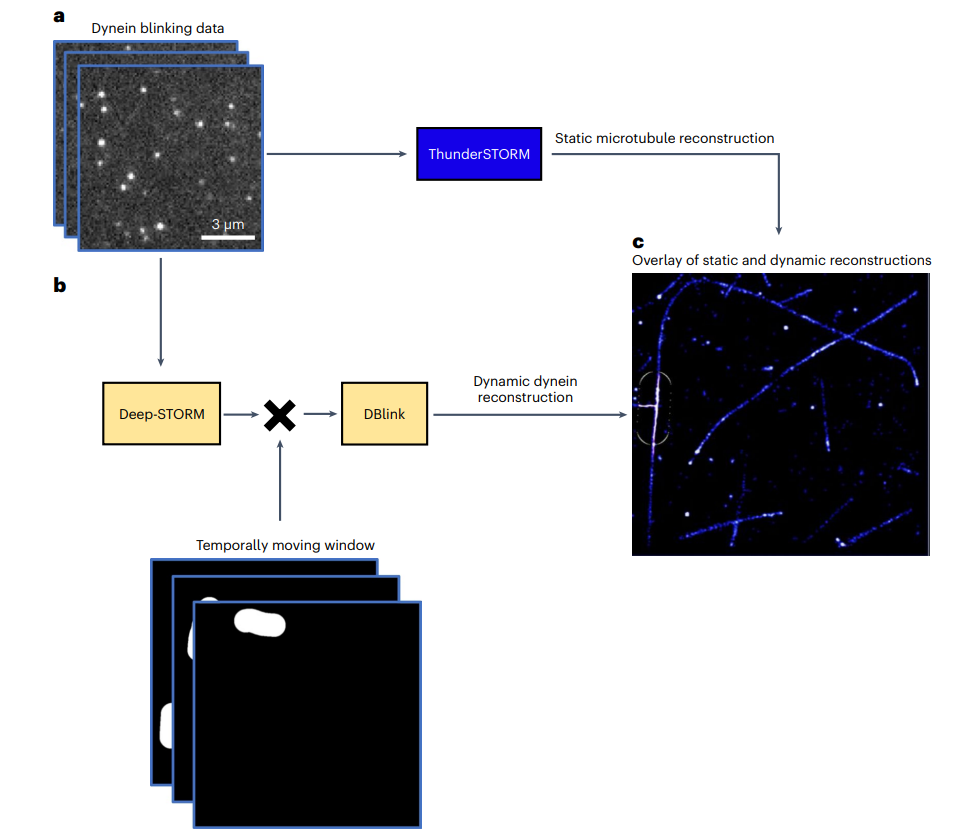 图4:在微管上,跟踪动态移动的动力蛋白马达。
图4:在微管上,跟踪动态移动的动力蛋白马达。
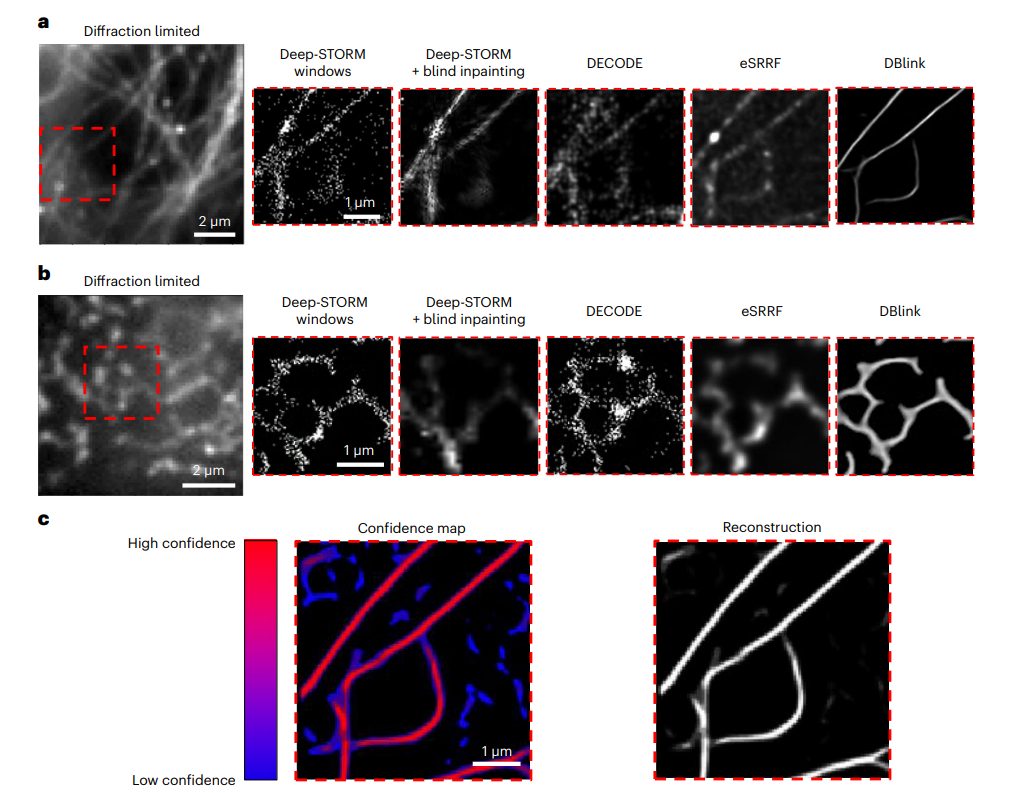 图5:DBlink与其他先进方法的定性比较。
图5:DBlink与其他先进方法的定性比较。
文献链接
Saguy, A., Alalouf, O., Opatovski, N. et al. DBlink: dynamic localization microscopy in super spatiotemporal resolution via deep learning. Nat Methods (2023).
https://doi.org/10.1038/s41592-023-01966-0
https://www.nature.com/articles/s41592-023-01966-0
本文译自Nature。
来源:今日新材料
声明:仅代表译者个人观点,小编水平有限,如有不当之处,请在下方留言指正!