
关键词:蛋白质结构,蛋白质动力学,生成对抗网络,分子动力学,粗粒化
论文题目:Direct generation of protein conformational ensembles via machine learning论文来源:Nature Communications
论文链接:https://www.nature.com/articles/s41467-023-36443-x
动力学和构象取样对于将蛋白质结构与生物功能联系起来至关重要。蛋白质结构在实验上很难探测,因此,计算机模拟被广泛用于描述蛋白质动力学,然而,过高的计算成本持续限制着可研究的系统。
近期发表于 Nature Communications 的这项研究证明机器学习可以用模拟数据进行训练,直接生成物理上真实的蛋白质构象集合,而不需要任何采样,并且计算成本可以忽略不计。作为原理证明,研究中训练了一个基于 transformer 架构的生成对抗网络,该架构具有对本质无序肽的粗粒度模拟的自注意力。
由此产生的模型 idpGAN 可以预测训练集中不存在的序列的依赖于序列的粗粒度集合,表明可以在有限的训练数据之外实现可迁移性。研究还在原子模拟数据上重新训练 idpGAN,以表明该方法在原则上可以扩展到更高分辨率的构象集合生成。
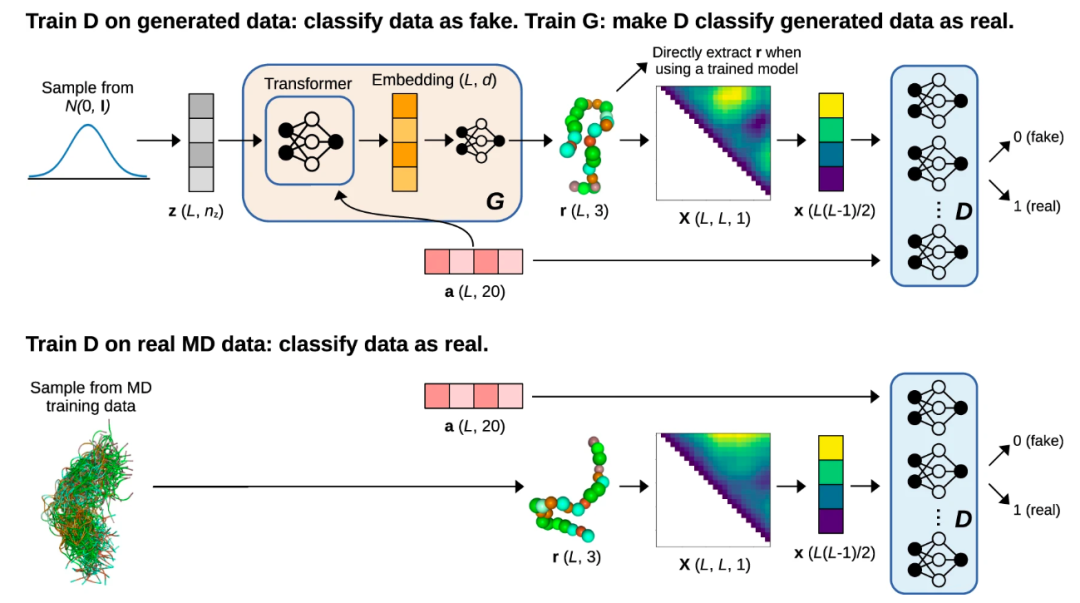
图1. idpGAN 的网络架构
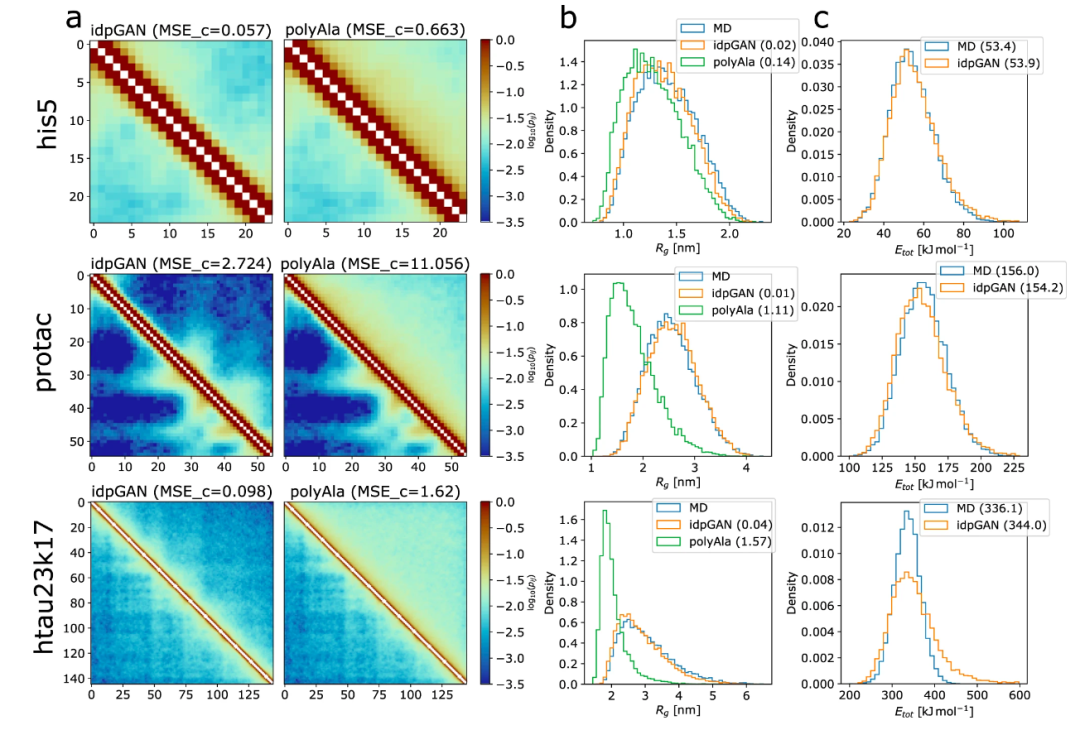
图2. 三种 IDP_test 蛋白质的 idpGAN
集智斑图顶刊论文速递栏目上线以来,持续收录来自Nature、Science等顶刊的最新论文,追踪复杂系统、网络科学、计算社会科学等领域的前沿进展。现在正式推出订阅功能,每周通过微信服务号「我的集智」推送论文信息。扫描下方二维码即可一键订阅:
