
关键词:
代谢系统,系统发生树,基因数据重构,宏观演化,演化压力,机器学习
论文题目:Machine learning enables prediction of metabolic system evolution in bacteria论文地址:https://www.science.org/doi/10.1126/sciadv.adc9130
进化可以预测吗?这是进化生物学的一个长期目标,对战略性病原体控制、基因组工程和合成生物学都具有潜在影响。虽然实验室的演化研究显示短期的和序列水平演化的可预测性,但很少有研究在宏观演化尺度上研究更显著、更长期和更系统水平的演化能否预测。这主要是因为无法直接在实验室里观察到系统层面的演化。然而,丰富的基因组数据、大规模系统发生树、祖先基因内容重构方法和机器学习技术是否可能系统地重构基因内容?
Science Advances 的这项研究通过对约3000个细菌基因组应用祖先基因数据重构和机器学习技术,证明代谢系统的基因内容演化通常是可预测的。论文提出的 Evodictor 框架成功预测了在参考系统发生树分支上,基因获得和丢失的演化,表明代谢系统的演化压力和限制普遍存在。对代谢途径结构的研究和宏基因组数据集的荟萃分析,证实了这些演化模式具有生理和生态学基础,即代谢反应和细菌栖息地变化之间的功能依赖性。最后,种内基因含量变异的泛基因组学分析证明,即使是现存细菌物种的“正在进行的”演化,在这个框架下也是可以预测的。
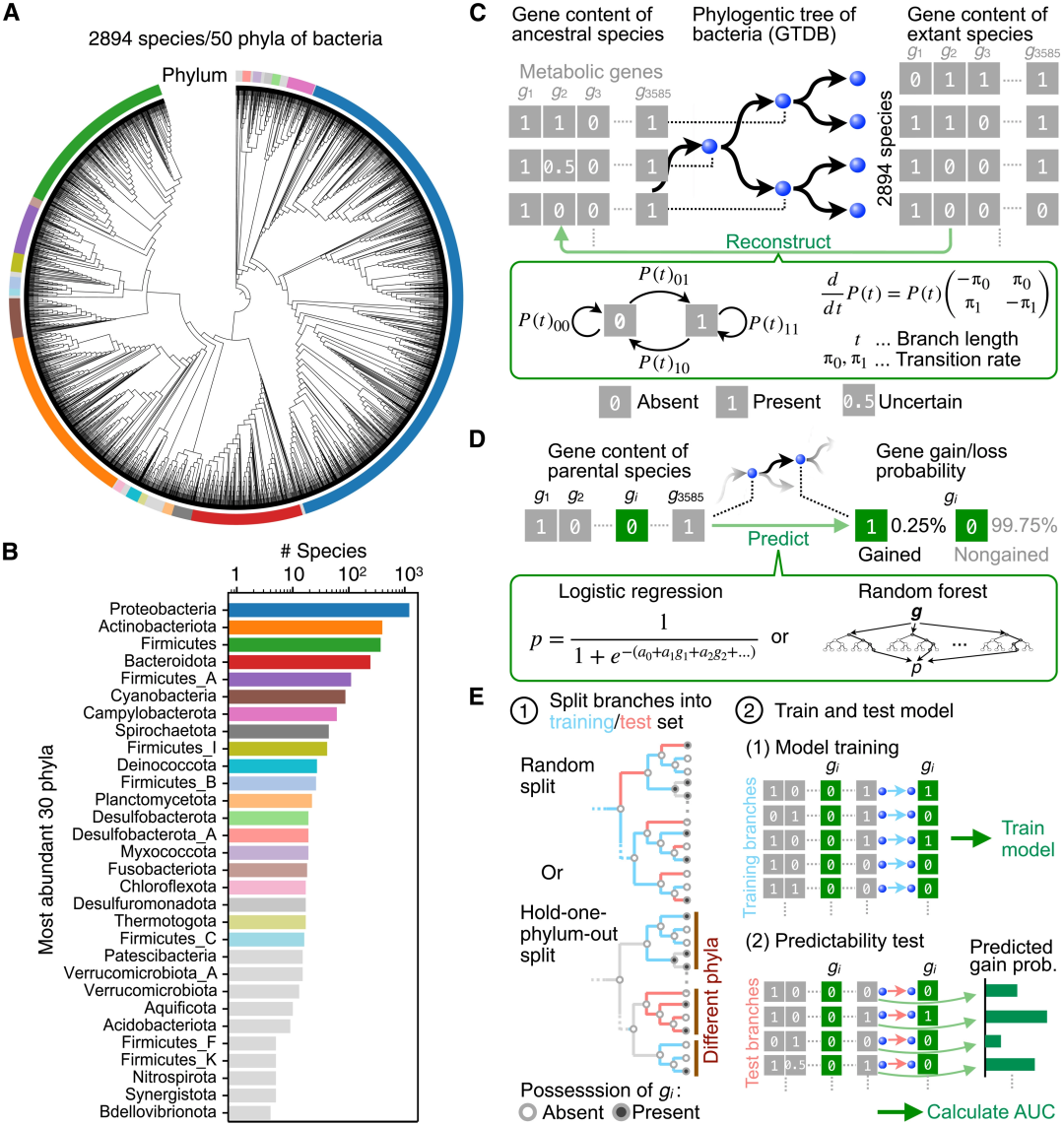
图1. 数据集和分析概述
图2. 通过逻辑回归预测基因获得与丢失
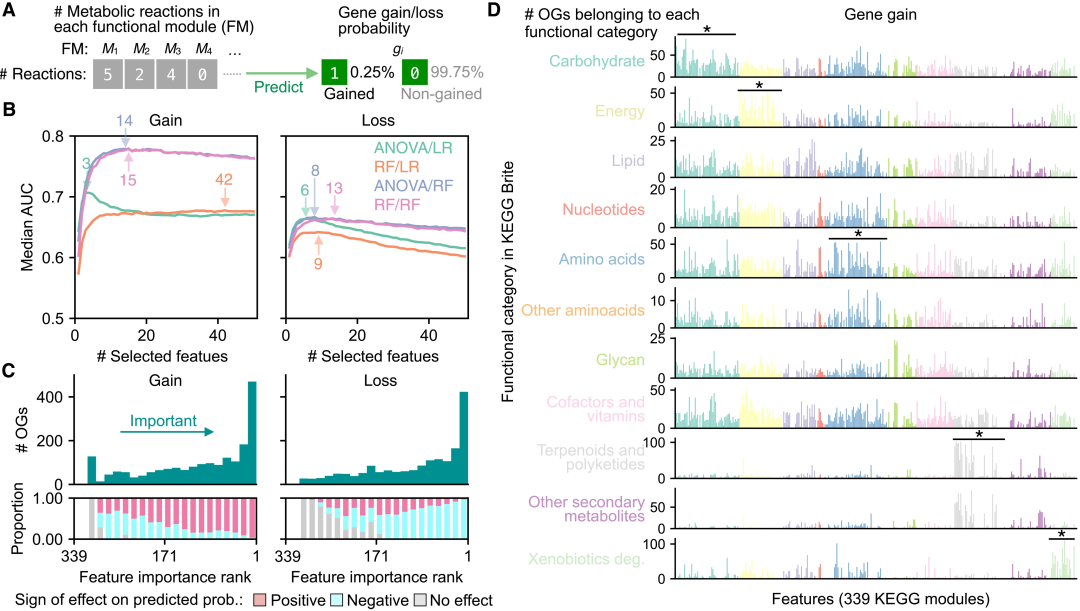
图3. 功能相关基因的存在对于预测基因获得/丢失的重要性。
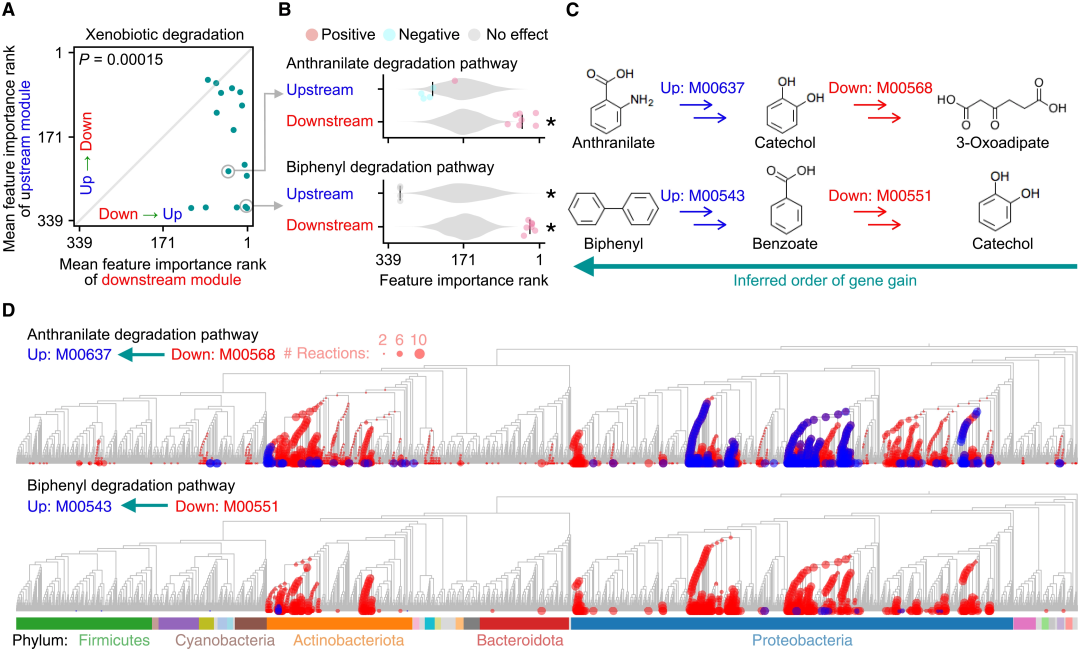
图4. 外源性物质降解途径基因增益顺序的上游至下游模式。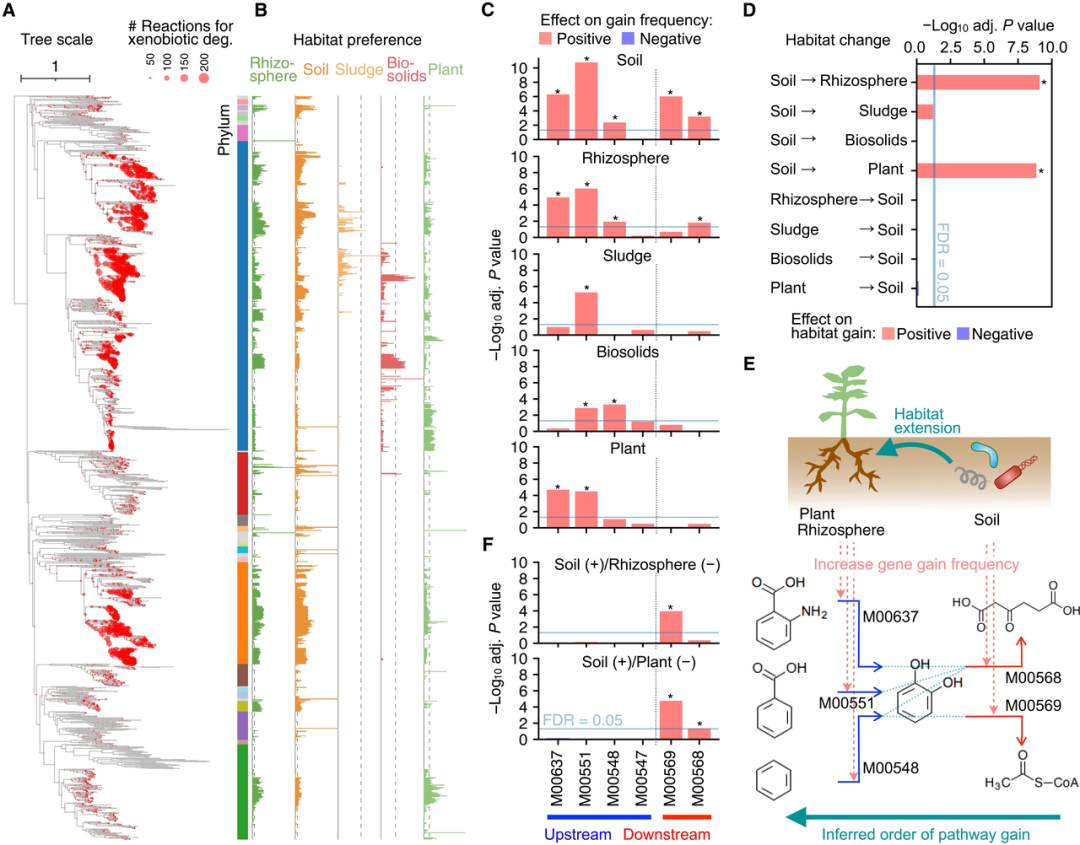
集智斑图顶刊论文速递栏目上线以来,持续收录来自Nature、Science等顶刊的最新论文,追踪复杂系统、网络科学、计算社会科学等领域的前沿进展。现在正式推出订阅功能,每周通过微信服务号「我的集智」推送论文信息。扫描下方二维码即可一键订阅:
