基于结构的虚拟筛选(structure-based virtual screening, SBVS)旨在根据目标靶点的三维结构,从海量的化合物中区分出能够与其产生一定强度相互作用的配体分子。为此,能够捕捉到蛋白质-配体相互作用特征,并将其转化为两者亲合性强弱得分的蛋白-配体相互作用打分函数,在其中起到了关键性的作用。在过去的十余年间,机器学习技术的发展也大大推动了这一类型打分函数的不断突破,在基准测试的各项指标上不断刷新着记录。
然而,现有的评价体系往往高估了这些机器学习打分函数(machine-learning scoring functions, MLSFs)的泛化能力,即在未曾见过的靶点上准确预测出小分子配体的亲合性得分的能力。令人遗憾的是,泛化能力的不足局限了机器学习打分函数在现实世界药物研发中的应用。为此,北京生命科学研究所的黄牛小组改进了此类机器学习模型泛化能力的评价手段,并深入分析了12种代表性打分函数的泛化能力。近日,该项研究工作发表在美国化学会出版的计算化学和化学信息学核心期刊Journal of Chemical Information and Modeling上(J. Chem. Inf. Model. 2022, 62, 22,
5485–5502)【1】。
改进聚类方案:结合口袋Pfam聚类
首先,作者基于蛋白质家族数据库(protein family database, Pfam)提出了结合口袋的Pfam聚类方案(pocket Pfam-based clustering approach)。蛋白质家族数据库对蛋白质序列进行系统聚类,并根据其进化关系、功能与结构等因素形成家族、结构域等水平的条目。作者将PDB中的蛋白质根据Pfam条目进行聚类,若同一蛋白质可被归属于不同的Pfam条目,则以配体结合口袋内更多的相同残基数为依据,指定唯一的口袋Pfam条目进行归属。
相比于以往基于蛋白质序列相似性的聚类方案,根据结合口袋的Pfam聚类能够避免序列相似性低但结合口袋结构相对保守的情况(如图1所示),更加适合于研究蛋白-配体相互作用的场景,尽可能地将结合部位相似的复合物分配到不同的聚类中。
图1:具有较低序列相似性但结合口袋结构保守的典型案例代表性机器学习打分函数整体泛化能力不如人意
在评价的模型方面,作者根据所采用的算法选取了代表性的12种蛋白-配体亲合性打分函数,包括了基于多元线性回归的LR::V、基于随机森林模型的RF-Score和ΔVinaRF20、基于3D-CNN的Pafnucy、基于GCN的IGN等等。
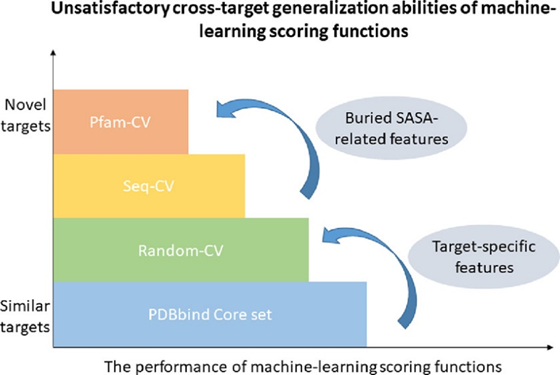
随后,以PDBbind v2020数据集为基础,作者分别采用了四种不同的数据集划分方案,构建了四套平行的训练-测试数据集。第一套方案从PDBbind精选集(refined set)中根据蛋白质序列相似性聚类,挑选出266个复合物作为核心集(core set),并以核心集为测试集,余下数据用于模型的训练和验证。与方案一不同,余下三套方案均采用三折交叉验证策略,将整个PDBbind v2020数据集分成三份,一份用于测试,另两份进一步按照80:20的比例划分训练集和验证集。其中,方案二采用随机交叉验证(Random-CV),数据集的划分完全随机;方案三(Seq-CV)与方案四(Pfam-CV)先分别根据蛋白质序列相似性与配体分子指纹相似性、结合口袋的Pfam条目进行聚类,确保同一聚类的复合物归属于同一份中,再随机划分成三份。
因此,四种数据集划分方案按照区分相似蛋白-配体复合物的能力,从方案一到方案四依次递增,所构建的测试集中靶点的新颖性也逐渐提高。
图2:12种打分函数的测试结果
基于上述的四种数据集划分方案,作者分别对12种打分函数进行了重新训练,并对相应的测试集复合物预测其亲合性,以预测值和实验值之间的Pearson相关系数(Rp)、平均绝对误差(MAE)作为主要的打分能力指标进行评价,结果如图2所示。自左向右,四种评价方案的测试集与训练集间相似性依次降低。作者直观地展示了一个令人遗憾的结果:尽管在core set上那些机器学习打分函数均展现出较为优异的预测表现,但除了两种线性回归的模型没有受到显著影响之外,机器学习打分函数对于蛋白-配体复合物亲合性的预测结果,随着靶点新颖性的提高而出现了明显的降低,即不具备理想的泛化能力。此外,相对更加复杂的深度学习模型降低的幅度也比经典的机器学习算法更加明显。
机器学习打分函数的预测与包埋溶剂可及表面积(buried SASA)明显相关
图3:模型预测打分与包埋溶剂可及表面积的相关性分析
作者观察测试结果后认为,RF-Score相较于其他机器学习模型而言泛化能力尚可,且随机森林模型的可解释性相对较高。因此,作者采用了SHAP方法对RF-Score进行了可解释性分析,发现其中贡献最大的C-C原子对计数一项与包埋的溶剂可及表面积高度相关,Rp可达0.84(如图3A)。进一步地,作者推测这些机器学习打分函数的预测结果均与这一性质有明显的关联。于是,在计算了模型预测得分和仅基于包埋的溶剂可及表面积进行计算的蛋白-配体亲合性之间的相关系数后,作者发现越是在新颖的靶点上,机器学习打分函数的亲合性预测结果就越依赖于该性质(如图3B)。
小结
本文报道了一种新颖而更加严格的机器学习打分函数泛化能力的评估方案,其核心是基于结合口袋的Pfam聚类方法及相应的交叉验证策略。12种代表性的蛋白-配体亲合性打分函数的系统测试,揭示了目前机器学习打分函数的泛化能力整体表现不佳,且模型更倾向于关注蛋白-小分子结合的表面,而因此受到包埋溶剂可及表面积等有关少数性质的显著影响。一方面表明需要采用更加符合真实世界研究条件的打分函数评估方案,来对模型给予更加公正且实用的评价,另一方面也提示了我们广泛用于打分函数训练的PDBbind数据集中,存在数据分布的偏差并会影响到模型的泛化能力。
【1】Hui Zhu, Jincai
Yang*, and Niu Huang*, Assessment of the Generalization Abilities of
Machine-Learning Scoring Functions for Structure-Based Virtual Screening, J.
Chem. Inf. Model. 2022, 62(22), 5485-5502.
http://doi.org/10.1021/acs.jcim.2c01149
感兴趣的读者,可以添加小邦微信(zhiyaobang2020)加入读者实名讨论微信群。添加时请主动注明姓名-企业-职位/岗位 或
姓名-学校-职务/研究方向。
