大家好,今天跟大家分享一篇最近发表在 nature communications 上的文章,文章的题目是“DeepPROTACs is a deep learning-based targeted degradation predictor for PROTACs”,这篇文章利用机器学习,实现了对 PROTACs 小分子靶向降解能力的预测,其平均预测准确性高达 77.95%。本文的三个通讯作者分别是来自于上海科技大学的 Fang Bai 和 Shenghua Gao,来自于上海标新生物公司 Gluetacs Therapeutics 的 Xiaobao Yang。

PROTACs 是一种双功能小分子,它的一头能够与 E3 泛素连接酶结合,另一头可以与靶蛋白结合。它的两头通过一段 linker 连接。当 E3 泛素连接酶-PROTACs-靶蛋白三者形成复合物以后,E3 连接酶给靶蛋白打上泛素化标签,随后,该泛素化的靶蛋白被蛋白酶体所识别和降解。传统意义上的小分子抑制剂大都是归属于占据驱使的药物(比如靶蛋白活性中心或者阻断靶蛋白与上下游蛋白的相互作用从而阻断信号的传递),这种作用模式有一些缺点,比如有一些蛋白不能够靶向、脱靶和耐药等等。随即,单克隆抗体和 RNAi 类的药物作为补充开始进入靶向治疗的舞台。尽管抗体对于细胞外的蛋白具有非常高的结合力,RNAi 可以将细胞内的靶蛋白的含量降到很低的水平,但是二者的缺点严重限制了其治疗前景。前者很难通过细胞膜,而后者不适合口服给药。2001 年,Crews 和其同事开发了 PROteolysis TArgeting Chimeras (PROTACs) 技术,自此为新药开发提供了新的思路。对于 PROTACs 小分子来说,其 linker 对于靶蛋白的降解能力是非常重要的,但是一个 linker 是否有效合适,只能够通过大量的实验试错进行验证。本文希望运用深度学习的技术对不同 linkers 的 PROTACs 的降解能力进行预测,从而助力降低总体的成本和缩短研发周期。本文借助图像卷积神经网络算法,通过借鉴 PROTAC-DB 提供的 PROTACs 的数据,包括 2258 PROTACs, 275 个 “弹头” (能够结合靶蛋白的小分子), 68 个 E3 配体 (能够招募 E3 连接酶的小分子) 和 1099 个 linkers。他们也汇总了其它已发表的 375 个 PROTACs。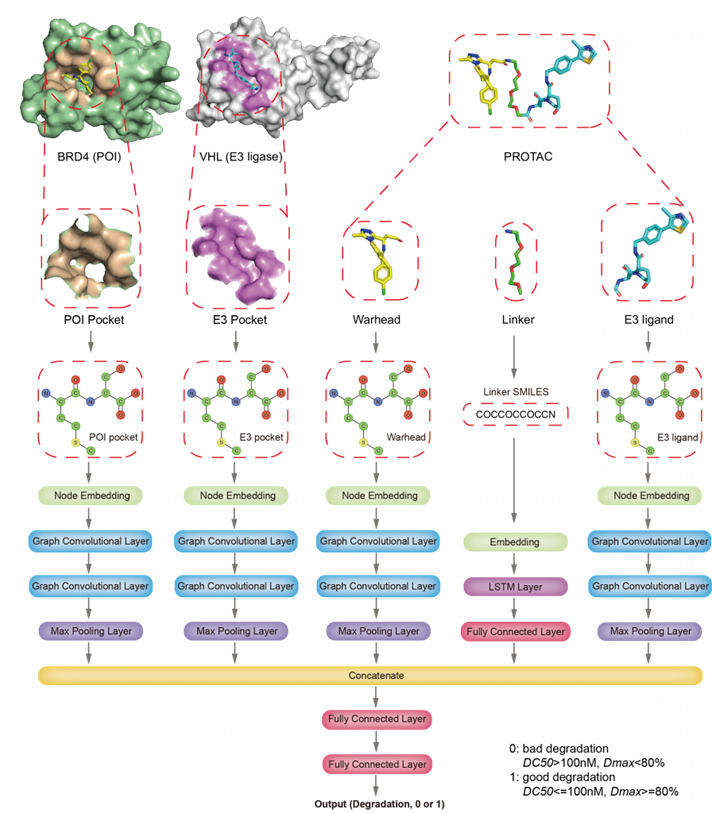
理性地设计出该预测器需要考虑以下 5 个变量:1)靶蛋白上与 PROTACs 小分子结合口袋的周围氨基酸的分布;2)E3 连接酶 PROTACs 小分子结合口袋的周围氨基酸的分布;3)PROTACs 小分子的 warhead;4)linker;5)PROTACs 小分子的 E3 连接酶配体。作者们对此五部分都单独通过神经网络算法训练出了各自的模型,最后串联起来,结果以 0 或者 1 的形式输出,分别表示可能是是一个不好的 PROTACs 或者是 degradation concentration DC50 不大于 100 nM 且降解能力不小于 80% 的,一个好的 PROTACs。最后,为了验证他们预测器的可靠性,他们选择了常见的雌激素受体 estrogen receptor ER 为靶蛋白,E3 连接酶选择了 VHL 配体,一共设计了 16 个 linkers,10 个长度不同的烷基链的 linkers,6 个长度不同的 PEG 链的 linkers。在两种细胞系中进行了实验上的验证,成功预测出了 11 个 PROTACs 的降解能力,预测的准确性为 68.75%。作者们也尝试了其它的 PROTACs,DeepPROTAC 整体预测的准确性在 60% 到 80% 之间。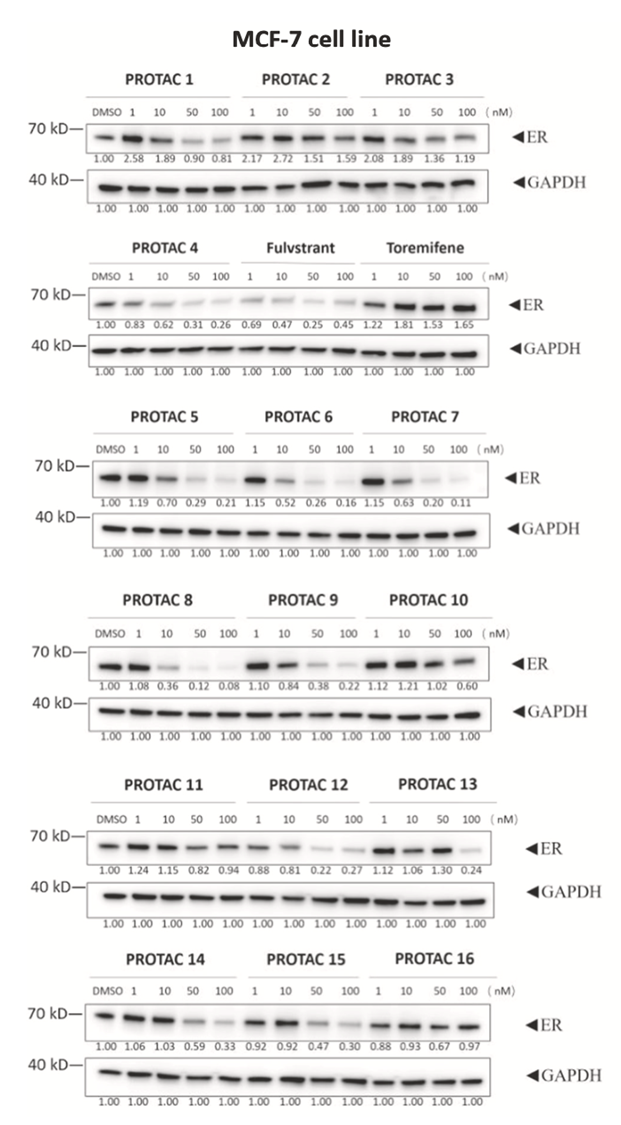
作者也为 DeepPROTACs 开发了一个网址(https://bailab.siais.shanghaitech.edu.cn/services/deepprotacs),同时开源了其代码(https://github.com/fenglei104/DeepPROTACs)。总之,作者们首次尝试用深度学习技术助力 PROTACs 小分子的开发和设计,在正式投入实验验证之前用 DeepPROTACs 提前进行预测,也许能为我们设计出更高效的 PROTACs 提供更多的可能性。本文作者:Zihua Liu
责任编辑:JGG
原文链接:https://www.nature.com/articles/s41467-022-34807-3
文章引用:DOI:10.1038/s41467-022-34807-3