随着测序技术的进步与发展,生物医学领域获得的组学数据正呈爆发式增长。不同的研究项目和研究手段,产生了以基因组学、转录组学、蛋白质组学和代谢组学为大类的多种组学数据。但是单独一种组学的数据只是从单一角度解释生物学问题,将多组学数据互补整合起来可以增深我们对生物的全面认识。为此,科学家已经开发了多种方法,例如多核学习、贝叶斯共识聚类、基于机器学习(ML)的降维、相似性网络融合和深度学习(DL)方法等。
近日,中国军事医学研究院伯晓晨/何松团队联合厦门大学张仲楠团队在Genome Biology期刊上发表了题为“A benchmark study of deep learning-based multi-omics data fusion methods for cancer”的研究文章。研究团队选取了16种具有代表性的深度学习方法,在模拟数据、单细胞数据和癌症多组学数据集上进行了综合评估。比较结果表明,moGAT具有最佳的数据分类能力;efmmdVAE、efVAE以及 lfmmdVAE具有最佳的数据聚类能力。
文章发表在Genome Biology上
研究人员采用了三种类型的数据:1.通过模拟获得的数据;2.单细胞多组学数据;3.癌症多组学数据。利用16种基于深度学习的数据融合算法对上述三种多组学数据进行评估,主要在分类、聚类和相关性分析三种工作场景下进行。(图1)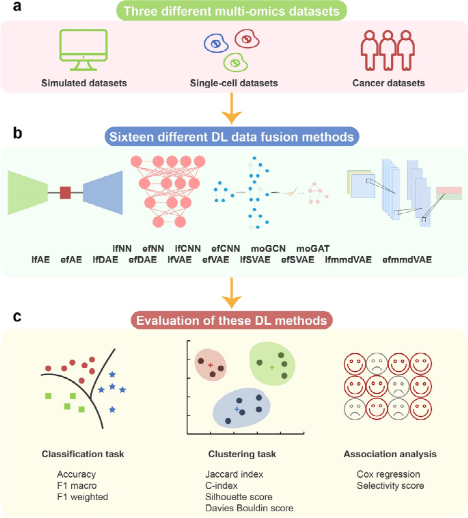
研究人员使用InterSIM CRAN包生成了模拟多组学数据,并使用基于深度学习的多组学算法进行计算与评估。这些模拟数据包括DNA甲基化、mRNA基因表达和蛋白质表达等数据。通过区分有监督方法和无监督方法,并通过一定的指标进行评价,最终获得分析的结果。
将多组学数据融合算法应用于单细胞多组学数据有助于系统地探索细胞的异质性。单细胞数据集由两种组学数据类型组成,即单细胞染色质可及性数据和单细胞基因表达数据。研究团队利用来自三种不同癌细胞系(HTC、Hela 和 K562)206个细胞的两种组学数据进行了算法评估 。与上述模拟多组学数据评估类似,研究人员将方法分为有监督分类方法和无监督分类法,进行评价和分析。近年来,高通量测序技术的快速发展使研究人员能够获得各种癌症类型的多组学图谱。为了更好地了解癌症的分子和临床特征,使用的多组学数据融合算法至关重要。研究人员从癌症基因组图谱(TCGA)数据库中获取了数据集,包括基因表达、DNA甲基化和miRNA表达等数据。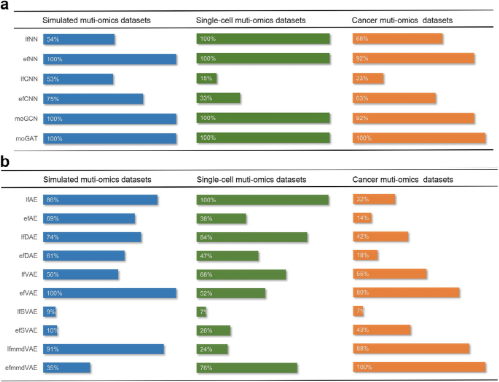
图2. 基于深度学习计算方法的测试与评价
在模拟多组学数据集上进行评估时,大多数监督方法在分类任务中表现出良好的性能,尤其是efNN、moGCN和moGAT。两种基于卷积神经网络(CNN)的方法(efCNN和lfCNN)效果较差,表明在输入上使用带有一维卷积层的CNN可能不适合多组学数据融合。对于聚类分析,efAE、lfmmdVAE和efVAE表现最好。与模拟数据集的结果类似,moGCN和moGAT在单细胞数据集的分类任务上表现非常出色。对于单细胞数据集上聚类性能的评价,efmmdVAE和lfAE是最有效的方法。在癌症数据集中,moGAT在分类任务上仍然优于其他监督方法。在评估聚类性能时,efmmdVAE、efVAE和lfmmdVAE在大多数场景下取得了较优的结果。在嵌入生存或临床注释等信息时,评估组学数据与之的关联,lfVAE和lfSVAE是最有效的。因此,对于需要嵌入额外信息的研究,lfVAE和lfSVAE值得优先考虑。基于上述结果,为了使评价更加直观,研究人员定义了一个统一的分数,并根据统一的分数对这些深度学习算法进行排序。如图2所示,对于分类任务moGAT在三个不同的多组学数据集上排名第一。对于聚类任务,efVAE、lfmmdVAE和lfAE是模拟数据集上排名前三的算法。lfAE、lfDAE和efmmdVAE是针对单细胞数据集的前三种算法。efmmdVAE、lfmmdVAE和efVAE是针对癌症数据集的前三种算法。越来越多的证据表明,多组学数据融合分析在广泛的生物医学研究中发挥着重要作用。该研究系统地评估了16种基于深度学习的算法,结果表明,moGAT具有最佳的数据分类能力;efmmdVAE、 efVAE以及lfmmdVAE具有最佳的数据聚类能力。总体而言,专注于分类任务的研究人员应优先考虑基于GNN的算法,基于GNN的算法可将多组学数据构建成相似网络,样本之间的相关性可以通过相似性网络捕获。因此,研究人员可以有效地利用数据的组学特征和几何结构,提高分类性能。在关注聚类任务时,可优先考虑efmmdVAE、efVAE和lfmmdVAE,这三种算法在所有不同的基准测试中具有最有效和最一致的表现。Leng, D., Zheng, L., Wen, Y. et al. A benchmark study of deep learning-based multi-omics data fusion methods for cancer. Genome Biol 23, 171 (2022).https://genomebiology.biomedcentral.com/articles/10.1186/s13059-022-02739-2