该平台由位于弗吉尼亚大学公共卫生基因组学中心的计算生物学和生物信息学研究小组(Sheffield lab of computational biology)建立。上次修改/更新是2021年11月。
http://refgenie.databio.org/Here we provide a web interface and a RESTful API to access genome assets for popular reference genome assemblies.
refgenie提供了人、鼠等常见物种的参考基因组以及注释文件等信息(fasta、gtf...)以及常用fastq比对工具的(star、hisat2...)索引文件。(如下图所示)
 部分物种
部分物种refgenie不仅提供了网站点击下载方式,也可以通过linux命令行的方式,下载并且管理我们的参考数据,在下载以及使用中大大提高了效率。以下简单总结下,基于命令行的下载、管理refgenie参考数据的用法。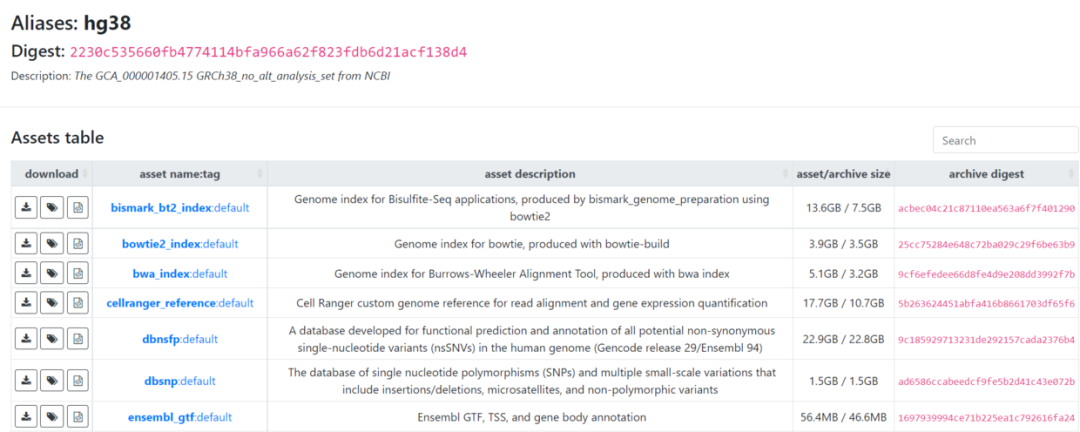
1、安装
pip install --user refgenie
#or
conda install refgenie
2、准备
mkdir ~/refgenie
refgenie init -c ~/refgenie/genome_config.yaml
#之后下载的数据基本都要使用 -c参数 用于指定genome_config.yaml文件路径
#列出所有的genome/asset
refgenie listr -c ~/refgenie/genome_config.yaml
#列出指定物种类型的所有数据类型(如下代码块所示)
refgenie listr -g hg38 -c ~/refgenie/genome_config.yaml
Remote refgenie assets
Server URL: http://refgenomes.databio.org
┏━━━━━━━━┳━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━┳━━━━━━━━━┓
┃ genome ┃ asset (seek_keys) ┃ tags ┃
┡━━━━━━━━╇━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━╇━━━━━━━━━┩
│ hg38 │ fasta (fasta, fai, chrom_sizes, dir) │ default │
│ hg38 │ gencode_gtf (gencode_gtf, dir) │ default │
│ hg38 │ refgene_anno (refgene_anno, refgene_tss, refgene_exon, refgene_intron, │ default │
│ │ refgene_pre_mRNA, dir) │ │
│ hg38 │ dbsnp (dbsnp, tabix, dir) │ default │
│ hg38 │ ensembl_gtf (ensembl_gtf, ensembl_tss, ensembl_gene_body, dir) │ default │
│ hg38 │ ensembl_rb (ensembl_rb, dir) │ default │
│ hg38 │ suffixerator_index (esa, dir) │ default │
│ hg38 │ bwa_index (bwa_index, dir) │ default │
│ hg38 │ bowtie2_index (bowtie2_index, dir) │ default │
│ hg38 │ dbnsfp (dbnsfp, tabix, dir) │ default │
│ hg38 │ star_index (star_index, dir) │ default │
│ hg38 │ fasta_txome (fasta_txome, fai, chrom_sizes, dir) │ default │
│ hg38 │ hisat2_index (hisat2_index, dir) │ default │
│ hg38 │ cellranger_reference (cellranger_reference, dir) │ default │
│ hg38 │ bismark_bt2_index (bismark_bt2_index, dir) │ default │
│ hg38 │ salmon_partial_sa_index (salmon_partial_sa_index, dir) │ default │
│ hg38 │ tgMap (tgMap, dir) │ default │
│ hg38 │ salmon_sa_index (salmon_sa_index, dir) │ default │
└────────┴─────────────────────────────────────────────────────────────────────────┴─────────
3、pull下载指定的genome/asset
 img
img#下载hg38版本的fasta文件
refgenie pull hg38/fasta -c ~/refgenie/genome_config.yaml
#下载hg38版本的gtf文件
refgenie pull hg38/gencode_gtf -c ~/refgenie/genome_config.yaml
#下载hg38版本的salmon软件的索引文件
refgenie pull hg38_cdna/salmon_index -c ~/refgenie/genome_config.yaml
#列出本地已经下载的参考数据
refgenie list -c ~/refgenie/genome_config.yaml
refgenie list -g hg38 -c ~/refgenie/genome_config.yaml
#返回已经下载的genome/asset的路径,这在批量化的脚本文件中十分有用
echo $(refgenie seek hg38/fasta -c ~/refgenie/genome_config.yaml)
# /home/ssli/refgenie/alias/hg38/fasta/default/hg38.fa
head $(refgenie seek hg38/fasta -c ~/refgenie/genome_config.yaml)
# >chr1 AC:CM000663.2 gi:568336023 LN:248956422 rl:Chromosome M5:6aef897c3d6ff0c78aff06ac189178dd AS:GRCh38
# NNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN
# NNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN
# NNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN
# NNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN
# NNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN
# NNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN
# NNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN
# NNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN
# NNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN
refgenie还可以建立比对索引文件等功能,详见帮助文档:http://refgenie.databio.org/en/latest/